
Wetenschap
Wiskunde stelt wetenschappers in staat de organisatie binnen een celkern te begrijpen
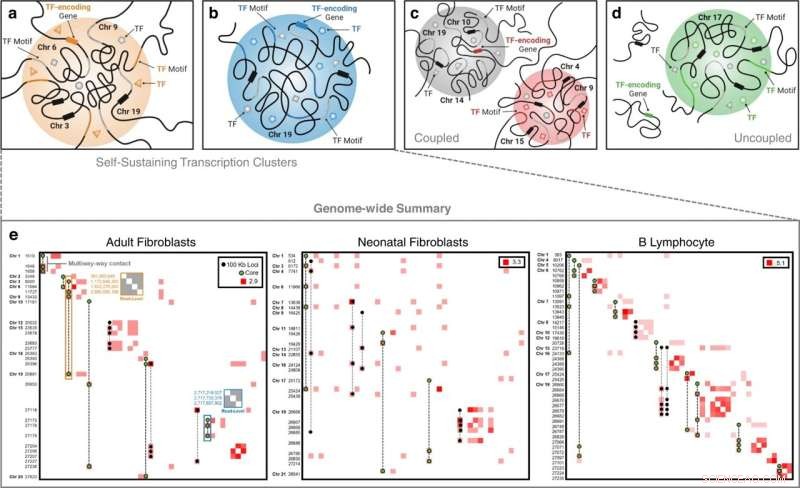
Klassen van transcriptieclusters. In een zichzelf in stand houdende transcriptiecluster zijn een TF en het gen dat voor die TF codeert beide aanwezig. De inter- en intra-chromosomale voorbeelden in (a) en (b), respectievelijk, illustreren dit fenomeen waar in a we de TF van belang (oranje driehoek) zien circuleren in het cluster, het bindende motief aanwezig op het chromatine (oranje gedeelte ), en het bijbehorende gen tot expressie gebracht (oranje rechthoek op chromosoom 6). De grijze vormen vertegenwoordigen extra TF's met bindingsmotieven (grijs deel van chromatine) op het cluster. Zwarte rechthoeken op Chromosomen 3, 9 en 19 vertegenwoordigen extra genen die in het cluster aanwezig zijn. c Een analoog-onafhankelijke klasse van transcriptieclusters waarbij we een TF (rood vierkant) zien binden aan een transcriptiecluster (rood cluster) en het overeenkomstige gen wordt uitgedrukt in een afzonderlijk transcriptiecluster (grijze cluster), maar niet in hetzelfde cluster. d Een analoog-onafhankelijke klasse van transcriptieclusters waarbij we een TF (groene cirkel) zien binden aan een transcriptiecluster (groen cluster) en het overeenkomstige gen wordt tot expressie gebracht maar niet binnen een transcriptiecluster. e Genoombrede celtype-specifieke zelfonderhoudende transcriptieclusters geëxtraheerd uit meerwegcontactgegevens en ontleed in Hi-C-contactmatrices met een resolutie van 100 kb. Contactfrequenties worden log-getransformeerd voor een betere visualisatie. Frequenties langs de diagonaal geven interactie aan tussen twee of meer unieke meerwegloci die binnen dezelfde 100 kb bin vallen. Aslabels zijn niet-aangrenzende bin-coördinaten van 100 kb in chromosomale volgorde. Multi-way contacten die deel uitmaken van de zichzelf onderhoudende transcriptie clusters worden over elkaar heen gelegd. Meerwegcontacten met groengekleurde loci vertegenwoordigen 'kern'-transcriptieclusters - transcriptieclusters die een hoofdregulator en zijn genanaloog bevatten. Een voorbeeld van een contactkaart op leesniveau voor de interchromosomale FOXO3-zelfvoorzienende transcriptiecluster wordt aangegeven door het oranje gemarkeerde vak in de volwassen fibroblast-contactmatrix en een contactkaart op leesniveau voor de intra-chromosomale ZNF320 zelfvoorzienende transcriptiecluster is aangegeven door het blauw gemarkeerde vak. Waarden langs de linkeras van deze contactmatrices op leesniveau zijn basenpaarposities van de contactplaatsen in het genoom. Krediet:Natuurcommunicatie (2022). DOI:10.1038/s41467-022-32980-z
De derde wet van sciencefictionschrijver Arthur C. Clarke zegt dat 'elke voldoende geavanceerde technologie niet te onderscheiden is van magie'.
Indika Rajapakse, Ph.D., is een gelovige. De ingenieur en wiskundige is nu een bioloog. En hij gelooft dat de schoonheid van het combineren van deze drie disciplines cruciaal is om te ontrafelen hoe cellen werken.
Zijn laatste ontwikkeling is een nieuwe wiskundige techniek om te begrijpen hoe de kern van een cel is georganiseerd. De techniek, die Rajapakse en medewerkers op verschillende soorten cellen testten, onthulde wat de onderzoekers zelfvoorzienende transcriptieclusters noemden, een subset van eiwitten die een sleutelrol spelen bij het behouden van de celidentiteit.
Ze hopen dat dit begrip kwetsbaarheden aan het licht zal brengen die als doelwit kunnen worden gebruikt om een cel te herprogrammeren om kanker of andere ziekten te stoppen.
"Steeds meer kankerbiologen denken dat de organisatie van het genoom een grote rol speelt bij het begrijpen van oncontroleerbare celdeling en of we een kankercel kunnen herprogrammeren. Dat betekent dat we meer details moeten begrijpen over wat er in de kern gebeurt", zegt Rajapakse, universitair hoofddocent computational geneeskunde en bio-informatica, wiskunde en biomedische technologie aan de Universiteit van Michigan. Hij is ook lid van het U-M Rogel Cancer Center.
Rajapakse is senior auteur van het artikel, gepubliceerd in Nature Communications . Het project werd geleid door een drietal afgestudeerde studenten met een interdisciplinair team van onderzoekers.
Het team verbeterde een oudere technologie om chromatine te onderzoeken, Hi-C genaamd, dat in kaart brengt welke delen van het genoom zich dicht bij elkaar bevinden. Het kan chromosoomtranslocaties identificeren, zoals die bij sommige kankers voorkomen. De beperking is echter dat het alleen deze aangrenzende genomische regio's ziet.
De nieuwe technologie, Pore-C genaamd, gebruikt veel meer gegevens om te visualiseren hoe alle stukjes in de celkern op elkaar inwerken. De onderzoekers gebruikten een wiskundige techniek genaamd hypergraphs. Denk aan:driedimensionaal Venn-diagram. Het stelt onderzoekers in staat om niet alleen paren van genomische regio's te zien die op elkaar inwerken, maar de totaliteit van de complexe en overlappende genoombrede relaties binnen de cellen.
"Deze multidimensionale relatie kunnen we ondubbelzinnig begrijpen. Het geeft ons een meer gedetailleerde manier om organisatorische principes in de kern te begrijpen. Als je dat begrijpt, kun je ook begrijpen waar deze organisatorische principes afwijken, zoals bij kanker," zei Rajapakse. "Dit is alsof je drie werelden samenvoegt - technologie, wiskunde en biologie - om meer details in de kern te bestuderen."
De onderzoekers testten hun aanpak op neonatale fibroblasten, biopsie van volwassen fibroblasten en B-lymfocyten. Ze identificeerden organisaties van transcriptieclusters die specifiek zijn voor elk celtype. Ze vonden ook wat ze zelfonderhoudende transcriptieclusters noemden, die dienen als belangrijke transcriptionele handtekeningen voor een celtype.
Rajapakse beschrijft dit als de eerste stap in een groter geheel.
"Mijn doel is om dit soort beeld over de celcyclus te construeren om te begrijpen hoe een cel verschillende stadia doorloopt. Kanker is een oncontroleerbare celdeling," zei Rajapakse. "Als we begrijpen hoe een normale cel in de loop van de tijd verandert, kunnen we gecontroleerde en ongecontroleerde systemen gaan onderzoeken en manieren vinden om dat systeem te herprogrammeren." + Verder verkennen
Onthulling van de mysteries van de genoomstructuur in de menselijke celkern met behulp van een 3D-computersimulatie
 Nieuw aminozuur kan leiden tot vooruitgang bij ernstige ziekten
Nieuw aminozuur kan leiden tot vooruitgang bij ernstige ziekten Innovatieve technieken voor DNA-herstel kunnen slachtoffers helpen verkrachters in Kenia te pakken
Innovatieve technieken voor DNA-herstel kunnen slachtoffers helpen verkrachters in Kenia te pakken Densiteitsexperimenten voor kinderen
Densiteitsexperimenten voor kinderen Uitbreiding van het temperatuurbereik van lithium-ionbatterijen
Uitbreiding van het temperatuurbereik van lithium-ionbatterijen Wetenschappers vinden lichtgewicht vloeibare metalen materialen uit
Wetenschappers vinden lichtgewicht vloeibare metalen materialen uit
 Nieuwe Londense vervuilingsbelasting wordt van kracht
Nieuwe Londense vervuilingsbelasting wordt van kracht Koolstofopname door Amazonebossen komt overeen met uitstoot van regio's
Koolstofopname door Amazonebossen komt overeen met uitstoot van regio's CryoSat onthult terugtrekking van Patagonische gletsjers
CryoSat onthult terugtrekking van Patagonische gletsjers Magnitude 6 aardbeving treft Samoa, Amerikaans Samoa
Magnitude 6 aardbeving treft Samoa, Amerikaans Samoa Sterke aardbevingen schudden een deel van West-Papoea in Indonesië
Sterke aardbevingen schudden een deel van West-Papoea in Indonesië
Hoofdlijnen
- Wetenschappers vinden antarctische microben die alleen van lucht leven
- Bacteriën levenscyclus
- Combinatie van warmer water, blootstelling aan chemicaliën versterkt schadelijke effecten bij een kustvis
- Welke soorten cellen delen door mitose en cytokinese?
- Veranderingen in diervoeder kunnen 1 miljard mensen van voedsel voorzien
- De hectische 24-uurs inspanning om een bedreigde, verweesde vleermuis
- Wat is het verschil tussen een bacteriële en virale infectie?
- Waarom het een goed idee is om met je dronken oom over politiek te praten
- Ideeën voor het maken van een 3-D DNA-standaard voor middelbare school
- Welk percentage botten in het lichaam bestaat uit het axiale skelet?
- Virusstamping - een veelzijdige nieuwe methode voor genetische manipulatie van afzonderlijke cellen

- Invasieve wormen verspreiden zich in arboretumbossen, beperkte effecten tot nu toe

- Slechts een kleine hoeveelheid olie beschadigt de veren van zeevogels, blijkt uit onderzoek

- Walvisstranden:vijf vragen beantwoord
 LA Unified cyberaanvallers eisen losgeld
LA Unified cyberaanvallers eisen losgeld Bestrijding van klimaatverandering: Waarom beleggers hun aandelen in fossiele brandstofbedrijven moeten houden
Bestrijding van klimaatverandering: Waarom beleggers hun aandelen in fossiele brandstofbedrijven moeten houden De verschillen tussen 1D, 2D en 3D Afbeeldingen
De verschillen tussen 1D, 2D en 3D Afbeeldingen Mechanisme achter door magnetisme aangedreven negatieve thermische uitzetting (NTE) voor het eerst uitgelegd
Mechanisme achter door magnetisme aangedreven negatieve thermische uitzetting (NTE) voor het eerst uitgelegd Rook van bosbranden in Australië gelinkt aan honderden doden
Rook van bosbranden in Australië gelinkt aan honderden doden Hier ziet u hoe het einde van de zomertijd u kan beïnvloeden
Hier ziet u hoe het einde van de zomertijd u kan beïnvloeden  Wat is een stormvloedkering en waarom is het zo gevaarlijk?
Wat is een stormvloedkering en waarom is het zo gevaarlijk? Het verschil tussen een grasland en savanne
Het verschil tussen een grasland en savanne
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

