
Wetenschap
Raman-microspectroscopie gebruiken om snel ziekteverwekkende bacteriën te detecteren
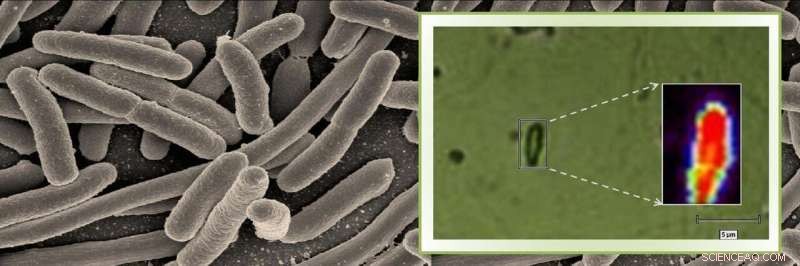
Krediet:Srividya Kumar
Onderzoekers van het Indian Institute of Science (IISc) hebben een methode ontwikkeld om snel te identificeren en te controleren of een ziekteverwekkende bacterie levend of dood is.
Het maakt gebruik van Raman-spectroscopie, een techniek die meestal wordt gebruikt om chemische bindingen in materialen te identificeren, om bacteriën te herkennen en te controleren op hun levensvatbaarheid, of hun staat van leven.
"Het unieke van deze studie is de snelheid en gevoeligheid van de methode, en het potentieel om te worden gewijzigd in een bed-side, tafelmodel voor diagnose, " zegt Srividya Kumar, een voormalig Ph.D. student aan de afdeling Anorganische en Fysische Chemie, en eerste auteur van de studie gepubliceerd in het tijdschrift Analytische en bioanalytische chemie .
Snelle detectie van de ziekteverwekker of ziekteverwekker, evenals het controleren of het in patiëntenmonsters leeft of niet, zijn de sleutel tot de behandeling van infectieziekten. Het identificeren van de levensvatbaarheid van de ziekteverwekker helpt artsen ook bij het bepalen van de voor te schrijven dosis antibiotica, en vermindert de mogelijkheid van te veel voorschrijven dat kan leiden tot antibioticaresistentie.
Besmettelijke bacteriën worden meestal geïdentificeerd door technieken zoals kweken - ze laten groeien op een voedingsbodem in een petrischaal. Het kan ongeveer twee tot drie dagen duren om hun groei te volgen en te bevestigen of ze dood of levend zijn. In aanvulling, bacteriën die moeilijk te kweken zijn in laboratoriumculturen blijven vaak onopgemerkt met conventionele methoden. Meer geavanceerde methoden zoals Polymerase Chain Reaction (PCR) creëren een genetisch profiel van de microbe, maar kan niet zeggen of het levend of dood is.
Raman-spectroscopie wordt veel gebruikt in de chemie om de structuur van moleculen te onderzoeken. In dit onderzoek, echter, Kumar en haar collega's hebben deze aanpak gecombineerd met geavanceerde microscopie om te controleren op de aanwezigheid van bacteriën. Ze gebruikten deze techniek om te detecteren of de tuberculosebacterie (Mycobacterium tuberculosis) aanwezig was in een monstermedium dat lijkt op sputum. Ze testten ook monsters met vijf andere microben.
Met behulp van de microspectroscoop, de onderzoekers kaatsten een laserstraal van het monster en vingen het door het monster verstrooide licht op in de vorm van een spectrum, die varieert met de biochemische samenstelling van de bacterie. Elke bacteriesoort genereert een uniek spectrum met specifieke waarden voor intensiteit en golflengtepositie van het verstrooide licht, afhankelijk van het type chemische bindingen dat aanwezig is in de bacteriële cel. De techniek was gevoelig genoeg om zelfs voor een enkele bacteriële cel een spectrum te genereren.
Het materiaal dat wordt gebruikt voor het monteren van de bacteriën in deze techniek, bekend als het substraat, is ook cruciaal omdat sommige substraten ruis kunnen toevoegen aan het biochemische profiel. De onderzoekers ontwikkelden een neutrale, op aluminium gebaseerd dunnefilmsubstraat dat geen achtergrondsignalen genereert die de spectra kunnen verstoren.
Met behulp van deze methode, de onderzoekers waren niet alleen in staat om de bacterie te identificeren, maar waren ook in staat om onderscheid te maken tussen levende en dode bacteriële cellen - aangezien de chemische samenstelling van de twee verschilt - binnen twee tot drie uur na monstername.
Aangezien elke bacteriesoort aanleiding geeft tot een uniek spectrum, het zou mogelijk kunnen zijn om uiteindelijk een database van pathogenen op te bouwen die kan worden gebruikt bij klinische diagnose.
"Hoewel we de methode voor tuberculose hebben gevalideerd, de methodologie kan worden uitgebreid tot elk type bacteriële infectie, " zegt Deepak Saini, universitair hoofddocent bij de afdeling Moleculaire Reproductie, Ontwikkeling en genetica, IISc, en een van de senior auteurs van de studie.
 Hout vormt de sleutel tot het verminderen van de voedselonzekerheid in Afrika bezuiden de Sahara
Hout vormt de sleutel tot het verminderen van de voedselonzekerheid in Afrika bezuiden de Sahara 6 stappen over hoe wolken worden gevormd
6 stappen over hoe wolken worden gevormd  Chinese boer die eco-strijder is geworden, gaat grote zaken aan
Chinese boer die eco-strijder is geworden, gaat grote zaken aan Bomen die snel leven sterven jong
Bomen die snel leven sterven jong Arctische rivieren bieden vingerafdrukken van koolstofafgifte door ontdooiende permafrost
Arctische rivieren bieden vingerafdrukken van koolstofafgifte door ontdooiende permafrost
Hoofdlijnen
- Thomas Malthus: Biografie, Populatietheorie & Feiten
- Hoe vogelgriep werkt
- EU-lidstaten stemmen volgende maand over vijfjarige verlenging van onkruidverdelger
- Sonic Kayaks:Milieumonitoring en experimentele muziek door burgers
- Zijn mensen zonder verstandskiezen hoger ontwikkeld?
- Wat zijn de verschillen tussen een CZS en een PNS?
- Woedend debat:houdt het ruimen van wolven stroperij tegen?
- Gestratificeerd epitheelweefsel: definitie, structuur, typen
- Verschillen en overeenkomsten tussen Unicellular & Cellular
- Nog een geheim van spinnenzijde ontrafelen - het is een kabel

- Nieuwe brandstofceltechnologie draait op vaste koolstof

- Zonnereiniger maakt zijn eigen ontsmettingsmiddel uit water en zonlicht

- Hogedrukwetenschappers ontdekken veelbelovend materiaal voor informatietechnologie

- Energie besparen bij de productie van chemicaliën

 Kunnen buitenaardse wezens sterren gebruiken om het uitdijende universum voor te blijven?
Kunnen buitenaardse wezens sterren gebruiken om het uitdijende universum voor te blijven? Vingertracering verbetert het leren:bewijs voor 100 jaar oude praktijk gebruikt door Montessori
Vingertracering verbetert het leren:bewijs voor 100 jaar oude praktijk gebruikt door Montessori Quantum dot solids:deze generatie siliciumwafel?
Quantum dot solids:deze generatie siliciumwafel? 'S Werelds allereerste grafeen wandelschoenen onthuld
'S Werelds allereerste grafeen wandelschoenen onthuld Akoestisch microfluïdisch platform scheidt circulerende tumorcellen voorzichtig en snel van bloedmonsters
Akoestisch microfluïdisch platform scheidt circulerende tumorcellen voorzichtig en snel van bloedmonsters Veerkrachtige en duurzame rubberen trottoirs kunnen duizenden levens redden, wetenschappers zeggen:
Veerkrachtige en duurzame rubberen trottoirs kunnen duizenden levens redden, wetenschappers zeggen: Hoe het gebied van een Pentagon te vinden
Hoe het gebied van een Pentagon te vinden  Moeten onderwijs en communicatie over de vooruitgang van microfluïdica worden verbeterd?
Moeten onderwijs en communicatie over de vooruitgang van microfluïdica worden verbeterd?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

