
Wetenschap
RNA-bewerkingstool een snelle, gevoelige test voor COVID-19
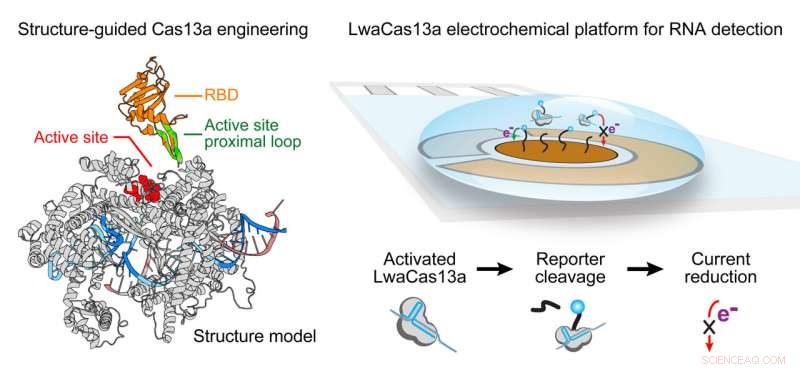
Met behulp van structuurgeleide Cas13 hebben onderzoekers van Rice University en de University of Connecticut een tool voor het bewerken van genen aangepast om te dienen als een zeer gevoelige diagnostische test voor de aanwezigheid van het SARS-CoV-2-virus. Ze gebruikten een kant-en-klare elektrochemische sensor om resultaten te leveren. Krediet:Jie Yang/Rice University
Een gemanipuleerde, op CRISPR gebaseerde methode die RNA vindt van SARS-CoV-2, het virus dat COVID-19 veroorzaakt, belooft het testen op die en andere ziekten snel en gemakkelijk te maken.
Medewerkers van Rice University en de University of Connecticut hebben het RNA-editing CRISPR-Cas13-systeem verder ontwikkeld om hun vermogen te vergroten voor het detecteren van minieme hoeveelheden van het SARS-CoV-2-virus in biologische monsters zonder de tijdrovende RNA-extractie en amplificatiestap die nodig is in gouden standaard PCR-testen.
Het nieuwe platform was zeer succesvol in vergelijking met PCR en vond 10 van de 11 positieven en geen valse positieven voor het virus in tests op klinische monsters rechtstreeks van neusuitstrijkjes. De onderzoekers toonden aan dat hun techniek tekenen van SARS-CoV-2 vindt in attomolair (10 -18 ) concentraties.
De studie onder leiding van chemisch en biomoleculair ingenieur Xue Sherry Gao van Rice's George R. Brown School of Engineering en postdoctoraal onderzoekers Jie Yang van Rice en Yang Song van Connecticut verschijnt in Nature Chemical Biology .
Cas13 maakt, net als zijn bekendere neef Cas9, deel uit van het systeem waarmee bacteriën zich van nature verdedigen tegen binnendringende fagen. Sinds de ontdekking is CRISPR-Cas9 door wetenschappers aangepast om levende DNA-genomen te bewerken en is het veelbelovend om ziekten te behandelen en zelfs te genezen.

Postdoctoraal onderzoeker Jie Yang van Rice University leidde een poging om Cas13-tools voor genoombewerking aan te passen om te dienen als een zeer gevoelige detector voor de aanwezigheid van het SARS-CoV-2-virus, dat COVID-19 veroorzaakt. Krediet:Jeff Fitlow/Rice University
En het kan ook op andere manieren worden gebruikt. Cas13 op zichzelf kan worden verbeterd met gids-RNA om doel-RNA-sequenties te vinden en te knippen, maar ook om 'onderpand' te vinden, in dit geval de aanwezigheid van virussen zoals SARS-CoV-2.
"Het gemanipuleerde Cas13-eiwit in dit werk kan gemakkelijk worden aangepast aan andere eerder gevestigde platforms," zei Gao. "De stabiliteit en robuustheid van de Cas13-varianten maken ze meer geschikt voor point-of-care-diagnostiek in gebieden met weinig middelen wanneer dure PCR-machines niet beschikbaar zijn."
Yang zei dat wildtype Cas13, afkomstig van een bacterie, Leptotrichia wadei, het attomolaire niveau van viraal RNA niet binnen een tijdsbestek van 30 tot 60 minuten kan detecteren, maar de verbeterde versie die bij Rice is gemaakt, doet het werk in ongeveer een half uur en detecteert SARS -CoV-2 in veel lagere concentraties dan de vorige tests.
Ze zei dat de sleutel een goed verborgen, flexibele haarspeldlus is in de buurt van de actieve site van Cas13. "Het zit in het midden van het eiwit in de buurt van de katalytische plaats die de activiteit van Cas13 bepaalt," zei Yang. "Omdat Cas13 groot en dynamisch is, was het een uitdaging om een site te vinden om een ander functioneel domein in te voegen."
De onderzoekers fuseerden zeven verschillende RNA-bindende domeinen aan de lus, en twee van de complexen waren duidelijk superieur. Wanneer ze hun doelwitten vonden, zouden de eiwitten fluoresceren, wat de aanwezigheid van het virus aan het licht bracht.
"We konden zien dat de verhoogde activiteit vijf- of zesvoudig was ten opzichte van het wildtype Cas13," zei Yang. "Dit aantal lijkt klein, maar het is behoorlijk verbazingwekkend met een enkele stap van eiwittechnologie.
"But that was still not enough for detection, so we moved the whole assay from a fluorescence plate reader, which is quite large and not available in low-resource settings, to an electrochemical sensor, which has higher sensitivity and can be used for point-of-care diagnostics," she said.
With the off-the-shelf sensor, Yang said the engineered protein was five orders of magnitude more sensitive in detecting the virus compared to the wild-type protein.
The lab wants to adapt its technology to paper strips like those in home COVID-19 antibody tests, but with much higher sensitivity and accuracy. "We hope that will make testing more convenient and with lower cost for many targets," Gao said.
The researchers are also investigating improved detection of the Zika, dengue and Ebola viruses and predictive biomarkers for cardiovascular disease. Their work could lead to rapid diagnosis of the severity of COVID-19.
"Different viruses have different sequences," Yang said. "We can design guide RNA to target a specific sequence that we can then detect, which is the power of the CRISPR-Cas13 system."
But because the project began just as the pandemic took hold, SARS-CoV-2 was a natural focus. "The technology is quite amenable to all the targets," she said. "This makes it a very good option to detect all kinds of mutations or different coronaviruses."
"We are very excited about this work as a combinational effort of structure biology, protein engineering and biomedical device development," Gao added. "I greatly appreciate all the efforts from my lab members and collaborators."
Co-authors of the paper are Rice postdoctoral researcher Xiangyu Deng, undergraduate Jeffrey Vanegas and graduate student Zheng You; graduate students Yuxuan Zhang and Zhengyan Weng of the University of Connecticut; microbiology supervisor Lori Avery and Kevin Dieckhaus, a professor of medicine, of UConn Health; Yi Zhang, an assistant professor of biomedical engineering at the University of Connecticut; and Yang Gao, an assistant professor of biosciences at Rice. + Verder verkennen
Compact CRISPR system enables portable COVID-19 testing
 Tumoren direct aanvallen bij identificatie
Tumoren direct aanvallen bij identificatie Polymeren die kameleonhuid nabootsen
Polymeren die kameleonhuid nabootsen Wetenschappers ontwikkelen volledig door zonne-energie aangedreven autonome chemische minifabriek
Wetenschappers ontwikkelen volledig door zonne-energie aangedreven autonome chemische minifabriek Hoe beïnvloedt het veranderen van de temperatuur de viscositeit en oppervlaktespanning van een vloeistof?
Hoe beïnvloedt het veranderen van de temperatuur de viscositeit en oppervlaktespanning van een vloeistof?  Berekeningen van scheikundigen kunnen de voorspelling van kanker bevorderen
Berekeningen van scheikundigen kunnen de voorspelling van kanker bevorderen
Hoofdlijnen
- Twee nieuwe ontdekkingen werpen licht op het mysterie van hoe cellen omgaan met stress
- Ontdekking van circadiane klokken kan de waterefficiëntie in voedselplanten helpen verhogen
- Je wandeling is zo duidelijk dat het diepe persoonlijkheidskenmerken kan onthullen
- EP-leden dringen aan op onderzoek naar de invloed van Monsanto op onderzoek
- Cheerleading Science Fair Projectideeën
- Geluidsoverlast storend voor scholende vissen
- Meer dan 75 procent afname van de totale biomassa van vliegende insecten in 27 jaar
- Zuid-Amerikaanse brulapen zijn mogelijk meer bedreigd dan eerder werd gedacht
- Cephalization of Earthworms
 Soorten wolken voor kinderen
Soorten wolken voor kinderen Hoe Force of Impact berekenen
Hoe Force of Impact berekenen  Watermonsters onthullen microplastics in afgelegen Patagonische fjordensysteem
Watermonsters onthullen microplastics in afgelegen Patagonische fjordensysteem Onderzoekers onthullen de nano-eigenschappen van Eucalypt
Onderzoekers onthullen de nano-eigenschappen van Eucalypt Wetenschappers vinden sneller uit, goedkopere strategie voor het ontwerpen van infrarood-emitterende materialen
Wetenschappers vinden sneller uit, goedkopere strategie voor het ontwerpen van infrarood-emitterende materialen Oud menselijk DNA in Afrika bezuiden de Sahara licht sluier over de prehistorie op
Oud menselijk DNA in Afrika bezuiden de Sahara licht sluier over de prehistorie op Mogelijk bewijs dat een extrasolair object in 2014 de aarde raakt
Mogelijk bewijs dat een extrasolair object in 2014 de aarde raakt De toxiciteit van Reddit-reacties beoordelen
De toxiciteit van Reddit-reacties beoordelen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com






