
Wetenschap
Benchmarking computationele methoden voor metanomen
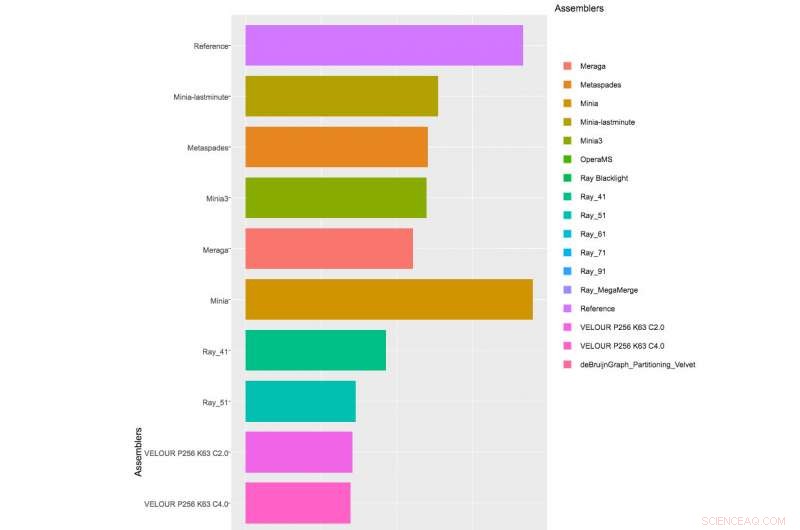
Tabel met gedeeltelijke resultaten van monteurs die zijn toegepast op de 1e CAMI-uitdaging, Dataset 1. Klik hier om de volledige tabel te zien.
Ze zijn overal, maar onzichtbaar voor het blote oog. Microben zijn het onzichtbare, invloedrijke krachten achter de regulering van belangrijke milieuprocessen zoals de koolstofcyclus, toch blijven de meeste onbekend. Al meer dan een decennium, het Amerikaanse Department of Energy Joint Genome Institute (DOE JGI), een DOE Office of Science gebruikersfaciliteit, heeft onderzoekers in staat gesteld om niet-gecultiveerde microben te bestuderen die niet in het laboratorium kunnen groeien, gebruikmakend van state-of-the-art benaderingen zoals high-throughput genomische sequencing van milieugemeenschappen ("metagenomics") en de ontwikkeling van computerhulpmiddelen om microbiële gemeenschappen uit de omgeving te ontdekken en te karakteriseren. Om het assembleren van metanomen in een reeks overlappende DNA-segmenten aan te pakken die samen een consensusgebied van DNA of contigs vertegenwoordigen, dan deze contigs in genoombins te plaatsen, en tot slot het uitvoeren van taxonomische profilering van genoombins, analisten over de hele wereld hebben een reeks verschillende rekentools ontwikkeld, maar tot nu toe was er een gebrek aan consensus over hoe hun prestaties te evalueren.
Gepubliceerd op 2 oktober 2017 in Natuurmethoden , een team met DOE JGI-onderzoekers beschreef de resultaten van de Critical Assessment of Metagenome Interpretation (CAMI) Challenge, de allereerste, door de gemeenschap georganiseerde benchmarkbeoordeling van rekenhulpmiddelen voor metanomen. De CAMI Challenge werd geleid door Alexander Sczyrba, hoofd van de Computational Metagenomics-groep aan de Universiteit van Bielefeld en voorheen een DOE JGI postdoctoraal onderzoeker, en Alice McHardy, hoofd van het Computational Biology of Infection Research Lab van het Helmholtz Center for Infection Research.
"Het is erg moeilijk voor onderzoekers om erachter te komen welk programma ze moeten gebruiken voor een bepaalde dataset en analyse op basis van de resultaten van method papers, " zei McHardy. "De datasets en evaluatiemaatregelen die bij evaluaties worden gebruikt, lopen sterk uiteen. Een ander probleem is dat ontwikkelaars gewoonlijk veel tijd besteden aan het benchmarken van de state-of-the-art bij het beoordelen van de prestaties van nieuwe software op die manier. CAMI wil deze dingen veranderen en betrekt de gemeenschap bij het definiëren van normen en best practices voor evaluatie en om deze principes toe te passen bij benchmarking-uitdagingen."
De CAMI Challenge vond plaats gedurende drie maanden in 2015. Om de rekenhulpmiddelen te beoordelen, de organisatoren ontwikkelden 3 gesimuleerde metagenoomdatasets met behulp van meer dan 300 conceptgenomen van bacteriële en archaeale isolaten, gesequenced en samengesteld door de DOE JGI, die deel uitmaakten van het project Genomic Encyclopedia of Bacteria and Archaeal dat onlangs is gepubliceerd in Natuur Biotechnologie . Deze genomen werden gedeeld met het CAMI Challenges-consortium voordat ze aan het publiek werden vrijgegeven om de objectieve benchmarking van verschillende tools te vergemakkelijken. De datasets bevatten ook ongeveer hetzelfde aantal genomen van het Max Planck Instituut in Keulen, Duitsland, samen met circulaire elementen en virussen. De gesimuleerde datasets waren een enkele voorbeelddataset van 15 miljard basen (Gb), een 40 Gb dataset met 40 genomen en 20 circulaire elementen, en een 75 Gb tijdreeksgegevensset bestaande uit vijf monsters en honderden genomen en cirkelvormige elementen.
"JGI heeft een sterke interesse in benchmarking van tools en technologieën die de analyse van metagenomen zouden bevorderen en de kwaliteit van de gegevens die we aan de gebruikers verstrekken zouden verbeteren. Na de allereerste studie gepubliceerd te hebben over het gebruik van gesimuleerde datasets voor benchmarking van metagenomics-tools uit de JGI, het is geweldig om te zien hoe deze methodologie zich in de loop der jaren heeft uitgebreid en nu door deze studie, evolueren naar een model voor gestandaardiseerde gemeenschapsinspanningen in het veld, " zei Nikos Kyrpides, DOE JGI Prokaryote Super Programma hoofd.
"JGI hecht veel waarde aan niet alleen benchmarking van laboratoriumprotocollen, maar ook computationele workflows, ", voegde Tanja Woyke, hoofd van DOE JGI Microbial Genomics, toe. "Dit maakt onze deelname aan cruciale gemeenschapsinspanningen zoals CAMI zo belangrijk."
Met meer dan 40 teams die zich hebben aangemeld voor de Challenge, en de CAMI-organisatoren ontvingen 215 inzendingen van 25 programma's over de hele wereld, hoewel slechts 17 teams bereid waren hun software-implementaties te publiceren. De CAMI-organisatoren evalueerden rekentools in 3 categorieën. Half a dozen assemblers and assembly pipelines were evaluated on assembling genome sequences generated from short-read sequencing technologies. In the binning challenge, five genome binners and 4 taxonomic binners were evaluated on criteria including the tools' efficacy in recovering individual genomes. Eindelijk, 10 taxonomic profilers with various parameter settings were evaluated on how well they could predict the identities and relative abundances of the microbes and circular elements. The benchmarking results are available on https://data.cami-challenge.org/results.
The CAMI organizers are already planning future benchmarking challenges, for example to evaluate and aid method development for long read sequencing technologies. "CAMI is an ongoing initiative, " noted Sczyrba. "We are currently further automating the benchmarking and comparative result visualizations. And we invite everyone interested to join and work with CAMI on providing comprehensive performance overviews of the computational metagenomics toolkit, to inform developers about current challenges in computational metagenomics and applied scientists of the most suitable software for their research questions."
 Goedkoop, fly footpad-achtige zelfklevende structuur die herhaaldelijk kan worden bevestigd/losgemaakt
Goedkoop, fly footpad-achtige zelfklevende structuur die herhaaldelijk kan worden bevestigd/losgemaakt Nieuw materiaal kan waterstof maken uit zout en vervuild water
Nieuw materiaal kan waterstof maken uit zout en vervuild water De energieniveaus van organische halfgeleiders afstemmen
De energieniveaus van organische halfgeleiders afstemmen Wat is normatieve en descriptieve wetenschap?
Wat is normatieve en descriptieve wetenschap?  Hoe vergroot een microscoop objecten?
Hoe vergroot een microscoop objecten?
 Een oplossing voor het opruimen van PFAS, een van 's werelds meest hardnekkige verontreinigende stoffen
Een oplossing voor het opruimen van PFAS, een van 's werelds meest hardnekkige verontreinigende stoffen Giftige algen nemen toe in Lake Okeechobee in Florida
Giftige algen nemen toe in Lake Okeechobee in Florida Na de goudkoorts:mijnbouw in Kameroen laat open graven achter
Na de goudkoorts:mijnbouw in Kameroen laat open graven achter Het verminderen van de CO2-uitstoot is niet genoeg, expert waarschuwt
Het verminderen van de CO2-uitstoot is niet genoeg, expert waarschuwt De toestand van magma in aardkorstreservoirs
De toestand van magma in aardkorstreservoirs
Hoofdlijnen
- Nieuwe hoop voor ernstig bedreigde aap met stompe neus in Myanmar
- Waarom zijn er veel verschillende soorten tRNA-moleculen?
- Caribische bidsprinkhanen hebben een oude Afrikaanse oorsprong
- Op zoek naar het CRISPR Zwitsers zakmes
- The Anatomy of the Hydra
- Grazende paarden op betere weiden
- We kregen te maken met misbruik omdat we mensen vroegen om wespen te doden voor de wetenschap - hier is waarom het de moeite waard was
- Vissenseks zo luid dat dolfijnen doof kunnen worden
- Nieuwe vogelsoort genoemd naar Harvard-vader van biodiversiteit
- Onderzoekers ontdekken wat er bij libellen op het menu staat

- Kan iemand zich herinneren dat hij geboren is?

- Kan eten mensen gelukkig maken?

- Wat is de volgorde van de opeenvolging van gebeurtenissen bij de bevruchting van een ei?
- Hoe gigantische tropische fruitvleermuizen te redden:werk samen met lokale jagers die vleermuistanden als geld gebruiken

 Grotten geven aan dat de Australische bergen nog steeds groeien
Grotten geven aan dat de Australische bergen nog steeds groeien Nieuwe methode om monomodale, mesoporeuze metaaloxiden
Nieuwe methode om monomodale, mesoporeuze metaaloxiden Wat is de Verzengende Zone?
Wat is de Verzengende Zone?
De verzengende zone verwijst naar het gebied van de aarde nabij de evenaar. Zoals de naam al doet vermoeden, is de verzengende zone over het algemeen warm. Het heeft een nat en droog seizoen, maar ervaart niet de vier se
 Ruimtevaart kan invloed hebben op hoe het lichaam omgaat met natrium
Ruimtevaart kan invloed hebben op hoe het lichaam omgaat met natrium Fenomeen in de Indische Oceaan zorgt voor klimaatproblemen voor Australië
Fenomeen in de Indische Oceaan zorgt voor klimaatproblemen voor Australië NASA's Distant Space Discovery (Ultima Thule) ziet eruit als een sneeuwpop
NASA's Distant Space Discovery (Ultima Thule) ziet eruit als een sneeuwpop  Nieuwe planeet ontdekt in baan om jonge Melkwegster
Nieuwe planeet ontdekt in baan om jonge Melkwegster Heeft de invloed van koude op de invloed van objecten op metaal?
Heeft de invloed van koude op de invloed van objecten op metaal?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | German | Dutch | Danish | Norway | Swedish |

-
Wetenschap © https://nl.scienceaq.com

