
Wetenschap
Statistische test relateert pathogeenmutatie aan progressie van infectieziekten
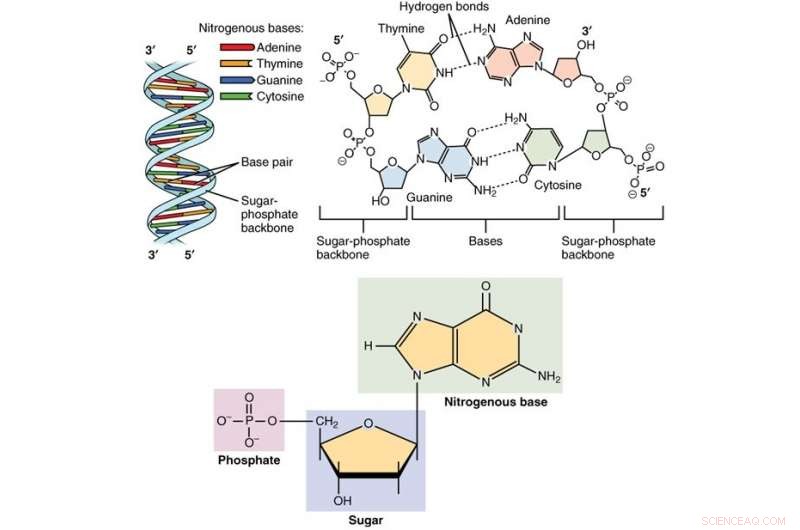
Ryosuke Omori en Jianhong Wu ontwikkelen een inductief algoritme om plaatsspecifieke nucleotidefrequenties te bestuderen met behulp van een multi-strain sensitive-infective-removed (SIR) model om de epistemologie van infectieziekten beter te begrijpen, evolutie van pathogenen, en bevolkingsdynamiek. Krediet:Wikimedia Commons.
Nucleïnezuur sequencing methoden, die de volgorde van nucleotiden in DNA-fragmenten bepalen, gaan snel vooruit. Deze processen leveren grote hoeveelheden sequentiegegevens op - waarvan sommige dynamisch zijn - die onderzoekers helpen begrijpen hoe en waarom organismen functioneren zoals ze doen. Sequencing komt ook ten goede aan epidemiologische studies, zoals de identificatie, diagnose, en behandeling van genetische en/of besmettelijke ziekten. Geavanceerde sequentietechnologieën onthullen waardevolle informatie over de tijdsevolutie van pathogene sequenties. Omdat onderzoekers kunnen inschatten hoe een mutatie zich gedraagt onder druk van natuurlijke selectie, ze zijn dus in staat om de impact van elke mutatie - in termen van overleving en voortplanting - op de geschiktheid van de ziekteverwekker in kwestie te voorspellen. Deze voorspellingen geven inzicht in de epistemologie van infectieziekten, evolutie van pathogenen, en bevolkingsdynamiek.
In een artikel dat eerder deze maand in de SIAM Journal on Applied Mathematics , Ryosuke Omori en Jianhong Wu ontwikkelen een inductief algoritme om plaatsspecifieke nucleotidefrequenties te bestuderen met behulp van een multi-stam gevoelig-infectief-verwijderd (SIR) model. Een SIR-model is een eenvoudig compartimentenmodel dat elk individu in een populatie op een bepaald moment in een van de drie bovengenoemde categorieën plaatst om het theoretische aantal mensen te berekenen dat door een infectieziekte is getroffen. De auteurs gebruiken hun algoritme om Tajima's D te berekenen, een populaire statistische test die natuurlijke selectie op een specifieke locatie meet door verschillen in een steekproef van sequenties uit een populatie te analyseren. In een niet-endemische situatie, Tajima's D kan in de loop van de tijd veranderen. Door de tijdsevolutie van Tajima's D tijdens een uitbraak te onderzoeken, kunnen onderzoekers mutaties schatten die relevant zijn voor de geschiktheid van pathogenen. Omori en Wu willen de impact van ziektedynamiek op Tajima's D begrijpen, wat leidt tot een beter begrip van de pathogeniteit van een mutatie, Ernst, en gastheerspecificiteit.
Het teken van Tajima's D wordt bepaald door zowel natuurlijke selectie als populatiedynamiek. "Tajima's D is gelijk aan 0 als de evolutie neutraal is - geen natuurlijke selectie en een constante populatiegrootte, " Zei Omori. "Een niet-nulwaarde van Tajima's D suggereert natuurlijke selectie en/of verandering in populatiegrootte. Als er geen natuurlijke selectie kan worden aangenomen, Tajima's D is een functie van de bevolkingsomvang. Vandaar, het kan worden gebruikt om tijdreeksveranderingen in populatieomvang te schatten, d.w.z., hoe de epidemie verloopt."
Differentiaalvergelijkingen, die de veranderingssnelheden van het aantal individuen in elk modelcompartiment modelleren, populatiedynamiek kan beschrijven. In dit geval, de populatiedynamiek van gastheren die zijn geïnfecteerd met de stam die een bepaalde sequentie draagt, wordt gemodelleerd door een reeks differentiaalvergelijkingen voor die sequentie, die termen bevatten die de mutatiesnelheid van de ene sequentie naar de andere beschrijven. Bij het opzetten van hun multi-strain SIR-model, Omori en Wu gaan ervan uit dat de populatiedynamiek van de ziekteverwekker evenredig is met de ziektedynamiek. d.w.z., het aantal ziekteverwekkers is evenredig met het aantal geïnfecteerde gastheren. Door deze aanname kan de waarde van Tajima's D veranderen.
In populatiegenetica, onderzoekers geloven dat het teken van Tajima's D wordt beïnvloed door populatiedynamiek. Echter, de auteurs laten zien dat in het geval van een SIR-deterministisch model, Tajima's D is onafhankelijk van de ziektedynamiek (in het bijzonder, onafhankelijk van de parameters voor ziekteoverdrachtssnelheid en ziekteherstelsnelheid). Ze merken ook op dat terwijl Tajima's D vaak negatief is tijdens het begin van een uitbraak, het wordt vaak positief met het verstrijken van de tijd. "Het negatieve teken impliceert geen uitbreiding van de geïnfecteerde populatie in een deterministisch model, " Zei Omori. "We vonden ook dat de afhankelijkheid van Tajima's D van de transmissiedynamiek van de ziekte kan worden toegeschreven aan de stochasticiteit van de transmissiedynamiek op populatieniveau. Deze afhankelijkheid is anders dan de eerder genoemde bestaande veronderstelling over de relatie tussen populatiedynamiek en het teken van Tajima's D."
uiteindelijk, Omori en Wu bewijzen dat Tajima's D in een deterministisch SIR-model volledig wordt bepaald door mutatiesnelheid en steekproefomvang, en dat de tijdsevolutie van de genetische diversiteit van een infectieziektepathogeen volledig wordt bepaald door de mutatiesnelheid. "Dit werk onthulde enige afhankelijkheid van Tajima's D van het (ziekteoverdrachtsdynamiek) basisreproductienummer (R 0 ) en mutatiesnelheid, " zei Omori. "Met de aanname van neutrale evolutie, we kunnen dan de mutatiesnelheid of R . schatten 0 van sequentiegegevens."
Gezien de vraag naar tools die evolutionaire en ziektedynamiek analyseren, de observatie dat Tajima's D afhangt van de stochasticiteit van de dynamiek is nuttig bij het schatten van epidemiologische parameters. Bijvoorbeeld, als sequenties van pathogenen worden bemonsterd uit een kleine uitbraak in een beperkte gastheerpopulatie, dan hangt Tajima's D af van zowel de mutatiesnelheid als R 0 ; daarom, een gezamenlijke schatting van deze parameters uit Tajima's D is mogelijk. "We passen dit theoretische resultaat toe om echte epidemiologische gegevens te analyseren, " Zei Omori. "We moeten ook zien of onze aanpak kan worden gebruikt om de dynamiek van niet-evenwichtsziektes met natuurlijke selectie te onderzoeken."
 Chemici ontwerpen mini-ecosystemen om de werking van medicijnen te testen
Chemici ontwerpen mini-ecosystemen om de werking van medicijnen te testen Blauw-emitterende diode demonstreert beperkingen en belofte van perovskiet-halfgeleiders
Blauw-emitterende diode demonstreert beperkingen en belofte van perovskiet-halfgeleiders S100A9-amyloïden op Aβ-fibrillaire oppervlakken onthuld
S100A9-amyloïden op Aβ-fibrillaire oppervlakken onthuld Levende mitochondriën in ongekend detail gezien:fotobleken in STED-microscopie overwonnen
Levende mitochondriën in ongekend detail gezien:fotobleken in STED-microscopie overwonnen De energie-implicaties van organische radicaalpolymeren
De energie-implicaties van organische radicaalpolymeren
 Superhittegolven van 55 C komen op als de opwarming van de aarde doorzet
Superhittegolven van 55 C komen op als de opwarming van de aarde doorzet Het oplossen van watercrises in steden kan onze klimaatdoelen naar beneden halen
Het oplossen van watercrises in steden kan onze klimaatdoelen naar beneden halen La Niña zal ons een natte zomer bezorgen. Dat is geweldig weer voor muggen
La Niña zal ons een natte zomer bezorgen. Dat is geweldig weer voor muggen Ecuador Amazon-stam wint eerste overwinning tegen oliemaatschappijen
Ecuador Amazon-stam wint eerste overwinning tegen oliemaatschappijen Eenvoudige regel voorspelt wanneer een ijstijd eindigt
Eenvoudige regel voorspelt wanneer een ijstijd eindigt
Hoofdlijnen
- DNA-replicatie vergelijken en contrasteren in prokaryoten en eukaryoten
- Schapen kunnen menselijke gezichten van foto's herkennen
- Ontdekking helpt de nauwkeurigheid van CRISPR-Cas9-genbewerking te verbeteren
- Vooruitgang bereikt in droge conservering van spermacellen van zoogdieren
- Een nieuwe rol voor insuline als vitale factor bij het in stand houden van stamcellen
- Samenstelling van de celwand van de zes koninkrijken
- Vasculaire planten: definitie, classificatie, kenmerken en voorbeelden
- Wat kan albatrospoep ons vertellen over klimaatverandering?
- Vermont ziet hedendaags record voor reproductie van Amerikaanse zeearenden
- Bijna de helft van de LA-huurders is huur verschuldigd
- Menselijk gedrag in operatiekamers loopt parallel met patronen van hiërarchie en geslacht van primaten
- Hoe vrouwen in de media in de jaren zeventig enige loongelijkheid wonnen, en waarom ze vandaag nog steeds vechten

- Nieuwe studie biedt antwoorden om de mobiliteit van ouderen te verbeteren

- Bristol-manuscriptfragmenten van de beroemde Merlijn-legende behoren tot de oudste in hun soort

 Hoe vindt u de fractionele overvloed van een isotoop
Hoe vindt u de fractionele overvloed van een isotoop Nucleaire pasta is de super-macaroni van het heelal
Nucleaire pasta is de super-macaroni van het heelal  Het vermogen van SIRT6 om de groei van kankercellen te onderdrukken wordt uitgelegd
Het vermogen van SIRT6 om de groei van kankercellen te onderdrukken wordt uitgelegd Hoe de wikkeling van een transformator te berekenen
Hoe de wikkeling van een transformator te berekenen  'S Werelds krachtigste microscoop klaar voor onderzoek
'S Werelds krachtigste microscoop klaar voor onderzoek Onderzoeksteam ontwikkelt kosteneffectieve techniek voor massaproductie van hoogwaardig grafeen
Onderzoeksteam ontwikkelt kosteneffectieve techniek voor massaproductie van hoogwaardig grafeen Wat moeten we weten om een asteroïde te delven?
Wat moeten we weten om een asteroïde te delven? 3D-bioprinttechniek kan kunstmatige bloedvaten creëren, orgaanweefsel
3D-bioprinttechniek kan kunstmatige bloedvaten creëren, orgaanweefsel
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

