
Wetenschap
Enkele moleculen in hele cellen visualiseren met een nieuwe draai
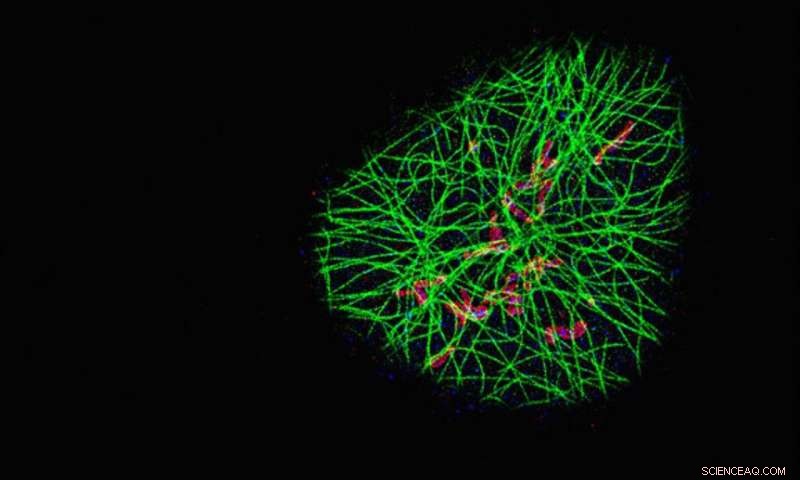
De onderzoekers gebruikten hun SDC-PAINT-methode om het netwerk van cytoskeletale microtubuli-filamenten (groen) en hun nabijheid te visualiseren met twee extra eiwitten genaamd TOM20 (rood) en HSP60 (blauw). Elke afbeelding toont de eiwitten in een ander vlak van de cel vanaf de bovenkant, en de vergrote afbeeldingen aan de onderkant vergelijken de resolutie bereikt met SDC-PAINT (links) met die mogelijk met conventionele confocale microscopie (rechts). Krediet:Florian Schueder, MPI/LMU
Celbiologen gebruiken traditioneel fluorescerende kleurstoffen om cellen en de moleculen erin onder een microscoop te labelen en te visualiseren. Met verschillende superresolutiemicroscopiemethoden, ze kunnen zelfs afzonderlijke moleculen en hun complexe interacties met elkaar verlichten. Echter, de microscopie-hardware waarmee ze dit kunnen doen, is zeer gespecialiseerd en duur en daarom relatief zeldzaam in laboratoria over de hele wereld, en de werking van dergelijke microscopen is ontmoedigend, omdat het unieke vaardigheden vereist.
Ralf Jungmann, doctoraat, een alumnus van het Wyss Institute for Biologically Inspired Engineering van Harvard en momenteel professor aan de Ludwig Maximilian University (LMU) en het Max Planck Institute (MPI) of Biochemistry in Duitsland en Peng Yin, kernfaculteitslid van het Wyss Institute, doctoraat, hebben DNA-PAINT ontwikkeld, een krachtige moleculaire beeldvormingstechnologie die voorbijgaande DNA-DNA-interacties omvat om fluorescerende kleurstoffen nauwkeurig te lokaliseren met superresolutie. Echter, hoewel de onderzoekers het potentieel van DNA-PAINT hebben aangetoond door afzonderlijke biomoleculen te visualiseren, zoals eiwitten, in vaste cellen op een vaste korte afstand, de technologie kon nog niet worden toegepast om moleculen diep in cellen te onderzoeken.
Nutsvoorzieningen, De teams van Jungmann en Yin rapporteren gezamenlijk een oplossing om deze beperking te overwinnen. In hun nieuwe studie ze hebben de DNA-PAINT-technologie aangepast aan microscopen die wijdverbreid zijn in celbiologische laboratoria, confocale microscopen genoemd, en die door onderzoekers worden gebruikt om hele cellen en dikkere weefsels met een lagere resolutie in beeld te brengen. Het team van MPI/Wyss Institute toont aan dat de methode een verscheidenheid aan verschillende moleculen kan visualiseren, inclusief combinaties van verschillende eiwitten, RNA's en DNA door de hele diepte van hele cellen met superresolutie. Gepubliceerd in Natuurcommunicatie , de aanpak zou de deur kunnen openen voor gedetailleerde lokalisatiestudies van één molecuul op veel gebieden van celonderzoek.
De DNA-PAINT-benadering hecht een DNA "ankerstreng aan het molecuul van belang. Vervolgens hecht een met kleurstof gelabelde DNA "imagerstreng" met een complementaire sequentie tijdelijk aan het anker en produceert een fluorescerend signaal, die optreedt als een gedefinieerde knipperende gebeurtenis op enkele moleculaire plaatsen. Omdat "knipperen" precies af te stemmen is, moleculen die slechts nanometers van elkaar verwijderd zijn, kunnen worden onderscheiden - aan het hogere resolutie-einde van superresolutie.
"Onze nieuwe aanpak, SDC-VERF, integreert de veelzijdige superresolutiemogelijkheden van DNA-PAINT met de optische snijfuncties van confocale microscopen. Zo creëerden we de middelen om de hele diepte van een cel te onderzoeken, en om de moleculen erin te visualiseren op nanometerschaal, " zei Jungmann. Het team bracht de aanwezigheid van verschillende combinaties van eiwitten in hele cellen in kaart en ging verder dan dat. "Door onze etiketteringsbenaderingen te diversifiëren, we visualiseerden ook verschillende soorten individuele biomoleculen in de chromosoombevattende kern, inclusief sequenties in het DNA, eiwitten gebonden aan DNA of het membraan dat de kern omsluit, evenals nucleaire RNA's, " voegt Yin toe, die ook co-leider is van het Molecular Robotics Initiative van het Wyss Institute, en hoogleraar systeembiologie aan de Harvard Medical School. .
In principe, confocale microscopen gebruiken zogenaamde pinholes om ongewenste onscherpe fluorescentie van beeldvlakken boven en onder het brandvlak te elimineren. Door het monster te scannen, vliegtuig na vliegtuig, onderzoekers kunnen de fluorescentiesignalen die worden uitgezonden door molecuulgebonden kleurstoffen over de hele diepte verzamelen. specifiek, het team van het MPI/Wyss Institute ontwikkelde de techniek voor "Spinning Disk Confocal" (SDC)-microscopen die fluorescentiesignalen van een heel vlak in één keer detecteren door ze te detecteren door een roterende schijf met meerdere gaatjes. Bovendien, "om 3D-superresolutie te bereiken, we hebben een extra lens in het detectiepad geplaatst, waarmee we sub-diffractie-beperkte resolutie in de derde dimensie kunnen archiveren", aldus eerste auteur Florian Schueder, een afgestudeerde student die werkte met Jungmann die ook werkte met het Wyss Institute-team van Yin als onderdeel van zijn masterscriptie.
"Deze toevoeging kan eenvoudig worden aangepast door fabrikanten van SDC-microscopen; dus implementeren we in principe superresolutiemicroscopie zonder complexe hardwarewijzigingen in microscopen die algemeen beschikbaar zijn voor celbiologen van alle locaties van biomedisch onderzoek. De aanpak heeft dus het potentieel om super -resolutie beeldvorming door hele cellen en weefsels, ’ zei Jungmann.
"Met deze belangrijke vooruitgang, superresolutiemicroscopie en DNA-PAINT zouden toegankelijker kunnen worden voor biomedische onderzoekers, het versnellen van onze inzichten in de functie van individuele moleculen en de processen die ze in cellen aansturen, " zei Donald Ingber, oprichter van het Wyss Institute, MD, doctoraat, die ook de Judah Folkman Professor of Vascular Biology is aan de HMS en het Vascular Biology Program aan het Boston Children's Hospital, evenals hoogleraar bio-engineering aan SEAS.
 Chemici testen een nieuwe nanokatalysator voor het verkrijgen van waterstof
Chemici testen een nieuwe nanokatalysator voor het verkrijgen van waterstof Engineering hartkleppen voor velen
Engineering hartkleppen voor velen Machine learning-techniek versnelt de bepaling van de kristalstructuur
Machine learning-techniek versnelt de bepaling van de kristalstructuur Hoe een atoom in kaart te brengen
Hoe een atoom in kaart te brengen  Verbeterde osteogene activiteit van pre-osteoblasten op aan het oppervlak gemodificeerde 3D-geprinte steigers
Verbeterde osteogene activiteit van pre-osteoblasten op aan het oppervlak gemodificeerde 3D-geprinte steigers
 Nieuwe modellering zal licht werpen op het effect van beleidsbeslissingen op migratie door zeespiegelstijging
Nieuwe modellering zal licht werpen op het effect van beleidsbeslissingen op migratie door zeespiegelstijging Welk type ecosysteem gebruikt een anaconda
Welk type ecosysteem gebruikt een anaconda  Droogte van de eeuw in de Middeleeuwen - met parallellen met de klimaatverandering vandaag?
Droogte van de eeuw in de Middeleeuwen - met parallellen met de klimaatverandering vandaag? Droogte en herstel in Californië dagen de kansen van meerdere eeuwen uit
Droogte en herstel in Californië dagen de kansen van meerdere eeuwen uit Mensen in Afrikaanse steden nemen de leiding over hun watervoorziening - en het werkt
Mensen in Afrikaanse steden nemen de leiding over hun watervoorziening - en het werkt
Hoofdlijnen
- Controversiële onkruidverdelger houdt EU in de knoop (Update)
- Moet het houden van reptielen en amfibieën als huisdier worden beperkt?
- Nieuwe tool kan helpen de kwaliteit te behouden tijdens de kaasproductie
- EP-leden dringen aan op onderzoek naar de invloed van Monsanto op onderzoek
- Uitwerpselen van verstrikte Noord-Atlantische walvissen onthullen torenhoge stressniveaus
- Wat zijn de kenmerken van een cel die een interfase ondergaat?
- Het grote structurele voordeel Eukaryoten hebben over prokaryoten
- 10 manieren waarop gegevens werden gebruikt om ziekten te bestrijden
- Twee hersengebieden werken samen om vinken te helpen weten wanneer en hoe ze hun liedjes moeten afstemmen op specifieke situaties
- Elektronen kijken met extreem ultraviolet licht

- Project creëert krachtiger, veelzijdige ultrasnelle laserpuls

- Ongekende plasmalens voor lasers met hoge intensiteit

- Onderzoekers plagen de unieke chemische vingerafdruk van de meest agressieve vrije radicalen in levende wezens
- 10 redenen waarom het multiversum een reële mogelijkheid is

 Onderzoekers kwantificeren de impact van autonome voertuigen op het verkeer
Onderzoekers kwantificeren de impact van autonome voertuigen op het verkeer IJzerrijke sterren herbergen planeten met een kortere periode
IJzerrijke sterren herbergen planeten met een kortere periode Ingenieurs gebruiken grafieknetwerken om eigenschappen van moleculen en kristallen nauwkeurig te voorspellen
Ingenieurs gebruiken grafieknetwerken om eigenschappen van moleculen en kristallen nauwkeurig te voorspellen Hoe schaaldieren ademen?
Hoe schaaldieren ademen?  Amerikaanse astronaut keert terug naar aarde na langste missie van vrouw
Amerikaanse astronaut keert terug naar aarde na langste missie van vrouw Wat is het verschil tussen een sporen- en een stuifmeelkorrel?
Wat is het verschil tussen een sporen- en een stuifmeelkorrel?  Duitsers ventileren VW-woede in massaal dieselgate-pak
Duitsers ventileren VW-woede in massaal dieselgate-pak Onderzoekers ontwikkelen nieuwe materialen voor energie en detectie
Onderzoekers ontwikkelen nieuwe materialen voor energie en detectie
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- Italian | Portuguese | Swedish | Dutch | Danish | Norway | French | Spanish | German |

-
Wetenschap © https://nl.scienceaq.com

