
Wetenschap
Machine learning-techniek versnelt de bepaling van de kristalstructuur
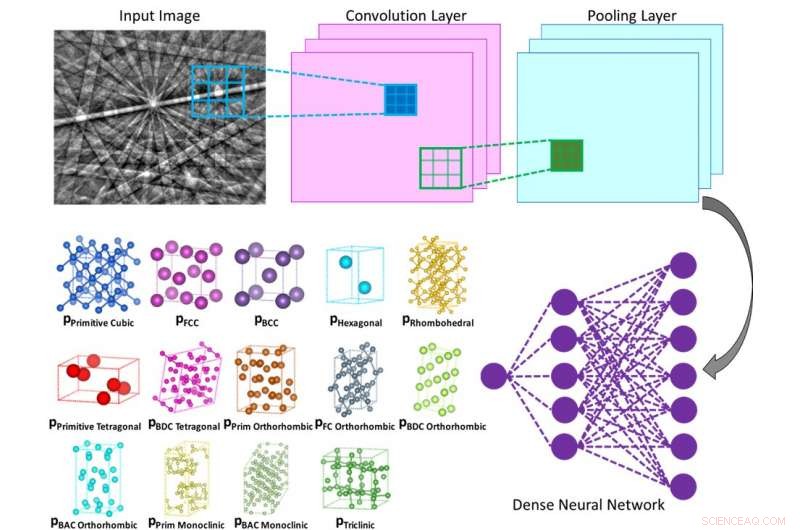
Illustratie van de innerlijke werking van een convolutief neuraal netwerk dat de kans berekent dat het ingangsdiffractiepatroon tot een bepaalde klasse behoort (bijv. Bravais-rooster of ruimtegroep). Krediet:Vecchio-lab / Wetenschap
Nano-ingenieurs van de Universiteit van Californië in San Diego hebben een computergebaseerde methode ontwikkeld die het minder arbeidsintensief zou kunnen maken om de kristalstructuren van verschillende materialen en moleculen te bepalen, inclusief legeringen, eiwitten en geneesmiddelen. De methode maakt gebruik van een machine learning-algoritme, vergelijkbaar met het type dat wordt gebruikt in gezichtsherkenning en zelfrijdende auto's, om onafhankelijk elektronendiffractiepatronen te analyseren, en doe dit met een nauwkeurigheid van ten minste 95%.
Het werk is gepubliceerd in het nummer van 31 januari van: Wetenschap .
Een team onder leiding van UC San Diego nano-engineering professor Kenneth Vecchio en zijn Ph.D. leerling Kevin Kaufmann, wie is de eerste auteur van het artikel, de nieuwe aanpak ontwikkeld. Hun methode omvat het gebruik van een scanning-elektronenmicroscoop (SEM) om elektronen-terugverstrooiingsdiffractie (EBSD) -patronen te verzamelen. In vergelijking met andere elektronendiffractietechnieken, zoals die in transmissie-elektronenmicroscopie (TEM), Op SEM gebaseerde EBSD kan op grote monsters worden uitgevoerd en op meerdere lengteschalen worden geanalyseerd. Dit levert lokale submicron-informatie in kaart gebracht op centimeterschalen. Bijvoorbeeld, een modern EBSD-systeem maakt bepaling van fijnkorrelige korrelstructuren mogelijk, kristal oriëntaties, relatieve restspanning of spanning, en andere informatie in een enkele scan van het monster.
Echter, het nadeel van commerciële EBSD-systemen is het onvermogen van de software om de atomaire structuur van de kristallijne roosters in het te analyseren materiaal te bepalen. Dit betekent dat een gebruiker van de commerciële software maximaal vijf kristalstructuren moet selecteren waarvan wordt aangenomen dat ze in het monster aanwezig zijn en vervolgens probeert de software waarschijnlijke overeenkomsten met het diffractiepatroon te vinden. De complexe aard van het diffractiepatroon zorgt er vaak voor dat de software valse structuurovereenkomsten vindt in de door de gebruiker geselecteerde lijst. Als resultaat, de nauwkeurigheid van de bepaling van het roostertype door de bestaande software is afhankelijk van de ervaring van de operator en de voorkennis van hun monster.
De methode die het team van Vecchio heeft ontwikkeld, doet dit allemaal autonoom, aangezien het diepe neurale netwerk elk diffractiepatroon onafhankelijk analyseert om het kristalrooster te bepalen, uit alle mogelijke roosterstructuurtypes, met een hoge mate van nauwkeurigheid (meer dan 95%).
Een breed scala aan onderzoeksgebieden, waaronder farmacologie, structurele biologie, en geologie zullen naar verwachting profiteren van het gebruik van vergelijkbare geautomatiseerde algoritmen om de hoeveelheid tijd die nodig is voor kristalstructurele identificatie te verminderen, aldus onderzoekers.
 Kunstmatige cyanobacteriële biofilm kan de productie van groene ethyleen meer dan een maand ondersteunen
Kunstmatige cyanobacteriële biofilm kan de productie van groene ethyleen meer dan een maand ondersteunen Leren van fotosynthese:synthetische circuits kunnen lichtenergie oogsten
Leren van fotosynthese:synthetische circuits kunnen lichtenergie oogsten Het moleculaire canvas schilderen in superresolutie
Het moleculaire canvas schilderen in superresolutie Gebruik maken van de slagvastheid van 's werelds hardste beton voor rampenpreventie
Gebruik maken van de slagvastheid van 's werelds hardste beton voor rampenpreventie Wetenschappers voorspellen nieuwe, moeilijk, en superharde ternaire verbindingen
Wetenschappers voorspellen nieuwe, moeilijk, en superharde ternaire verbindingen
 Klimaatverandering kan leiden tot meer aardverschuivingen in Hooggebergte Azië
Klimaatverandering kan leiden tot meer aardverschuivingen in Hooggebergte Azië Het Lidar-netwerk heeft de potentie om vervuilende natuurbranden op het hele continent te volgen
Het Lidar-netwerk heeft de potentie om vervuilende natuurbranden op het hele continent te volgen Kan het verwijderen van koolstof uit de atmosfeer ons redden van een klimaatcatastrofe?
Kan het verwijderen van koolstof uit de atmosfeer ons redden van een klimaatcatastrofe? Lijst van onderwater oceaanplanten
Lijst van onderwater oceaanplanten NASA-NOAA-satelliet vindt overschietende toppen, zwaartekrachtgolven in tropische storm Nestor
NASA-NOAA-satelliet vindt overschietende toppen, zwaartekrachtgolven in tropische storm Nestor
Hoofdlijnen
- Stamboom van Homo Sapiens blijft evolueren
- Voorbeelden van stoffen die gefaciliteerde diffusie gebruiken
- Wat is urushiol?
- Mysterieuze nieuwe zeebodemsoorten werpen licht op vroege evolutie van dieren
- Denk je dat je een hersenschudding hebt? Een bloedtest kan het diagnosticeren
- Statistische modellering helpt visserijbeheerders invasieve soorten te verwijderen
- Wat is vergelijkende biochemie?
Vergelijkende biochemie kan een vaag begrip zijn met meerdere betekenissen, alhoewel het boeiende interacties tussen organismen en hun biologieën kan onthullen. Op zijn minst noemen wetenschappers het een interdiscip
- Voor bonobo's, het loont om machtige bondgenoten te hebben
- Energiebesparende LED's stimuleren wereldwijd lichtvervuiling
- Kathodefabricage voor oxide solid-state batterijen bij kamertemperatuur
- Nieuwe inzichten in bacteriële toxines
- De geheimen van oude rotskunst ontdekken met behulp van röntgenzicht

- Nobelprijswinnende techniek zoals Google Earth voor moleculen

- Zelfaangedreven op papier gebaseerde SPED's kunnen leiden tot nieuwe medisch-diagnostische hulpmiddelen

 Thermische oorkappen beschermen de batterijen van mobiele telefoons tegen extreme temperaturen
Thermische oorkappen beschermen de batterijen van mobiele telefoons tegen extreme temperaturen Het belang van creatieve probleemoplossing op de werkvloer
Het belang van creatieve probleemoplossing op de werkvloer Vernietiging van het milieu in verband met de Afrikaanse bevolking roept vragen op over gezinsgroottes
Vernietiging van het milieu in verband met de Afrikaanse bevolking roept vragen op over gezinsgroottes Veelvoorkomend voedseladditief veroorzaakt nadelige gezondheidseffecten bij muizen
Veelvoorkomend voedseladditief veroorzaakt nadelige gezondheidseffecten bij muizen Opruiming Indonesische eilanden levert dagelijks 40 ton afval op
Opruiming Indonesische eilanden levert dagelijks 40 ton afval op Hoe onze biologische klokken synchroon zijn vergrendeld
Hoe onze biologische klokken synchroon zijn vergrendeld Onderzoekers houden stand in een touwtrekken op atoomschaal
Onderzoekers houden stand in een touwtrekken op atoomschaal Nieuwe filmtechniek onthult bacteriële signalering in scherpere resolutie
Nieuwe filmtechniek onthult bacteriële signalering in scherpere resolutie
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Dutch | Danish | Swedish | German | Norway |

-
Wetenschap © https://nl.scienceaq.com

