
Wetenschap
Onderzoekers bouwen een AI die enzymen ontdekt
Hoewel E. coli een van de meest bestudeerde organismen is, is de functie van 30% van de eiwitten waaruit E. coli bestaat nog niet duidelijk onthuld. Hiervoor werd een kunstmatige intelligentie gebruikt om 464 soorten enzymen te ontdekken uit de eiwitten die onbekend waren, en de onderzoekers gingen verder met het verifiëren van de voorspellingen van drie soorten eiwitten die met succes waren geïdentificeerd via in vitro enzymtests.
Een gezamenlijk onderzoeksteam, bestaande uit Gi Bae Kim, Ji Yeon Kim, Dr. Jong An Lee en Distinguished Professor Sang Yup Lee van de afdeling Chemische en Biomoleculaire Technologie van KAIST, en Dr. Charles J. Norsigian en Professor Bernhard O. Palsson van de afdeling Bioengineering van UCSD heeft DeepECtransformer ontwikkeld, een kunstmatige intelligentie die de enzymfuncties kan voorspellen op basis van de eiwitsequentie. Daarnaast heeft het team een voorspellingssysteem opgezet door gebruik te maken van de AI om de enzymfunctie snel en nauwkeurig te identificeren.
Het werk van het team wordt beschreven in het artikel met de titel "Functionele annotatie van enzymcoderende genen met behulp van deep learning met transformatorlagen." Het artikel werd op 14 november gepubliceerd in Nature Communications .
Enzymen zijn eiwitten die biologische reacties katalyseren, en het identificeren van de functie van elk enzym is essentieel voor het begrijpen van de verschillende chemische reacties die voorkomen in levende organismen en de metabolische kenmerken van die organismen.
Het Enzyme Commission (EC)-nummer is een classificatiesysteem voor enzymfuncties, ontworpen door de International Union of Biochemistry and Molecular Biology, en om de metabolische kenmerken van verschillende organismen te begrijpen, is het noodzakelijk om een technologie te ontwikkelen die enzymen en EG-nummers snel kan analyseren. van de enzymen die in het genoom aanwezig zijn.
Er zijn verschillende op deep learning gebaseerde methoden ontwikkeld om de kenmerken van biologische sequenties te analyseren, waaronder de voorspelling van de eiwitfunctie, maar de meeste ervan hebben het probleem van een black box, waarin het gevolgtrekkingsproces van AI niet kan worden geïnterpreteerd.
Er zijn ook verschillende voorspellingssystemen gerapporteerd die AI gebruiken voor het voorspellen van de enzymfunctie, maar deze lossen dit black box-probleem niet op, of kunnen het redeneerproces niet op een fijnkorrelig niveau interpreteren (bijvoorbeeld het niveau van aminozuurresiduen in de enzymsequentie). ).
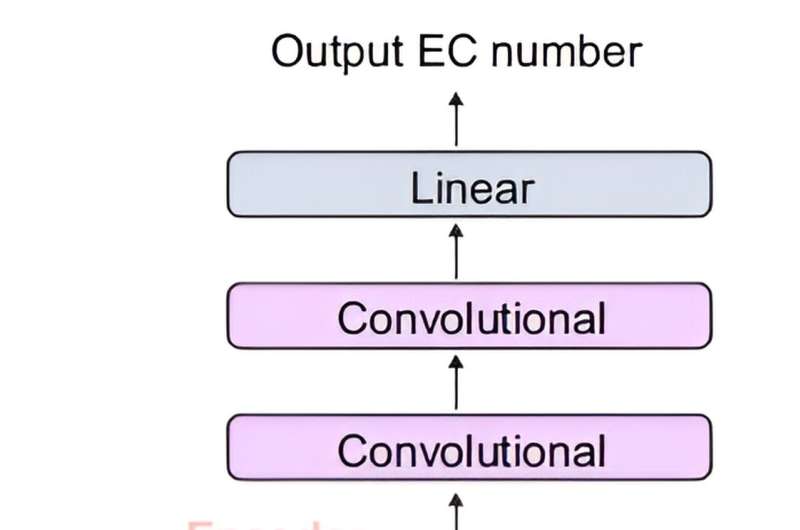
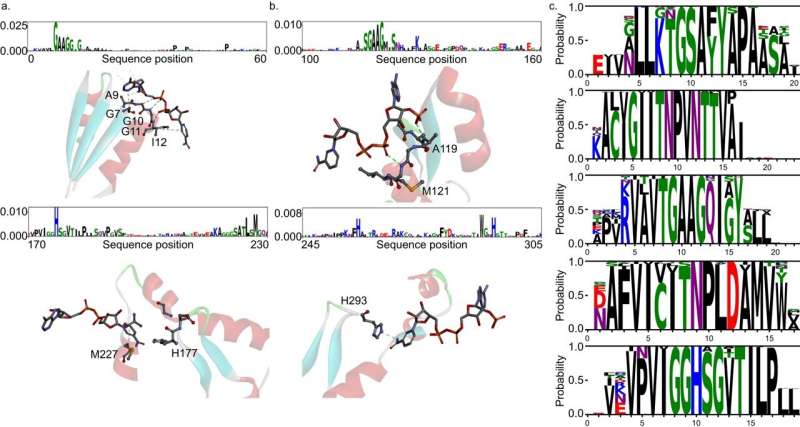
Het gezamenlijke team ontwikkelde DeepECtransformer, een AI die gebruik maakt van deep learning en een eiwithomologie-analysemodule om de enzymfunctie van een bepaalde eiwitsequentie te voorspellen.
Om de kenmerken van eiwitsequenties beter te begrijpen, werd de transformatorarchitectuur, die vaak wordt gebruikt bij natuurlijke taalverwerking, bovendien gebruikt om belangrijke kenmerken over enzymfuncties in de context van de gehele eiwitsequentie te extraheren, waardoor het team de EC nauwkeurig kon voorspellen. nummer van het enzym. De ontwikkelde DeepECtransformator kan in totaal 5360 EC-getallen voorspellen.
Het gezamenlijke team analyseerde de transformatorarchitectuur verder om het inferentieproces van DeepECtransformer te begrijpen, en ontdekte dat de AI bij het inferentieproces informatie gebruikt over katalytisch actieve plaatsen en/of de cofactorbindingsplaatsen die belangrijk zijn voor de enzymfunctie. Door de black box van DeepECtransformer te analyseren, werd bevestigd dat de AI tijdens het leerproces zelfstandig de kenmerken kon identificeren die belangrijk zijn voor de enzymfunctie.
"Door gebruik te maken van het voorspellingssysteem dat we hebben ontwikkeld, konden we de functies voorspellen van enzymen die nog niet waren geïdentificeerd en deze experimenteel verifiëren", zegt Gi Bae Kim, de eerste auteur van het artikel.
"Door DeepECtransformer te gebruiken om voorheen onbekende enzymen in levende organismen te identificeren, zullen we in staat zijn om verschillende facetten die betrokken zijn bij de metabolische processen van organismen nauwkeuriger te analyseren, zoals de enzymen die nodig zijn om verschillende nuttige verbindingen te biosynthetiseren of de enzymen die nodig zijn om plastics biologisch af te breken," voegde hij eraan toe.
"DeepECtransformer, die snel en nauwkeurig enzymfuncties voorspelt, is een sleuteltechnologie in functionele genomica, waardoor we de functie van hele enzymen op systeemniveau kunnen analyseren", zegt professor Sang Yup Lee.
Hij voegde eraan toe:"We zullen het kunnen gebruiken om milieuvriendelijke microbiële fabrieken te ontwikkelen op basis van uitgebreide metabolische modellen op genoomschaal, waardoor de ontbrekende informatie over het metabolisme mogelijk wordt geminimaliseerd."
Meer informatie: Gi Bae Kim et al, Functionele annotatie van voor enzymen coderende genen met behulp van deep learning met transformatorlagen, Nature Communications (2023). DOI:10.1038/s41467-023-43216-z
Journaalinformatie: Natuurcommunicatie
Aangeboden door het Korea Advanced Institute of Science and Technology (KAIST)
 Onderzoekers ontwikkelen 3D-geprinte gelei
Onderzoekers ontwikkelen 3D-geprinte gelei Voorbeelden van zuurbuffers
Voorbeelden van zuurbuffers  Het begrijpen van bederf- en kwaliteitsproblemen kan de Amerikaanse ambachtelijke kaasindustrie verbeteren
Het begrijpen van bederf- en kwaliteitsproblemen kan de Amerikaanse ambachtelijke kaasindustrie verbeteren Elektriciteit opwekken in estuaria met licht en osmose
Elektriciteit opwekken in estuaria met licht en osmose Maak een lijst van enkele factoren die de diffusiesnelheid zouden verhogen
Maak een lijst van enkele factoren die de diffusiesnelheid zouden verhogen
 Vissen dragen jaarlijks ongeveer 1,65 miljard ton koolstof bij in uitwerpselen en andere stoffen
Vissen dragen jaarlijks ongeveer 1,65 miljard ton koolstof bij in uitwerpselen en andere stoffen Nachtvlinders met markeringen van een kruis op Wings
Nachtvlinders met markeringen van een kruis op Wings Verschillen tussen Bison & Cattle
Verschillen tussen Bison & Cattle  Onderzoekers vergelijken globale temperatuurvariabiliteit in glaciale en interglaciale perioden
Onderzoekers vergelijken globale temperatuurvariabiliteit in glaciale en interglaciale perioden Hoe verontreiniging te identificeren
Hoe verontreiniging te identificeren
Hoofdlijnen
- Samengestelde ogen een continu kenmerk van evolutie
- Ga je dat eten? Onderzoek onthult dat hondenpoep een belangrijk onderdeel is van het dieet van vossen
- Voordelen en nadelen van klonen
- Wat is beter voor uw hond:brokjes of rauw vlees? Onderzoek levert verrassende gezondheidsresultaten op
- Onderzoekers onthullen mechanismen van de accumulatie van organische koolstof in de bodem in verzuurde bosbodems
- Celademhalingsexperimenten
- Woedend debat:houdt het ruimen van wolven stroperij tegen?
- Proteomische analyse laat zien hoe fosfiet bijdraagt aan de strijd tegen chemisch resistente afsterving
- Identificatie van een nieuwe familie van op licht reagerende eiwitten
- Het bereik van therapeutische antilichamen vergroten
- Nieuwe software brengt cryo-EM-kaarten met een lagere resolutie in beeld

- Wetenschappers ontrafelen het mysterie van hoe straling metaal verzwakt, één atoom tegelijk

- Chemici ontwikkelen motion capture-achtige technologie voor het volgen van eiwitvorm
- Nieuw type binnenzonnecellen voor slimme verbonden apparaten

 Kenmerken van zeewier
Kenmerken van zeewier Het verschil tussen tong en bot
Het verschil tussen tong en bot  Afbeelding:De capsule van de testploeg van Orion
Afbeelding:De capsule van de testploeg van Orion Latino's, Zwarten worden minder beïnvloed door studievrienden
Latino's, Zwarten worden minder beïnvloed door studievrienden Chemici nemen de plek waar water en lucht samenkomen onder de loep
Chemici nemen de plek waar water en lucht samenkomen onder de loep SDO ziet twee maantransities in de ruimte
SDO ziet twee maantransities in de ruimte Deep learning maakt gebruik van stroomafvoer om de doorlaatbaarheid van de waterscheiding onder het oppervlak te schatten
Deep learning maakt gebruik van stroomafvoer om de doorlaatbaarheid van de waterscheiding onder het oppervlak te schatten Meer rendement op retargetingcampagnes voor e-commerce
Meer rendement op retargetingcampagnes voor e-commerce
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

