
Wetenschap
Internationaal team kraakt genomische code voor vroegste vormen van terrestrische planten
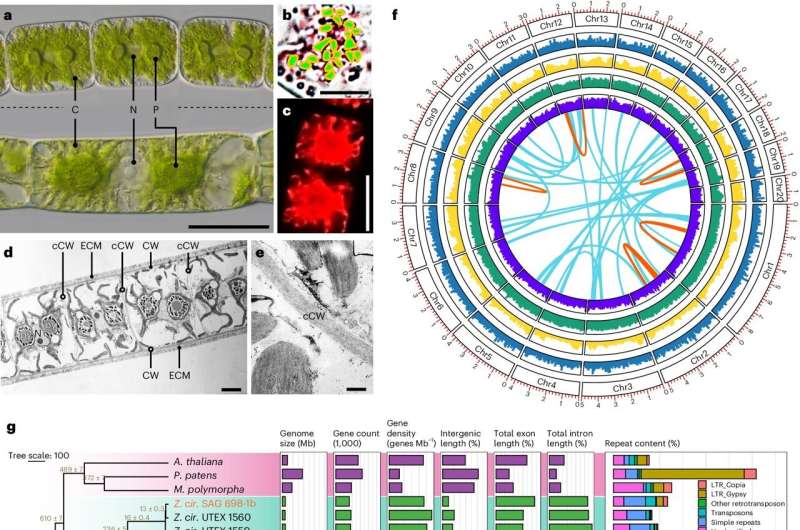
Het plantenleven verscheen ongeveer 550 miljoen jaar geleden voor het eerst op het land, en een internationaal onderzoeksteam onder leiding van computerbioloog Yanbin Yin van de Universiteit van Nebraska-Lincoln heeft de genomische code van zijn bescheiden begin gekraakt, die al het andere aardse leven op aarde mogelijk heeft gemaakt. inclusief mensen.
Het team – ongeveer vijftig wetenschappers in acht landen – heeft de eerste genomische sequentie gegenereerd van vier stammen van Zygnema-algen, de nauwste levende verwanten van landplanten. Hun bevindingen werpen licht op het vermogen van planten om zich aan te passen aan de omgeving en bieden een rijke basis voor toekomstig onderzoek.
De studie werd op 1 mei gepubliceerd in het tijdschrift Nature Genetics.
“Dit is een evolutionair verhaal”, zegt Yin, die samen met een wetenschapper uit Duitsland het onderzoeksteam leidde. "Het beantwoordt de fundamentele vraag hoe de vroegste landplanten zijn geëvolueerd uit zoetwateralgen in het water."
Yins laboratorium in het Nebraska Food for Health Center en het Department of Food Science and Technology heeft een lange geschiedenis in het bestuderen van koolhydraten uit plantencelwanden, een belangrijk onderdeel van voedingsvezels voor mensen en boerderijdieren; lignocellulosen voor de productie van biobrandstoffen; en natuurlijke barrières om gewassen te beschermen tegen ziekteverwekkers en omgevingsstress.
Al het huidige plantenleven op het land is voortgekomen uit een eenmalige evolutionaire gebeurtenis die bekend staat als de terrestrialisatie van planten uit oude zoetwateralgen. De eerste landplanten, bekend als embryophyta binnen de clade van streptophyta, verschenen ongeveer 550 miljoen jaar geleden op het land. Hun komst veranderde het oppervlak en de atmosfeer van de planeet fundamenteel.
Ze maakten al het andere leven op aarde, inclusief mensen en dieren, mogelijk door te dienen als evolutionaire basis voor toekomstige flora en voedsel voor fauna.
De onderzoekers werkten met vier algenstammen van het geslacht Zygnema:twee uit een cultuurcollectie in de Verenigde Staten en twee uit Duitsland. Wetenschappers hebben een reeks geavanceerde DNA-sequencingtechnieken gecombineerd om de volledige genoomsequenties van deze algen te bepalen.
Deze methoden stelden wetenschappers in staat complete genomen voor deze organismen te genereren op het niveau van hele chromosomen – iets wat nog nooit eerder bij deze groep algen was gedaan. Het vergelijken van de genomen met die van andere planten en algen leidde tot de ontdekking van een specifieke overvloed aan celwandenzymen, signaalgenen en omgevingsreactiefactoren.

Een uniek kenmerk van deze algen, onthuld door microscopische beeldvorming – uitgevoerd aan de Universiteit van Innsbruck in Oostenrijk, de Universität Hamburg in Duitsland en het Centrum voor Biotechnologie van UNL – is een dikke en zeer kleverige laag koolhydraten buiten de celwanden, de slijmlaag genoemd.
Xuehuan Feng, de eerste auteur van het artikel en een postdoctoraal onderzoeksmedewerker van Husker, ontwikkelde een nieuwe en effectieve DNA-extractiemethode om deze slijmlaag te verwijderen voor zeer zuivere en hoogmoleculaire DNA's.
"Het is fascinerend dat de genetische bouwstenen, waarvan de oorsprong miljoenen jaren ouder is dan de landplanten, zich vermenigvuldigden en diversifieerden in de voorouders van planten en algen, en daarmee de evolutie van meer gespecialiseerde moleculaire machines mogelijk maakten", zegt Iker Irisarri van het Leibniz Instituut voor de Analyse van Biodiversiteitsverandering en co-eerste auteur van het artikel.
De andere co-leider van het team, Jan de Vries van de Universiteit van Göttingen, zei:"Niet alleen bieden we een waardevolle, hoogwaardige hulpbron voor de hele plantwetenschappelijke gemeenschap, die nu deze genoomgegevens kan onderzoeken, onze analyses hebben ingewikkelde informatie blootgelegd verbindingen tussen milieureacties."
De vier meercellige Zygnema-algen behoren tot de klasse Zygnematophyceae, de nauwste levende verwanten van landplanten; het is een klasse van zoetwater- en semi-terrestrische algen met meer dan 4.000 beschreven soorten.
Zygnematophyceae bezit aanpassingen om aardse stressoren te weerstaan, zoals uitdroging, ultraviolet licht, bevriezing en andere abiotische spanningen. De sleutel tot het begrijpen van deze aanpassingen zijn de genoomsequenties. Vóór dit artikel waren genoomsequenties alleen beschikbaar voor vier eencellige Zygnematophyceae.
Yin zei dat dit onderzoek aansluit bij een van de tien grote ideeën van de National Science Foundation – ‘Understanding the Rules of Life’ – om maatschappelijke uitdagingen aan te pakken, van schoon water tot klimaatbestendigheid. De ontdekking is ook van belang voor toegepaste wetenschappen, zoals bio-energie, waterduurzaamheid en koolstofvastlegging.
"Onze gennetwerkanalyses onthullen co-expressie van genen, vooral die voor celwandsynthese en remodificaties die werden uitgebreid en verworven in de laatste gemeenschappelijke voorouder van landplanten en Zygnematophyceae," zei Yin.
"We werpen licht op de diepe evolutionaire wortels van het mechanisme voor het balanceren van omgevingsreacties en meercellige celgroei."
De internationale onderzoekssamenwerking omvat ongeveer vijftig onderzoekers van twintig onderzoeksinstellingen in acht landen:de Verenigde Staten, Duitsland, Frankrijk, Oostenrijk, Canada, China, Israël en Singapore. Andere Husker-onderzoekers in het team zijn Chi Zhang, hoogleraar biologische wetenschappen, en Jeffrey Mower, hoogleraar agronomie en tuinbouw.