
Wetenschap
Moleculaire atlas van een Australisch drakenbrein werpt nieuw licht op meer dan 300 miljoen jaar hersenevolutie

De Australische baardagaam Pogona vitticeps. Credit:Max Planck Instituut voor Hersenonderzoek / G. Laurent
Tegenwoordig houden draken de fans van "Game of Thrones" scherp. Maar ze bieden ook belangrijke inzichten in de evolutie van de hersenen van gewervelde dieren, zoals blijkt uit het werk van Max Planck-wetenschappers aan de hersenen van de Australische baardagaam Pogona vitticeps. De evolutie van gewervelde dieren nam 320 miljoen jaar geleden een grote wending toen vroege tetrapoden (dieren met vier ledematen) overgingen van water naar land, wat uiteindelijk aanleiding gaf tot drie grote clades:de reptielen, de vogels (een uitloper van de reptielenboom) en de zoogdieren. Vanwege de gemeenschappelijke voorouders delen de hersenen van alle tetrapoden een vergelijkbare basale architectuur die tijdens de vroege ontwikkeling is vastgesteld.
Maar hoe variaties op deze gemeenschappelijke "Bauplan" hebben bijgedragen aan clade-specifieke attributen blijft onduidelijk. Neurowetenschappers van het Max Planck Instituut voor Hersenonderzoek in Frankfurt hebben deze vraag aangepakt door een moleculaire atlas van het drakenbrein te genereren en deze te vergelijken met een van muizen. Hun bevindingen suggereren dat, in tegenstelling tot wat vaak wordt gedacht dat een zoogdierbrein bestaat uit een oud "reptielachtig" brein aangevuld met nieuwe zoogdierkenmerken, zowel reptielen- als zoogdierhersenen hun eigen clade-specifieke neurontypes en circuits hebben ontwikkeld, uit een gemeenschappelijke voorouderlijke set.
"Neuronen zijn de meest diverse celtypes in het lichaam. Hun evolutionaire diversificatie weerspiegelt veranderingen in de ontwikkelingsprocessen die ze produceren en kan veranderingen veroorzaken in de neurale circuits waartoe ze behoren", zegt prof. Gilles Laurent, directeur van het Max Planck Instituut voor Brain Research die de nieuwe studie leidde, gepubliceerd in Science .
"Afzonderlijke hersengebieden werken bijvoorbeeld niet geïsoleerd, wat suggereert dat de evolutie van onderling verbonden regio's, zoals de thalamus en hersenschors, op de een of andere manier gecorreleerd zou kunnen zijn. Ook een hersengebied bij reptielen en zoogdieren dat is afgeleid van een gemeenschappelijke voorouderlijke structuur kan zo zijn geëvolueerd dat het vandaag de dag voorouderlijk blijft in de ene clade, terwijl het 'modern' is in de andere. Omgekeerd kan het zijn dat beide clades nu een mix bevatten van gemeenschappelijke (oude) en specifieke (roman) neurontypes. Dit zijn het soort vragen dat onze experimenten probeerden aan te pakken", voegt Laurent toe.
Terwijl traditionele benaderingen om ontwikkelingsgebieden en projecties in de hersenen te vergelijken niet de nodige resolutie hebben om deze overeenkomsten en verschillen te onthullen, kozen Laurent en zijn team voor een cellulaire transcriptomische benadering. Met behulp van een techniek genaamd single-cell RNA-sequencing die een groot deel van de RNA-moleculen (transcriptomen) detecteert die aanwezig zijn in afzonderlijke cellen, hebben de wetenschappers een celtype-atlas van de hersenen van de Australische baardagaam Pogona vitticeps gegenereerd en deze vergeleken met bestaande muis hersendatasets.
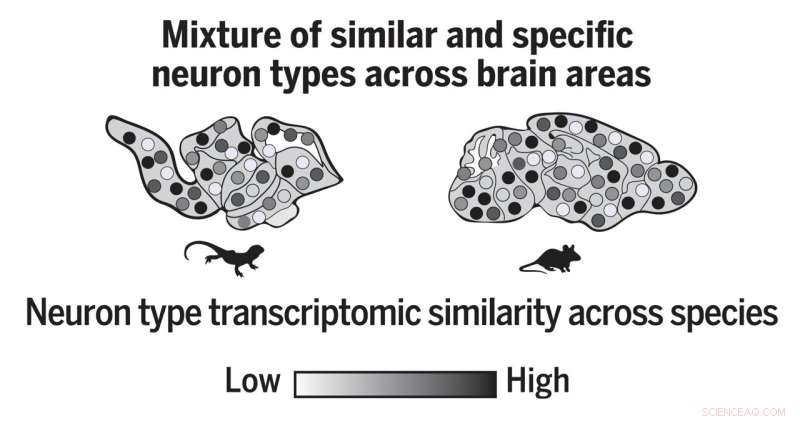
Max Planck-wetenschappers genereerden een celtype-atlas uit de hersenen van een hagedis en integreerden deze gegevens computationeel met muistransciptomics, waardoor werd onthuld dat meerdere hersengebieden mengsels van vergelijkbare en uiteenlopende neuronen bevatten. Krediet:Max Planck Instituut voor Hersenonderzoek / G. Laurent; Hain et al.
Transcriptomische vergelijkingen onthullen gedeelde klassen van neurontypes
"We hebben meer dan 280.000 cellen uit de hersenen van Pogona geprofileerd en 233 verschillende soorten neuronen geïdentificeerd", legt David Hain, afgestudeerde student in het Laurent Lab en mede-eerste auteur van de studie, uit. "Computationele integratie van onze gegevens met muisgegevens onthulde dat deze neuronen transcriptomisch kunnen worden gegroepeerd in gemeenschappelijke families, die waarschijnlijk voorouderlijke neurontypen vertegenwoordigen", zegt Hain. Bovendien ontdekte hij dat de meeste delen van de hersenen een mix van gewone (oude) en specifieke (nieuwe) neurontypes bevatten.
Afgestudeerde student Tatiana Gallego-Flores gebruikte histologische technieken om deze celtypen in het hele drakenbrein in kaart te brengen en observeerde (onder andere) dat neuronen in de thalamus konden worden gegroepeerd in twee transcriptomische en anatomische domeinen, gedefinieerd door hun connectiviteit met andere hersengebieden. Omdat deze verbonden regio's een verschillend lot hebben gehad bij zoogdieren en bij reptielen, waarvan een van deze regio's zeer uiteenlopend is, bleek het zeer interessant om de thalamische transcriptomen van deze twee domeinen te vergelijken. Het onthulde inderdaad dat de transcriptomische divergentie overeenkwam met die van de doelregio's.
"Dit suggereert dat neuronale transcriptomische identiteit enigszins, althans gedeeltelijk, de langeafstandsconnectiviteit van een regio met zijn doelen weerspiegelt. Aangezien we niet de hersenen hebben van oude gewervelde dieren, reconstrueert de evolutie van de hersenen in de afgelopen half miljard jaar vereist het verbinden van zeer complexe moleculaire, ontwikkelings-, anatomische en functionele gegevens op een manier die zelfconsistent is. We leven in zeer opwindende tijden, omdat dit mogelijk wordt", besluit Laurent. + Verder verkennen
Moleculaire atlassen van schildpadden- en hagedishersenen werpen licht op de evolutie van het menselijk brein
 Een nieuwe manier om zachte materialen te combineren
Een nieuwe manier om zachte materialen te combineren Nieuw antibioticum verslaat bacteriële resistentie
Nieuw antibioticum verslaat bacteriële resistentie Hoe geactiveerde houtskool te regenereren
Hoe geactiveerde houtskool te regenereren  Nieuwe herbruikbare hydrogel kan schoon oogsten, ontzilt water uit vochtige lucht boven zeeoppervlakken
Nieuwe herbruikbare hydrogel kan schoon oogsten, ontzilt water uit vochtige lucht boven zeeoppervlakken Drie overeenkomsten tussen een verbinding en een Element
Drie overeenkomsten tussen een verbinding en een Element
 Hoe een klein dorp in Duitsland zichzelf opnieuw uitvond om te overleven
Hoe een klein dorp in Duitsland zichzelf opnieuw uitvond om te overleven Luchtfoto's werpen licht op het verlies van ijs op de Mont Blanc
Luchtfoto's werpen licht op het verlies van ijs op de Mont Blanc Afvalberg India stijgt hoger dan Taj Mahal
Afvalberg India stijgt hoger dan Taj Mahal Veranderingen in de Antarctische mariene ecosystemen
Veranderingen in de Antarctische mariene ecosystemen Hoe vond de vroege mensachtigen voedsel tijdens de Oude Steentijd?
Hoe vond de vroege mensachtigen voedsel tijdens de Oude Steentijd?
Hoofdlijnen
- Nieuwe vlindersoort ontdekt in Rusland met een ongewone set van 46 chromosomen
- Wat is het Forer-effect?
- Hoeveel onontdekte wezens zijn er in de oceaan?
- Wat zijn de functies van longblaasjes in de longen?
- Zalmen helpen hun nakomelingen door te sterven op de paaigronden
- Diverse landschappen in het hart van bijenbescherming
- Voordelen van biosolids verspreid over tientallen jaren van onderzoek
- Een bitter mysterie:wetenschappers sequencen 's werelds oudste plantengenoom van 6000 jaar oude watermeloenzaden
- Vier klassen van macromoleculen belangrijk voor levende dingen
- Centriolen:je kunt cellen niet verdelen zonder ze

- Wetenschappers volgen haaien die DNA-fragmenten uit de zee plukken

- Klimaatveranderingen verkorten de mariene voedselketen voor de kust van Californië

- De wortels van biodiversiteit:hoe eiwitten verschillen tussen soorten

- Duizenden pinguïnkuikens verhongeren op Antarctica

 Internationaal onderzoeksproject onderzoekt lichtgevoelige koolstofnanodeeltjes
Internationaal onderzoeksproject onderzoekt lichtgevoelige koolstofnanodeeltjes Krachtige nieuwe techniek bepaalt tegelijkertijd de chemische samenstelling van nanomaterialen, topografie
Krachtige nieuwe techniek bepaalt tegelijkertijd de chemische samenstelling van nanomaterialen, topografie De ongebruikelijke moleculaire en isotopeninhoud van planetaire nevels
De ongebruikelijke moleculaire en isotopeninhoud van planetaire nevels College Elektrische Projecten
College Elektrische Projecten  Asteroïde-mijnbouw kan over 10-20 jaar beginnen, zegt branche-expert
Asteroïde-mijnbouw kan over 10-20 jaar beginnen, zegt branche-expert Onderzoekers ontwikkelen nieuw proces om een van de sterkste materialen op aarde in 3D te printen
Onderzoekers ontwikkelen nieuw proces om een van de sterkste materialen op aarde in 3D te printen Astronomen ontdekken een zwerm sterrenstelsels die rond een hyperlicht sterrenstelsel draaien
Astronomen ontdekken een zwerm sterrenstelsels die rond een hyperlicht sterrenstelsel draaien Algoritme voor machinaal leren classificeert automatisch slaapstadia van laboratoriummuizen
Algoritme voor machinaal leren classificeert automatisch slaapstadia van laboratoriummuizen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

