
Wetenschap
Technologie die complexe moleculaire interacties simuleert, kan leiden tot betere behandelingen voor kanker en COVID-19
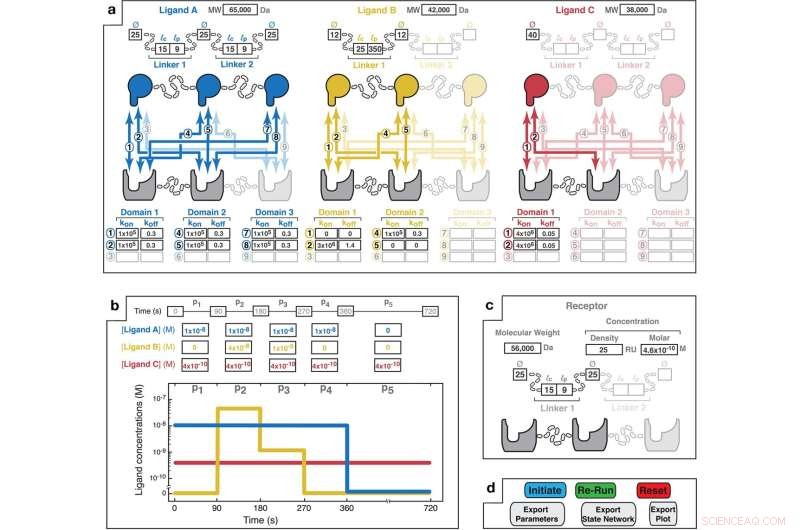
De MVsim-invoerontwerpinterface biedt interactieve parameterspecificatie voor systemen met multivalente, multimoleculaire interactie. een Een point-and-click-interface stelt de gebruiker in staat om het aantal liganden (maximaal drie) en valenties van de ligand(en) en receptor (tot driewaardig) te selecteren die het multivalente systeem vormen. Op basis van het gekozen ontwerp specificeert de gebruiker de structuur van elk van de liganden door het toepasselijke molecuulgewicht (MW) in te voeren; de bindingsdomeindiameters (Ø); de contourlengtes (lc van de linkers (d.w.z. de maximale end-to-end afstand; bijv. 3,5 en 1,5 per aminozuur voor respectievelijk een willekeurige spoel en alfa-helix); en de persistentielengtes (lp) van de linkers. Verder worden de toepasselijke combinatorische interacties (genummerd van 1 tot 9) die uniek zijn voor elke receptor-ligand-paring gemarkeerd. Parametervelden maken de invoer van monovalente snelheidsconstanten voor elke paarsgewijze interactie mogelijk. Niet-bindende interacties kunnen worden aangegeven met k aan en k uit waarden van nul (bijv. zoals geïllustreerd met Ligand B in geel voor interacties "1" en "5"). b Met een invoerveld kan de gebruiker patronen van de totale, bulkligandconcentraties specificeren. Een associatiefase vindt plaats tijdens perioden van niet-nul bulkligandconcentratie (bijv. 90-270 s voor ligand B). Dissociatiefasen treden op wanneer het ligand uit de bulkoplossing wordt verwijderd (bijv. 360-720 s voor ligand A). Hier wordt ligand C gespecificeerd als continu aanwezig in oplossing gedurende de 720 s van het tijdsverloop van de interactie. Het grafische display maakt visualisatie van het gespecificeerde pulspatroon van de bulkconcentratie mogelijk. c Gebruikersinvoerparameters voor de receptor. Receptorconcentratie kan worden gespecificeerd als een SPR-nabootsende oppervlaktedichtheid (gemeten in RU; waarbij 1 RU gelijk is aan ~1 pg/mm 2 ) of een molaire concentratie. Receptortopologie wordt gespecificeerd in dezelfde vorm als hierboven beschreven voor de liganden. d De MVsim controller-tabblad maakt het starten, herhalen en exporteren van bindingssimulaties mogelijk. "Initieren" voert een simulatie uit. "Re-run" voert een verkorte simulatie uit die wordt gebruikt wanneer er geen wijzigingen zijn aangebracht in de valentie of topologie van het systeem. "Reset" start de app opnieuw en wist de invoerparameters van de gebruiker uit alle velden. Krediet:Natuurcommunicatie (2022). DOI:10.1038/s41467-022-32496-6
Een team onder leiding van biomedische ingenieurs van de Universiteit van Minnesota Twin Cities heeft een universeel toegankelijke applicatie ontwikkeld die complexe moleculaire interacties kan simuleren, waardoor onderzoekers betere behandelingen kunnen ontwerpen voor ziekten zoals kanker en COVID-19.
De paper bouwt voort op een studie die de onderzoekers in 2019 publiceerden. Nu hebben ze de technologie uitgebreid om nog complexere moleculaire interacties te simuleren, de applicatie gemakkelijk te gebruiken voor niet-experts en hun bevindingen toegepast om licht te werpen op hoe de SARS -CoV-2-virus infecteert het lichaam.
De studie is gepubliceerd in Nature Communications , en de app, MVsim genaamd, is gratis beschikbaar voor andere onderzoekers op GitHub.
De simulator voorspelt de sterkte, snelheid en selectiviteit van multivalente interacties, waarbij moleculen betrokken zijn die meerdere bindingsplaatsen hebben en die kunnen worden gebruikt om medicijnen te ontwikkelen voor ziekten, met name kanker en COVID-19.
"Multivalente interacties zijn erg belangrijk in natuurlijke biologische systemen, en ze beginnen nu creatief te worden gebruikt voor het creëren van nieuwe therapeutische geneesmiddelen die hun unieke bindende eigenschappen benutten", zegt Casim Sarkar, senior auteur van het artikel en een professor aan de Universiteit van Minnesota. Afdeling Biomedische Technologie.
"Met multivalente medicijnen kun je in principe cellen heel specifiek targeten op een manier die niet mogelijk is met standaard, monovalente medicijnen, maar er zijn veel variabelen waarmee rekening moet worden gehouden in hun ontwerp en veel van het werk in het veld is tot nu toe gedaan door experimenteel vallen en opstaan", voegde Sarkar eraan toe. "Nu we MVsim gebruiken, kunnen we goede voorspellingen doen die kunnen worden gebruikt om dergelijke therapieën rationeler te ontwerpen."
Veel kankermedicijnen binden niet alleen aan tumorcellen, maar ook aan cellen waar ze niet op gericht zijn, wat vaak ongewenste bijwerkingen voor de patiënt veroorzaakt. Door de specificiteit van multivalente interacties te optimaliseren met behulp van MVsim, kunnen onderzoekers geneesmiddelen ontwerpen die zich specifieker richten op de cellen in een tumor, terwijl de binding aan andere cellen in het lichaam wordt geminimaliseerd.
Een ander voorbeeld is het SARS-CoV-2-virus. Wetenschappers weten dat het virus evolueert om onze cellen beter te infecteren en ons immuunsysteem te ontwijken, maar de moleculaire mechanismen achter hoe het virus dit doet, zijn relatief onbekend. Met behulp van hun MVsim-technologie konden de onderzoekers van de Universiteit van Minnesota dit proces diepgaander onderzoeken en de snelheden blootleggen waarmee individuele bindingsdomeinen binnen het multivalente spike-eiwit van het virus schakelen tussen een cel-infecterende toestand en een immuun-ontwijkende toestand.
"We hebben in wezen een computationele microscoop waarmee we onder de motorkap kunnen kijken en zien wat multivalente eiwitten zoals het SARS-CoV-2-spike-eiwit op moleculair niveau doen", legt Sarkar uit. "This level of molecular detail is hard to capture with a physical experiment. One of the real powers of MVsim is that we can not only learn more about how these systems work but we can also use this tool to design new multivalent interactions for diseases like cancer and COVID-19."
The researchers have already identified potential ways to limit the infectivity of current and future SARS-CoV-2 variants, which they plan to test soon. + Verder verkennen
Ontwikkeld multivalent zelf-geassembleerd bindereiwit tegen SARS-CoV-2 RBD
Hoofdlijnen
- Cellulaire ademhaling in ontkiemende zaden
- Wetenschapsproject over waarom botten rubberachtig worden in azijn
- Een nieuwe rol voor insuline als vitale factor bij het in stand houden van stamcellen
- Onderzoek toont aan dat commerciële oogst van brekende schildpadden leidt tot bevolkingsafname
- Voorbij geluid:roodoogboomkikkers gebruiken geluid en trillingen bij het zoeken naar vrienden en agressie
- Biosfeer: definitie, hulpbronnen, cycli, feiten en voorbeelden
- Driedimensionale puntenwolken geven inzicht in de structurele complexiteit van bossen
- Een driedimensionaal tandmodel maken voor een schoolproject
- Eukaryotische celkarakteristieken
- Wetenschappers vinden potentiële wapens voor de strijd tegen antibioticaresistentie
- Dolfijnen gebruiken kenmerkende fluitjes om andere dolfijnen te vertegenwoordigen, vergelijkbaar met hoe mensen namen gebruiken

- Is lachen besmettelijk?

- Masterchef-techniek blijkt levensreddend te zijn voor bedreigde zeeschildpadeieren

- Oude kikkers in massagraf stierven door te veel seks, zegt nieuw onderzoek

 Plastic-etend enzym kan miljarden tonnen afval op de stortplaats elimineren
Plastic-etend enzym kan miljarden tonnen afval op de stortplaats elimineren Een betere manier om miljoenen eenzame sterren te wegen
Een betere manier om miljoenen eenzame sterren te wegen Japan informeert diplomaten over kernwaterproblemen in Fukushima
Japan informeert diplomaten over kernwaterproblemen in Fukushima Verschrikkelijke bazen veroorzaken race naar de bodem, studie vindt
Verschrikkelijke bazen veroorzaken race naar de bodem, studie vindt  Onderzoekers creëren nieuw metallisch materiaal voor flexibele zachte robots
Onderzoekers creëren nieuw metallisch materiaal voor flexibele zachte robots Studenten dichter bij explosieve zonne-evenementen brengen
Studenten dichter bij explosieve zonne-evenementen brengen Meer dan 90% van het gletsjervolume in de Alpen zou tegen 2100 . verloren kunnen gaan
Meer dan 90% van het gletsjervolume in de Alpen zou tegen 2100 . verloren kunnen gaan Afwisselende stromingen en een oostwaartse jet op hoge breedte verklaren de polaire zeshoek van Saturnus, onderzoekers rapporteren
Afwisselende stromingen en een oostwaartse jet op hoge breedte verklaren de polaire zeshoek van Saturnus, onderzoekers rapporteren
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com


