Wetenschap
Een functionele genomics-database voor onderzoeken naar het microbioom van planten
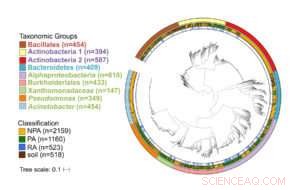
Fylogenetische boom van meer dan 3, 800 hoogwaardige en niet-overtollige bacteriële genomen. Buitenring geeft de taxonomische groep aan, centrale ring geeft de isolatiebron aan, en binnenring geeft de wortel-geassocieerde genomen binnen plant-geassocieerde genomen. Taxonnamen zijn kleurgecodeerd op basis van phylum:groen - Proteobacteria, rood – Firmicutes, blauw -Bacteroides, paars - Actinobacteriën. Krediet:Asaf Levy
Naarmate de wereldbevolking stijgt, geschat op bijna 10 miljard tegen 2050, dat geldt ook voor de noodzaak om de gewasopbrengst te verhogen en voldoende plantmateriaal te produceren voor zowel voedsel als duurzame alternatieve brandstoffen. Om gewasveredelingsstrategieën te helpen verbeteren en uitdagingen te overwinnen, zoals het toleranter maken van planten ten opzichte van marginale gronden, en stress zoals droogte en lage beschikbaarheid van nutriënten, onderzoekers richten zich op het begrijpen en bevorderen van gunstige plant-microbe relaties.
Gepubliceerd op 18 december uitgave 2017 in Natuurgenetica , een team onder leiding van onderzoekers van het Joint Genome Institute (JGI) van het Amerikaanse Department of Energy (DOE), een DOE Office of Science gebruikersfaciliteit, en het Howard Hughes Medical Institute aan de Universiteit van North Carolina in Chapel Hill (UNC) hebben een catalogus van bacteriële genomen gebruikt om kandidaatgenen te identificeren en te karakteriseren die bacteriën helpen zich aan te passen aan plantenomgevingen, specifiek genen die betrokken zijn bij bacteriële wortelkolonisatie.
De meeste onderzoeken in het veld tot nu toe hebben zich gericht op de gemeenschapsstructuur van het plantenmicrobioom, dat wil zeggen "wie is daar, " en minder over de functie, dat wil zeggen "wat ze doen, hoe en wanneer ze het doen." Eerdere studies die de functie hebben overwogen, hebben voornamelijk gekeken naar een enkele gastheer-microbe-interactie, zoals die tussen een Arabidopsis-plant en een pathogeen.
"Als we het juiste microbioom willen ontwikkelen om de plantengroei te ondersteunen, we moeten de echte functie van het microbioom begrijpen en niet alleen sequentiemarkergenen, " zei studie co-eerste auteur Asaf Levy, een onderzoeker bij het JGI. "Hier hebben we een enorme genomische en computationele inspanning gebruikt om de fundamentele en belangrijke vraag te beantwoorden:'Hoe interageert het plantenmicrobioom met de plant?'"
De meeste interactie tussen microben en planten vindt plaats op het grensvlak tussen de wortels en de bodem. Onderzoekers van UNC, Oak Ridge Nationaal Laboratorium, en het Max Planck Instituut isoleerde nieuwe bacteriën uit het wortelmilieu van Brassicaceae (191), populieren (135), en maïs (51). De genomen van deze 377 bacteriële isolaten, plus nog eens 107 enkele bacteriële cellen van wortels van A. thaliana, werden vervolgens gesequenced, geassembleerd, en geannoteerd bij de JGI.
De auteurs combineerden vervolgens de nieuwe genomen met duizenden openbaar beschikbare genomen die de belangrijkste groepen plant-geassocieerde bacteriën vertegenwoordigen, en omvatte bacteriën uit meerdere plantaardige en niet-plantaardige omgevingen, zoals de menselijke darm, ter vergelijking. De resulterende database van 3837 genomen, waarvan 1160 van planten, werd gebruikt in een vergelijkende genomics-analyse.
De onderzoekers identificeerden vervolgens genen die verrijkt zijn in de genomen van plant- en wortel-geassocieerde organismen.
"Het is erg belangrijk voor ons om te begrijpen welke genen en functies microben gebruiken om planten te koloniseren, want alleen dan hebben we een kans om rationeel bruikbare 'plantenprobiotica' te bedenken om ons te helpen meer voedsel- en energiegewassen te verbouwen met minder chemische inputs zoals meststoffen en pesticiden of fungiciden, " zei senior auteur Jeff Dangl, een onderzoeker van het Howard Hughes Medical Institute en de John N. Couch Professor of Biology aan de University of North Carolina in Chapel Hill
Een van de belangrijkste inzichten die uit de studie werden verkregen, was dat plant- en bodem-geassocieerde genomen meestal groter zijn dan controlegenomen van dezelfde clade. Dit bleek gedeeltelijk te wijten te zijn aan verrijking van genen die betrokken zijn bij het suikermetabolisme en -transport, waarschijnlijk een aanpassing aan fotosynthese-afgeleide plantaardige koolstof, gegenereerd door de 'snoepfabrieken' van de natuur, "zei Asaf Levy. Tot 20% van de koolstof die door planten door fotosynthese wordt vastgelegd, wordt via de wortels uitgescheiden als suikers om microben aan te trekken.
Talloze genen die plantfuncties lijken na te bootsen - door te coderen voor "Plant-Resembling PA and RA Domains" of PREPARADO's - werden ook geïdentificeerd. "Het is algemeen bekend dat plantpathogenen eiwitten gebruiken die plantendomeinen nabootsen die nodig zijn voor de immuunfunctie, " zei Dangl. "Stel je voor dat de ziekteverwekker rechtstreeks in de plantencel een eiwit injecteert dat een deel van een bepaald immuunsysteem nabootst. Het is alsof je een gedeeltelijk defect tandwiel in een wiel plaatst - de wielen kunnen niet meer draaien. We denken dat de plant-geassocieerde eiwitdomeinen die we hebben geïdentificeerd op dezelfde manier kunnen werken."
Snel evoluerende genen zijn vaak een kenmerk van een moleculaire wapenwedloop tussen organismen die een omgeving delen. Deze genen worden vaak gebruikt als aanval of verdediging tegen andere organismen. Twee nieuwe snel evoluerende eiwitfamilies geassocieerd met verschillende "levensstijlen" van verwante plant-geassocieerde bacteriën werden in het onderzoek geïdentificeerd. Een, gevonden in commensale bacteriën, werd "Jekyll" genoemd; de andere, gevonden in pathogene bacteriën, heette "Hyde". Met medewerkers van Virginia Tech en ETH (Zwitserland), JGI-wetenschappers ontdekten dat deze laatste zeer efficiënt zijn in het doden van concurrerende bacteriën, mogelijk om deze "Hydes" te helpen de bladnis over te nemen.
De volledige catalogus van nieuwe genomen en plant-geassocieerde genen is beschikbaar voor de onderzoeksgemeenschap via een speciaal webportaal:Genomic Features of Bacterial Adaptation to Plants.
"De database is een waardevolle bron voor de onderzoeksgemeenschap die plant-microbe-interacties bestudeert, omdat het een onbevooroordeelde manier is om potentieel interessante genen te identificeren die betrokken zijn bij interactie met een plant, waaronder veel totaal nieuwe genen. We bestuderen momenteel de functie van veel van deze genen om een beter functioneel begrip van het plantenmicrobioom te krijgen." zei Levy.
 Succesvolle synthese van perovskiet zichtbaar lichtabsorberend halfgeleidermateriaal
Succesvolle synthese van perovskiet zichtbaar lichtabsorberend halfgeleidermateriaal Nobelprijs voor baanbrekende manier om moleculen te bouwen die chemie groener maakten
Nobelprijs voor baanbrekende manier om moleculen te bouwen die chemie groener maakten Ontleding van H2O
Ontleding van H2O  Designer-eiwitten vormen draden en roosters op een mineraal oppervlak
Designer-eiwitten vormen draden en roosters op een mineraal oppervlak Slim water kan helpen bij het terugwinnen van olie
Slim water kan helpen bij het terugwinnen van olie
Hoofdlijnen
- Het belang van het bestuderen van menselijke DNA-genetica
- Geometrie speelt een belangrijke rol in hoe cellen zich gedragen, onderzoekers rapporteren
- Heb jij een dubbelganger? Er is een kans van 1 op een biljoen
- Wat maakt de aarde precies goed voor leven?
- Knoflook kan chronische infecties bestrijden
- Laat je huisdier niet per ongeluk dronken worden dit gekke seizoen (sorry Tiddles)
- Slangenmans gifgewoonte houdt hoop op nieuw tegengif
- Wat gebeurt er als er geen zuurstof beschikbaar is aan het einde van de langzame glycolyse?
- Whodunnit, als Aussie-reptielen uitsterven:studie (update)
 Om te knikken of niet om te gespen
Om te knikken of niet om te gespen Recordbrekende bosbranden in Californië overtreffen 4 miljoen hectare
Recordbrekende bosbranden in Californië overtreffen 4 miljoen hectare Splashdown:Supersonische koudmetaalverbinding in 3D
Splashdown:Supersonische koudmetaalverbinding in 3D Studie:Oorlog, klimaat ontheemde tientallen miljoenen in 2020
Studie:Oorlog, klimaat ontheemde tientallen miljoenen in 2020 Crowdsourced stamboom levert nieuwe inzichten over de mensheid op
Crowdsourced stamboom levert nieuwe inzichten over de mensheid op Veeweiden en andere aangetaste gronden worden nieuwe oliepalmplantages
Veeweiden en andere aangetaste gronden worden nieuwe oliepalmplantages Ammoniak voor brandstofcellen
Ammoniak voor brandstofcellen Heeft Salt Affect Photosynthesis?
Heeft Salt Affect Photosynthesis?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway | Italian |

-
Wetenschap © https://nl.scienceaq.com