
Wetenschap
LipidOz:Nieuwe software maakt identificatie van locaties van dubbele lipidebindingen mogelijk
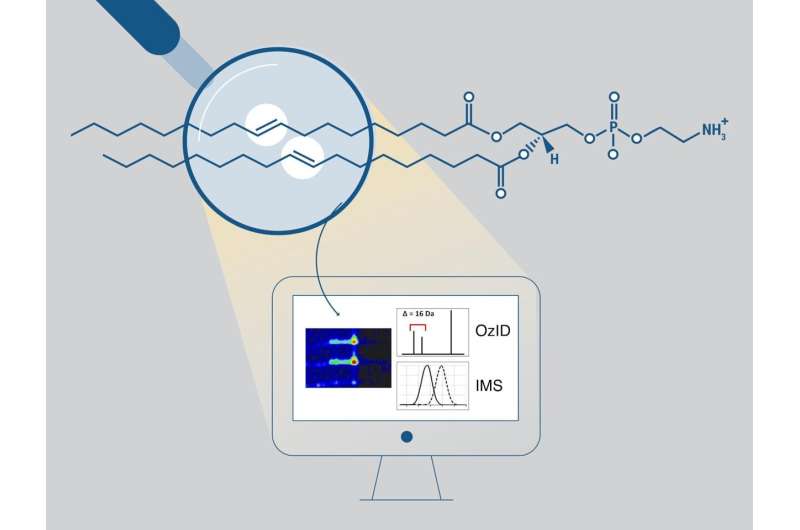
Lipiden zijn een klasse biomoleculen die een belangrijke rol spelen in veel cellulaire processen. Analyses die proberen alle lipiden in een monster te karakteriseren (lipidomics genoemd) zijn cruciaal voor het bestuderen van complexe biologische systemen.
Een belangrijke uitdaging in de lipidomics is het verbinden van de verscheidenheid aan structuren van lipiden met hun biologische functies. De posities van de dubbele bindingen binnen vetzuurketens zijn bijzonder belangrijk. Dit komt omdat ze de fysieke eigenschappen van celmembranen kunnen beïnvloeden en celsignaleringsroutes kunnen moduleren.
Deze informatie wordt niet routinematig gemeten in lipidomics-onderzoeken, omdat hiervoor een ingewikkelde experimentele opstelling vereist die complexe gegevens oplevert. Zo ontwikkelden wetenschappers van het Pacific Northwest National Laboratory (PNNL) een gestroomlijnde workflow om de posities van dubbele bindingen te bepalen. Deze workflow maakt gebruik van zowel automatisering als machine learning-benaderingen.
Hun nieuwe methode, LipidOz, stroomlijnt de data-analyse om de posities van dubbele bindingen te bepalen. Door dit belangrijke onderdeel van de analyse van lipiden aan te pakken, biedt LipidOz onderzoekers een efficiëntere en nauwkeurigere methode voor de karakterisering van lipiden. Het onderzoek is gepubliceerd in het tijdschrift Communications Chemistry .
De ondubbelzinnige identificatie van lipiden wordt bemoeilijkt door de aanwezigheid van moleculaire delen die dezelfde chemische formule hebben maar verschillende fysieke configuraties. De verschillen in deze moleculaire delen omvatten specifiek de lengte van de vetzuuracylketen, de stereospecifiek genummerde (sn) positie en de positie/stereochemie van dubbele bindingen.
Conventionele analyses kunnen de lengtes van de vetzuuracylketens, het aantal dubbele bindingen en – in sommige gevallen – de sn-positie bepalen, maar niet de posities van dubbele koolstof-koolstofbindingen. De posities van deze dubbele bindingen kunnen met groter vertrouwen worden bepaald met behulp van een gasfase-oxidatiereactie genaamd ozon-geïnduceerde dissociatie (OzID), die karakteristieke fragmenten produceert.
De analyse van de uit deze reactie verkregen gegevens is echter complex en repetitief, en er is een gebrek aan ondersteuning door softwaretools. De open-source Python-tool LipidOz bepaalt en wijst automatisch de dubbele bindingsposities van lipiden toe met behulp van een combinatie van traditionele automatisering en deep learning-benaderingen. Nieuw onderzoek toont dit vermogen aan voor standaard lipidenmengsels en complexe lipidenextracten, waardoor praktische toepassing van OzID voor toekomstige lipidomics-studies mogelijk wordt.
Meer informatie: Dylan H. Ross et al., LipidOz maakt geautomatiseerde opheldering van posities van dubbele lipide-koolstof-koolstofbindingen mogelijk op basis van door ozon geïnduceerde dissociatie-massaspectrometriegegevens, Communicatiechemie (2023). DOI:10.1038/s42004-023-00867-9
Journaalinformatie: Communicatiechemie
Geleverd door Pacific Northwest National Laboratory
 Team werpt nieuw licht op fotosynthese
Team werpt nieuw licht op fotosynthese Wat zijn de zes belangrijkste elementen in levende organismen?
Wat zijn de zes belangrijkste elementen in levende organismen?  Wat is het verschil tussen Celsius en Fahrenheit?
Wat is het verschil tussen Celsius en Fahrenheit?  Onderzoekers zetten belangrijke stap richting gonorroevaccin
Onderzoekers zetten belangrijke stap richting gonorroevaccin Chemici maken doorbraak in de chemie van de ontdekking van geneesmiddelen:twee methoden om koolstof te vervangen door een stikstofatoom in een molecuul
Chemici maken doorbraak in de chemie van de ontdekking van geneesmiddelen:twee methoden om koolstof te vervangen door een stikstofatoom in een molecuul
 Wereldwijde wetlands aan de kust moeten landinwaarts verhuizen in strijd tegen klimaatverandering
Wereldwijde wetlands aan de kust moeten landinwaarts verhuizen in strijd tegen klimaatverandering 22 miljoen pond plastic komt elk jaar de Grote Meren binnen
22 miljoen pond plastic komt elk jaar de Grote Meren binnen Bosbrandnevel verdwijnt boven Singapore voor F1
Bosbrandnevel verdwijnt boven Singapore voor F1 WHO schrijft voorschrift voor gezondheid op COP26
WHO schrijft voorschrift voor gezondheid op COP26 Behoud van visbiomassa de sleutel tot het behoud van de biodiversiteit van rifvissen
Behoud van visbiomassa de sleutel tot het behoud van de biodiversiteit van rifvissen
Hoofdlijnen
- Hoe hoog kan een boom worden?
- Landbouwfungicide trekt honingbijen aan, studie vondsten
- Camponotini-mierensoorten hebben hun eigen verschillende microbiomen
- Het dubbele leven van Sp1:een nieuwe rol van eiwitten zet het denken over de impact ervan op kanker op zijn kop
- Whodunnit, als Aussie-reptielen uitsterven:studie (update)
- Onderzoek naar plantenenzymen toont aan dat eiwitten hun structurele rangschikking met verrassend gemak kunnen veranderen
- Anatomische structuren: homoloog, analoog & vestigiaal
- Feiten over de menselijke schedel voor kinderen
- Chili-pinguïns winnen slag in oorlog tegen de mijne
- Virtual reality-systemen gebruiken om scheikunde in 3D te onderwijzen

- Multi-state schakelbare stationaire fase opent nieuwe deuren in chirale scheiding
- Pas op voor mede-bacteriën die geschenken dragen:onderzoek presenteert nieuwe potentiële antimicrobiële middelen
- Nieuw printproces verbetert de 3D-mogelijkheden

- Wereldprimeur:nieuw polymeer gaat wandelen als het verlicht is

 Onderzoekers identificeren genen die zoogdieren van andere dieren onderscheiden
Onderzoekers identificeren genen die zoogdieren van andere dieren onderscheiden Het water kan binnenkort tegen de deur kabbelen, maar toch willen sommige huiseigenaren de boot niet laten schommelen
Het water kan binnenkort tegen de deur kabbelen, maar toch willen sommige huiseigenaren de boot niet laten schommelen Het verschil tussen eenfase- en driefasige elektrische bedrading
Het verschil tussen eenfase- en driefasige elektrische bedrading  Lamprei-tanden voor het eerst geïdentificeerd in archeologische vondsten in Londen
Lamprei-tanden voor het eerst geïdentificeerd in archeologische vondsten in Londen Goedkope legering concurreert met duur platina om brandstofcellen te stimuleren
Goedkope legering concurreert met duur platina om brandstofcellen te stimuleren Parasitisme in de toendra
Parasitisme in de toendra De samenstelling van de maricultuur:onderzoekers onderzoeken wereldwijde trends in de visteelt
De samenstelling van de maricultuur:onderzoekers onderzoeken wereldwijde trends in de visteelt Een eenvoudige manier om materie tot complexe vormen te boetseren
Een eenvoudige manier om materie tot complexe vormen te boetseren
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

