
Wetenschap
Supercomputing van de geheimen in antibiotica voor rundvee
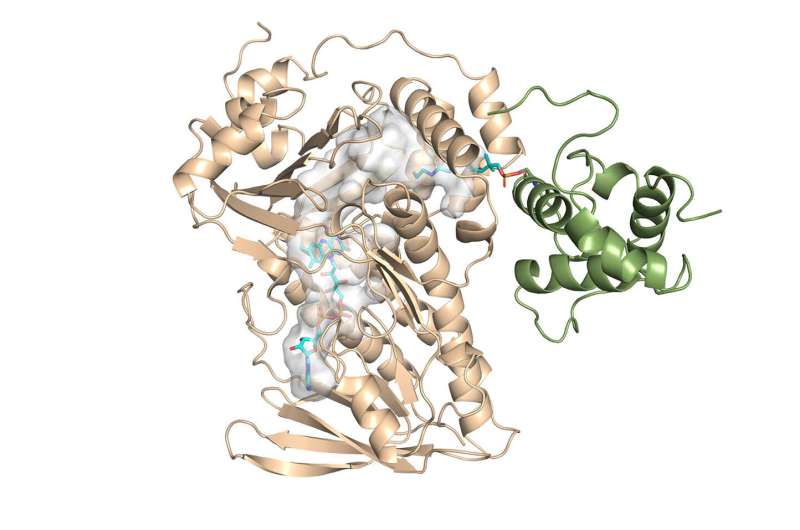
Chemici hebben voor het eerst de kristalstructuur van een sleutelcomponent van het monensin-enzym bepaald en het mechanisme van zijn reactieactiviteit ontsluierd.
"De belangrijkste bevinding was de eerste kristalstructuur voor deze familie van enzymen", zegt Chu-Young Kim, hoogleraar biochemie aan de Universiteit van Illinois Urbana-Champaign (UIUC), die de experimentele kant van het onderzoek leidde. Hij en zijn collega's hebben de kristalstructuur van MonCI opgelost, een sleutelenzym in bodembacteriën dat op natuurlijke wijze monensine synthetiseert.
Lela Vukovic, universitair hoofddocent aan de Universiteit van Texas, in El Paso (UTEP), voerde de computationele onderzoeken uit naar het monensin-onderzoek, gepubliceerd in Nature Communications .
Het initiatief van de University of Texas Research Cyberinfrastructure (UTRC) kende Vukovic supercomputertoewijzingen toe op het Lonestar6-systeem van het Texas Advanced Computing Center (TACC) om deze uitdagingen aan te gaan. UTRC biedt geavanceerde computermogelijkheden aan onderzoekers in alle 14 UT-systeeminstellingen.
Lonestar6 van TACC hielp bij het onthullen van de reactiesequentie die monensine produceert. Dit onderzoek opent de deur voor toekomstig ontwerp van veiligere, effectievere antibiotica.
"We ontdekten dat MonCI wordt gebruikt om drie cruciale epoxidatiereacties uit te voeren," zei Kim. "Dit is zeer ongebruikelijk en heeft gevolgen voor de manier waarop we de bacterie kunnen manipuleren om nieuwe antibiotica te produceren."
Kim, die onlangs UTEP verliet om zich bij UIUC aan te sluiten, raadpleegde het laboratorium van Vukovic met de structuurresultaten en vol nieuwe vragen. Wat hij ontdekte was een interessante opeenvolgende reactie binnen het enzym. Het was echter experimenteel nog steeds onmogelijk om de kristalstructuur van het enzym te verkrijgen met het substraat erin in zijn actieve modus.
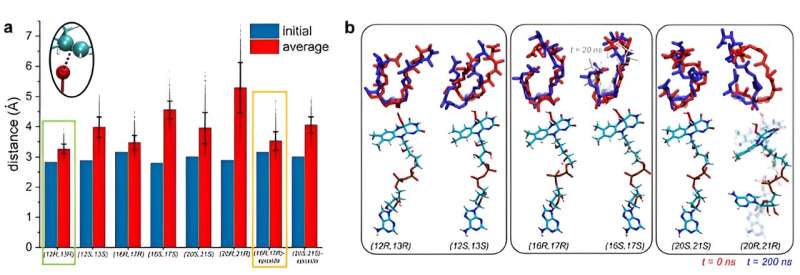
Toen besloten Kim en Vukovic om het enzym en de substaat in simulaties te modelleren wanneer het substraat stabiel is. Als een substraat op een bepaalde positie stabiel is, kan er voor die positie een reactie optreden.
Vukovic en haar studenten Tara Nitka en Anju Yadav ontwikkelden volledige modellen van het grofweg 78.000 atomen tellende systeem op basis van de kristalstructuren bepaald door de onderzoekers.
"De computationele uitdagingen kwamen voort uit het onderzoeken van meerdere systemen om de positie te bepalen waarin premonensine A en zijn geëpoxideerde versies het meest stabiel zijn en het meest waarschijnlijk eerste, tweede en derde epoxidatiereacties ondergaan", aldus Vukovic.
"Supercomputers zijn zeer nuttig geweest bij het karakteriseren van deze biologische moleculen die van nature antibiotica produceren", voegde Vukovic eraan toe. "Zonder hen zouden we de computationele studies niet kunnen uitvoeren. Computationele studies helpen ons deze complexe opeenvolgende reacties die belangrijk zijn voor de samenleving te ontdekken en te begrijpen."
Vukovic voerde haar postdoctoraal onderzoek uit bij UIUC onder wijlen Klaus Schulten, wiens nalatenschap voortleeft in de NAMD-software die zijn groep ontwikkelde en werd gebruikt in deze studie en talloze andere.
"UIUC doet veel werk om NAMD te optimaliseren voor gebruik op de supercomputers van het land, zoals Lonertar6 en Stampede2 bij TACC", zei ze. "NAMD stelde ons in staat om in te zoomen op dit enzym en te zien welke reacties als eerste, tweede en derde plaatsvinden om monensine te genereren."
"Voor de biosynthese van monensine zijn minstens 14 verschillende enzymen nodig, waarvan er één MonCI is", voegde Kim eraan toe. “We moeten ook alle andere enzymen onderzoeken. In de toekomst verwachten we verbeterde versies van monensin te genereren om beter voor vee en pluimvee te zorgen. Bovendien is monensin giftig voor paarden en honden, dus deze boerderijdieren worden soms per ongeluk vergiftigd. en gedood. Daarom is een niet-giftige monensin nodig."
De auteurs zijn Qian Wang, Tara A. Nitka, Anju Yadav en Lela Vukovic van de Universiteit van Texas in El Paso; Ning Liu, Hongli Xiao, Hui Yang en Xi Chen van de Northwest University, China; Irimpan I. Mathews van het SLAC National Accelerator Laboratory; en Chu-Young Kim van de Universiteit van Illinois Urbana-Champaign.
Meer informatie: Qian Wang et al, Triepoxidevorming door een flavine-afhankelijke mono-oxygenase in de biosynthese van monensine, Nature Communications (2023). DOI:10.1038/s41467-023-41889-0
Aangeboden door de Universiteit van Texas in Austin
 Een verbinding die van vitaal belang is voor al het leven heeft waarschijnlijk een rol gespeeld bij het ontstaan van het leven, suggereert syntheseonderzoek
Een verbinding die van vitaal belang is voor al het leven heeft waarschijnlijk een rol gespeeld bij het ontstaan van het leven, suggereert syntheseonderzoek  Polarisatie van broommolecuul in vanadiumoxideclusterholte en nieuwe bromering van alkaan
Polarisatie van broommolecuul in vanadiumoxideclusterholte en nieuwe bromering van alkaan Microtexturering van zachte materialen om waterige microvervuilingen te verwijderen
Microtexturering van zachte materialen om waterige microvervuilingen te verwijderen  Traditionele geheimen om koel te blijven - onderzoek naar Okinawa-textiel
Traditionele geheimen om koel te blijven - onderzoek naar Okinawa-textiel Wetenschappers gebruiken chemische hulpmiddelen om kankerverwekkende eiwitten op te sporen
Wetenschappers gebruiken chemische hulpmiddelen om kankerverwekkende eiwitten op te sporen
 Beving in Mexico veroorzaakt tsunami in de woestijn, 1500 mijl verderop in Death Valley-grot
Beving in Mexico veroorzaakt tsunami in de woestijn, 1500 mijl verderop in Death Valley-grot MABI-instrument kan de concentratie en bron van zwarte koolstofvervuiling in de atmosfeer bepalen
MABI-instrument kan de concentratie en bron van zwarte koolstofvervuiling in de atmosfeer bepalen Slangen identificeren in Pennsylvania
Slangen identificeren in Pennsylvania Door de klimaatverandering zal de Australische bodem CO₂ uitstoten en bijdragen aan de opwarming van de aarde, zo blijkt uit onderzoek
Door de klimaatverandering zal de Australische bodem CO₂ uitstoten en bijdragen aan de opwarming van de aarde, zo blijkt uit onderzoek  Nepal weigert Everest opruiming te midden van pandemische rust
Nepal weigert Everest opruiming te midden van pandemische rust
Hoofdlijnen
- De fabrieksproductie afremmen kan averechts werken, studie vondsten
- Biology Speech Topics
- Voors en tegens van Recombinant DNA Technology
- Functie van T-cellen in de Thymus Gland
- Welke sierplanten presteren het beste voor bestuivers?
- Dode vissen stapelen zich op aan de kusten van de San Francisco Bay Area:een giftige algenbloei is de waarschijnlijke oorzaak
- Parasitaire gewoonte stimuleert structurele variatie in het plastidegenoom en genverlies bij Cuscuta-soorten
- Levenslange bloedproductie is afhankelijk van honderden cellen die zich vóór de geboorte vormen
- Genetische modificatie en genoombewerking zijn afhankelijk van actieve rollen voor onderzoekers en de industrie
- Combinatorische strategie met hoge doorvoer voorgesteld om reactiekatalysatoren voor waterstofontwikkeling te screenen

- Super sterk, biocompatibel materiaal kan kapotte vullingen tot het verleden maken
- Wetenschappers bouwen M29-clustermodelkatalysator
- Nieuw membraanmateriaal kan zuivering van gassen aanzienlijk efficiënter maken

- Parallel gekoppelde celgecentreerde eindig volume thermische rooster Boltzmann-methode op ongestructureerde roosters
 Nog te vroeg om de impact van #MeToo te zeggen
Nog te vroeg om de impact van #MeToo te zeggen Netflix zegt dat het de streamingkroon zal behouden; geen zorgen over Disney Plus en Apple TV Plus
Netflix zegt dat het de streamingkroon zal behouden; geen zorgen over Disney Plus en Apple TV Plus De meest voorkomende organische moleculen in cellen
De meest voorkomende organische moleculen in cellen  De vorm, niet maat, van de hersenen van onze voorouders hebben hen mogelijk geholpen om de Neanderthalers te overleven
De vorm, niet maat, van de hersenen van onze voorouders hebben hen mogelijk geholpen om de Neanderthalers te overleven De weg verlichten naar gerichte therapieën en minder bijwerkingen
De weg verlichten naar gerichte therapieën en minder bijwerkingen Wat zijn de Planten van de Tropische Oceanen?
Wat zijn de Planten van de Tropische Oceanen?  Met big data in vroegmiddeleeuws Europa duiken
Met big data in vroegmiddeleeuws Europa duiken Moleculair-achtige fotochemie van halfgeleider nanokristallen
Moleculair-achtige fotochemie van halfgeleider nanokristallen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

