
Wetenschap
Hoe heb je geprobeerd het uit en weer aan te zetten? werkt voor de chemie, niet alleen voor computers
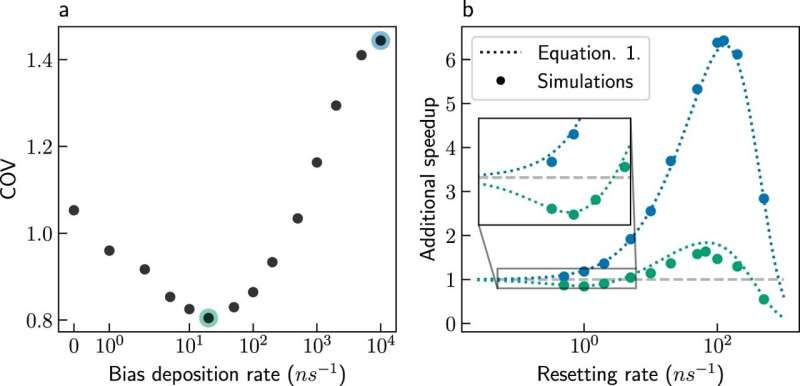
Een nieuwe studie van de Universiteit van Tel Aviv heeft ontdekt dat een bekende praktijk in de informatietechnologie ook kan worden toegepast op de chemie. Onderzoekers ontdekten dat om de bemonstering bij chemische simulaties te verbeteren, u alleen maar hoeft te stoppen en opnieuw te starten.
Het onderzoek werd geleid door Ph.D. student Ofir Blumer, in samenwerking met professor Shlomi Reuveni en Dr. Barak Hirshberg van de Sackler School of Chemistry aan de Universiteit van Tel Aviv. Het onderzoek is gepubliceerd in Nature Communications .
De onderzoekers leggen uit dat simulaties van moleculaire dynamica lijken op een virtuele microscoop. Ze volgen de beweging van alle atomen in chemische, fysische en biologische systemen, zoals eiwitten, vloeistoffen en kristallen. Ze bieden inzicht in verschillende processen en hebben verschillende technologische toepassingen, waaronder medicijnontwerp.
Deze simulaties zijn echter beperkt tot processen die langzamer zijn dan een miljoenste van een seconde en kunnen dus geen langzamere processen beschrijven, zoals eiwitvouwing en kristalkiemvorming. Deze beperking, bekend als het tijdschaalprobleem, is een grote uitdaging in het veld.
Ph.D. student Ofir Blumer zegt:"In ons nieuwe onderzoek laten we zien dat het tijdschaalprobleem kan worden overwonnen door het stochastisch resetten van de simulaties. Het lijkt op het eerste gezicht contra-intuïtief:hoe kunnen de simulaties sneller eindigen als ze opnieuw worden opgestart? Toch blijkt die reactie De tijden variëren aanzienlijk tussen simulaties. In sommige simulaties treden reacties snel op, maar andere simulaties gaan voor lange perioden verloren in tussenliggende toestanden
De onderzoekers combineerden stochastisch resetten ook met Metadynamics, een populaire methode om de simulaties van langzame chemische processen te versnellen. De combinatie maakt een grotere acceleratie mogelijk dan beide methoden afzonderlijk. Bovendien vertrouwt Metadynamica op voorkennis:de reactiecoördinaten moeten bekend zijn om de simulatie te versnellen.
De combinatie van Metadynamica met resetten vermindert de afhankelijkheid van voorkennis aanzienlijk, waardoor beoefenaars van de methode tijd besparen. Ten slotte lieten de onderzoekers zien dat de combinatie nauwkeurigere voorspellingen geeft over de snelheid van langzame processen. De gecombineerde methode werd gebruikt om de simulaties van eiwitvouwing in water met succes te verbeteren, en zal naar verwachting in de toekomst op meer systemen worden toegepast.
Meer informatie: Ofir Blumer et al, Stochastische reset combineren met metadynamica om simulaties van moleculaire dynamica te versnellen, Natuurcommunicatie (2024). DOI:10.1038/s41467-023-44528-w
Journaalinformatie: Natuurcommunicatie
Aangeboden door de Universiteit van Tel-Aviv
 Zout zet een snelle stap voordat het uit het water valt
Zout zet een snelle stap voordat het uit het water valt Spannende tijden voor efficiënte, zware atoomvrije OLED's
Spannende tijden voor efficiënte, zware atoomvrije OLED's Inzichten op atoomniveau helpen de degradatie in brandstofcellen te verminderen en hun levensduur te verlengen
Inzichten op atoomniveau helpen de degradatie in brandstofcellen te verminderen en hun levensduur te verlengen Ontdekking van een oermetabolisch systeem dat ons een glimp geeft van de oorsprong van het leven op aarde
Ontdekking van een oermetabolisch systeem dat ons een glimp geeft van de oorsprong van het leven op aarde Nieuw reagens verbetert het proces van het maken van zwavelhoudende verbindingen die in medicijnen kunnen worden gebruikt
Nieuw reagens verbetert het proces van het maken van zwavelhoudende verbindingen die in medicijnen kunnen worden gebruikt
 NASA's IMERG schat zware regenval in het oosten van de VS
NASA's IMERG schat zware regenval in het oosten van de VS NASA-sneeuwcampagne loopt af voor 2021
NASA-sneeuwcampagne loopt af voor 2021 Nieuw onderzoek onthult het belang van ondergrondse estuaria voor duurzame visserij en aquacultuur
Nieuw onderzoek onthult het belang van ondergrondse estuaria voor duurzame visserij en aquacultuur Het spijsverteringsstelsel van een Kangaroo
Het spijsverteringsstelsel van een Kangaroo Wereldwijde wetlands aan de kust moeten landinwaarts verhuizen in strijd tegen klimaatverandering
Wereldwijde wetlands aan de kust moeten landinwaarts verhuizen in strijd tegen klimaatverandering
Hoofdlijnen
- AI brengt cruciale hiaten aan het licht in het mondiale onderzoek naar antimicrobiële resistentie
- Hoe verschillen schimmelsporen van bacteriële endosporen?
- Hoe erfelijke ziekten werken
- Worden je hersenen moe zoals de rest van je lichaam?
- Hoe het uitsterven van de dinosauriërs de evolutie van planten veranderde
- Een biohybride robot gemaakt van meel en haver zou kunnen fungeren als een biologisch afbreekbare vector voor herbebossing
- Belangrijke feiten over het koninkrijk Monera
- Wat zijn drie primaire doelen van mitose?
- Wat is de volgorde van de gebeurtenissen in de bevruchting van een ei?
- Elektrische velden in kaart brengen om te helpen ontrafelen hoe enzymen werken
- Sommige bestaande geneesmiddelen tegen kanker kunnen gedeeltelijk werken door zich te richten op RNA, studie toont

- Zou goud de sleutel kunnen zijn tot gentherapie voor HIV, bloedaandoeningen toegankelijker?
- Onderzoekers ontwikkelen een reactor die chemicaliën voor altijd kan vernietigen

- Gericht op het verbeteren van verbindingstechnologieën voor lichtgewicht en zeer sterke constructies

 Lego-achtige assemblage van zeolietmembranen verbetert de koolstofopname
Lego-achtige assemblage van zeolietmembranen verbetert de koolstofopname De ladder beklimmen naar levensdetectie
De ladder beklimmen naar levensdetectie Het modificeren van oppervlakken met polymeerketens bevordert een gelijkmatige droging van deeltjesbevattende vloeistoffen
Het modificeren van oppervlakken met polymeerketens bevordert een gelijkmatige droging van deeltjesbevattende vloeistoffen Giftig kwik vergiftigt de Amazone
Giftig kwik vergiftigt de Amazone Gouden nano-sterren voor intracellulaire levering
Gouden nano-sterren voor intracellulaire levering Lineair programmeren in Excel oplossen
Lineair programmeren in Excel oplossen  Andesconcepten van dood en vernieuwing begrijpen
Andesconcepten van dood en vernieuwing begrijpen Team demonstreert kwantumstippen die zichzelf assembleren
Team demonstreert kwantumstippen die zichzelf assembleren
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

