
Wetenschap
Neurale netwerk detecteert eiwit-peptide bindingsplaatsen om de ontdekking van peptidegeneesmiddelen op gang te brengen
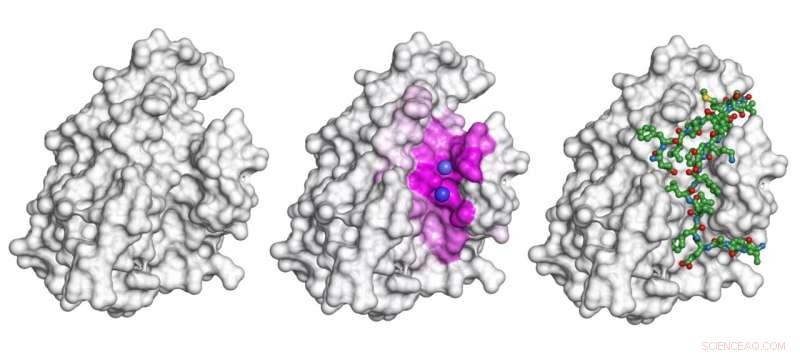
De grijze vorm is een eiwit. Voor het scenario van dit eiwit dat aan het peptide bindt, weergegeven als een groenachtig stick-and-ball-model aan de rechterkant, het model gepresenteerd in de studie benadrukt het oppervlak dat betrokken is bij de interactie (het roze gebied in het midden) en voorspelt de exacte bindingsplaatsen (paarse bollen). Krediet:Igor Kozlovskii en Petr Popov / Skoltech
Twee Skoltech-onderzoekers hebben een zeer efficiënt neuraal netwerkmodel gepresenteerd dat gegevens over de structuur van eiwitten gebruikt om te voorspellen welke van hun onderdelen interageren met andere biologische moleculen die peptiden worden genoemd. Dit wetende is nuttig voor het ontwikkelen van geneesmiddelen op basis van peptiden, die eiwit-eiwit interacties in cellen op een gerichte en niet-toxische manier kunnen beïnvloeden, reguleren van een breed scala aan cellulaire processen. De studie kwam uit in de Tijdschrift voor chemische informatie en modellering .
Eiwitten zijn de machinerie van cellen, rond bewegen, met elkaar bezig zijn, en het uitvoeren van allerlei operaties. Farmacologen zijn altijd geïntrigeerd geweest door het vooruitzicht om te sleutelen aan de interacties tussen eiwitten. Toch leken ze verboden terrein te zijn als een potentieel medicijndoelwit:de grotere therapeutische moleculen, genaamd biologische, konden de cel niet binnendringen om op eiwitten in te werken, terwijl agentia met kleine moleculen vaak niet in staat bleken tot een dergelijke actie.
peptiden, die van nature ongeveer 40% van de cellulaire processen bemiddelen of reguleren, een veelbelovende middenweg bezetten en vooruitzichten bieden voor medicijnen die gericht zijn op eiwit-eiwitinteracties. Peptiden bieden het beste van twee werelden:net als kleine moleculen, ze kunnen het celmembraan binnendringen om hun doelen daadwerkelijk te bereiken, en ze vertonen ook een lage toxiciteit, samen met een hoge affiniteit en specificiteit (sterke en gerichte actie) - de kenmerken van biologische geneesmiddelen.
Om op peptiden gebaseerde medicijnen te ontwerpen, farmacologen moeten de zogenaamde bindingsplaatsen voor een bepaald eiwitdoelwit kennen. Dat is, de vlekken op het eiwit die aan een peptide kunnen binden. Hoe meer van dergelijke sites bekend zijn, hoe meer mogelijkheden er zijn voor het ontwerpen van medicijnen.
Onderzoekers kunnen bindingsplaatsen experimenteel identificeren, bijvoorbeeld, met behulp van röntgenkristallografie, die de 3D-structuur van gekristalliseerde eiwitten onthult door te bestuderen hoe ze röntgenstralen afbuigen. Maar dit is erg duur om te doen voor een lange lijst van moleculen, en computationele methoden bieden een sneller en goedkoper alternatief. Sommigen van hen maken gebruik van machine learning-technieken, en naarmate er meer gegevens over de structuren van eiwit-peptidecomplexen worden verzameld, deze methoden worden krachtiger en leveren steeds betere voorspellingen van bindingsplaatsen op.
In hun krant van 22 juli in de Tijdschrift voor chemische informatie en modellering , Skoltech Ph.D. student Igor Kozlovskii en universitair docent Petr Popov van de iMolecule-groep presenteerden een rekenmethode genaamd BiteNetPp, die gebruikmaakt van de kracht van 3D convolutionele neurale netwerken om eiwit-peptide bindingsplaatsen te detecteren. In BiteNetPp, een bekende eiwitstructuur wordt toegevoerd aan een neuraal netwerk, die vervolgens verdachte peptidebindingsplaatsen benadrukt, en voert een set vermeende 3D-coördinaten uit, samen met de bijbehorende waarschijnlijkheidsscores.
Petr Popov geeft commentaar op de benadering van bindingsplaatsdetectie als beeldherkenning, oorspronkelijk geïntroduceerd in het eerdere artikel van het team en overgenomen in de studie die in dit verhaal wordt gerapporteerd:"Net zoals neurale netwerken kunnen worden getraind om te herkennen, zeggen, voetgangers of fietsers in gewone 2D-foto's, we beschouwen detectie van bindingsplaatsen als het spotten van een bepaald soort object in een afbeelding. Het verschil is dat we 3D-atoomstructuurgegevens gebruiken als onze invoer, dus het model werkt op 'voxels, "een driedimensionale analoog van pixels."
Het nieuw gepresenteerde model bouwt eigenlijk voort op dat in het vorige artikel. "Dit wordt domeinaanpassing genoemd. BiteNetPp is het eerste model dat is verfijnd op een eiwit-peptide-dataset nadat het aanvankelijk was getraind op eiwit-kleine-molecuulgegevens, " legt Popov uit. "Je kunt je dit voorstellen als het trainen van een model om plaatsen te identificeren waar fietsers de neiging hebben om op straat te stoppen, maar u begint met gegevens over waar voetgangers de neiging hebben om te stoppen - en pas daarna breidt u uw domein uit naar fietsers. In plaats van helemaal opnieuw te beginnen, je traint het model om, anticiperend dat de 'bindingsplaatsen' voor fietsers enige overeenkomsten zouden kunnen vertonen met diegene die voetgangers aantrekken:weet je, ijskraampjes, verkeerslichten, dat soort dingen."
De makers van het model hebben aangetoond dat BiteNetPp consistent beter presteert dan bestaande state-of-the-art methoden door hun voorspellingen te vergelijken voor die eiwit-peptide bindingsplaatsen die bekend zijn door experimentele observaties. belangrijk, het nieuwe model heeft minder dan een seconde nodig om een enkele eiwitstructuur te analyseren, waardoor het zeer geschikt is voor grootschalige studies. Er zijn duizenden eiwit-eiwit-interacties die mogelijk kunnen worden getarget door op peptiden gebaseerde geneesmiddelen, dus computationele methoden moeten snel genoeg zijn om hun screening haalbaar te maken in een farmacologische context.
 Hoe beginconcentraties te berekenen
Hoe beginconcentraties te berekenen Hoe het aantal isomeren te berekenen
Hoe het aantal isomeren te berekenen NIST vindt een manier om het printen van brandstofcellen met een snelle clip te meten
NIST vindt een manier om het printen van brandstofcellen met een snelle clip te meten Functionele films gemaakt van milieuvriendelijke kleimineralen en kleurstoffen
Functionele films gemaakt van milieuvriendelijke kleimineralen en kleurstoffen Onderzoeksteam ontwikkelt bioinks om therapieën in 3D te printen
Onderzoeksteam ontwikkelt bioinks om therapieën in 3D te printen
 Hoe Land Pollution
Hoe Land Pollution Nu moeten wijnliefhebbers iets weten over geologie - of toch niet?
Nu moeten wijnliefhebbers iets weten over geologie - of toch niet? Onder de oppervlakte kijken - steden leefbaarder maken
Onder de oppervlakte kijken - steden leefbaarder maken Na 1984:nauwe focus op natuurbrandtrends onderschat toekomstige risico's voor waterveiligheid
Na 1984:nauwe focus op natuurbrandtrends onderschat toekomstige risico's voor waterveiligheid Om de opwarming tot 2 graden Celsius te beperken, zijn emissiereducties in het hele voedselsysteem nodig
Om de opwarming tot 2 graden Celsius te beperken, zijn emissiereducties in het hele voedselsysteem nodig
Hoofdlijnen
- Stop en ga in het kaliumkanaal
- Statistische modellering helpt visserijbeheerders invasieve soorten te verwijderen
- 5 manieren waarop je hersenen je emoties beïnvloeden
- Wat is een kruisverwijzingssysteem?
- Genetische aandoeningen: definitie, oorzaken, lijst met zeldzame en veel voorkomende ziekten
- Sommige lemuren zijn eenlingen, anderen hunkeren naar verbinding
- Niet alle kroontjeskruid is gelijk voor eierleggende monarchen, studie onthult
- Mongoolse microfossielen wijzen op de opkomst van dieren op aarde
- Zijn er verschillen tussen mannelijke en vrouwelijke pesters?
- Binnenkort biologisch afbreekbare teenslippers

- Berekeningen voor microdruppels per minuut oefenen
- Onderzoekers ontwikkelen zelfaangedreven microfluïdische plaat die zich wikkelt, flappen en kruipen
- Milieuvriendelijk kooldioxide-conversieproces kan een revolutie teweegbrengen in de bestaande methode

- Een nieuw systeem om een verbinding te bouwen die cruciaal is voor de ontwikkeling van geneesmiddelen

 Gezamenlijk onderzoek toont aan dat fijner onbewerkt katoen het beste is voor de sanering van olievlekken
Gezamenlijk onderzoek toont aan dat fijner onbewerkt katoen het beste is voor de sanering van olievlekken Het voordeel van Moneyball verdwijnt zodra iedereen het doet
Het voordeel van Moneyball verdwijnt zodra iedereen het doet Drones betere voeten geven zodat ze kunnen rusten op nabijgelegen structuren
Drones betere voeten geven zodat ze kunnen rusten op nabijgelegen structuren De architectuur van het leven beschreven door computermodellering
De architectuur van het leven beschreven door computermodellering Fotokatalysator maakt waterstofproductie 10 keer efficiënter
Fotokatalysator maakt waterstofproductie 10 keer efficiënter Bidens hoopt over een decennium op onafhankelijkheid van zeldzame aardmetalen
Bidens hoopt over een decennium op onafhankelijkheid van zeldzame aardmetalen Synthese van een van de meest voorkomende organische lipiden verheldert de structuur ervan
Synthese van een van de meest voorkomende organische lipiden verheldert de structuur ervan Wanneer drones de nacht verlichten, gaan ze vuurwerk vervangen?
Wanneer drones de nacht verlichten, gaan ze vuurwerk vervangen?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Spanish | Portuguese | Swedish | German | Dutch | Danish | Italian | Norway |

-
Wetenschap © https://nl.scienceaq.com

