
Wetenschap
Neutronen onderzoeken biologische materialen voor inzicht in COVID-19-virusinfectie
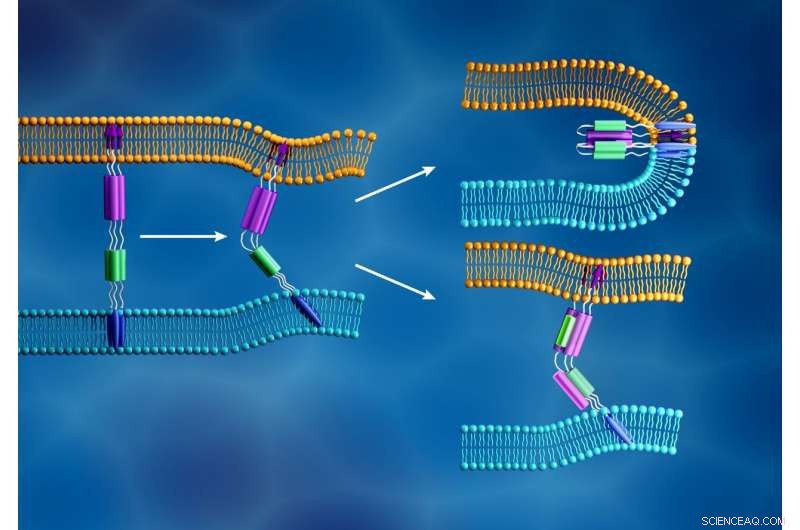
Het nieuwe coronavirusmembraan (lichtblauw) en het menselijke celmembraan (oranje) smelten samen wanneer het fusiepeptide van de virale S2-subeenheid (paarse pijlen) in het celmembraan wordt ingebracht en een andere component van de S2-subeenheid (paars en groen) vouwt om te vormen een strakke structuur, zoals weergegeven in de rechterbovenhoek. In tegenstelling tot, zoals weergegeven in de rechterbenedenhoek, fusieremmers zijn ontworpen om virale infectie te voorkomen door dit proces te verstoren. Krediet:ORNL/Jill Hemman
SARS-CoV-2, het coronavirus dat verantwoordelijk is voor de ziekte COVID-19, infecteert de wereld in hoog tempo. Als u begrijpt hoe deze infectie op moleculair niveau werkt, kunnen experts manieren vinden om de verspreiding te matigen of te stoppen.
Een team van wetenschappers van het Oak Ridge National Laboratory (ORNL) van het Department of Energy (DOE's) gebruikt neutronenreflectometrie om precies dat te doen.
Neutronen zijn in staat biologische materialen onder fysiologische omstandigheden te onderzoeken zonder ze te beschadigen. Door gebruik te maken van deze eigenschappen, de onderzoekers kunnen de infectiedynamiek van het virus namelijk meten.
Hun missie is om een gedetailleerd beeld te krijgen van enkele van de eerste stadia van infectie die optreden bij het celmembraan. Deze bevindingen zullen het team helpen antivirale kandidaat-geneesmiddelen te testen die dit proces zouden kunnen verstoren. De gegevens die uit deze experimenten zijn verkregen, zouden ook kunnen dienen als informatie voor andere onderzoeken die gericht zijn op de ontwikkeling van therapieën en vaccins.
De onderzoekers richten hun analyse op SARS-CoV-2 spike-eiwitten, weerhaakachtige structurele eiwitten die het oppervlak van het virus bedekken en het infectieproces in gang zetten. Het spike-eiwit bindt aan een receptor op de buitenste laag van de gastheercel en vergemakkelijkt de fusie tussen de virale en celmembranen, waardoor het virus de cel kan binnendringen en zijn genetisch materiaal kan vrijgeven. Het eiwitproducerende apparaat van de cel gebruikt deze genetische informatie vervolgens om nieuwe kopieën van het virus te maken.
Wanneer SARS-CoV-2 een gastheercel kaapt, zijn spike-eiwit splitst zich in twee subeenheden, genaamd S1 en S2. De twee delen zijn beide essentieel voor infectie. De S1-subeenheid bevat een receptorbindend domein dat een celreceptor herkent en eraan vasthoudt. Celreceptoren zijn eiwitten die zijn ingebed in het celmembraan en die kunnen binden aan specifieke moleculen buiten de cel. Door deze verbinding kunnen de componenten van vorm veranderen, wat op zijn beurt trapsgewijze veranderingen in de cel zou kunnen veroorzaken. Voor het SARS-CoV-2 spike-eiwit, deze verbinding activeert de S2-subeenheid, dat helpt het virus zijn membraan te laten samensmelten met dat van de cel. Daarom, de functie van het spike-eiwit is vergelijkbaar met het openen van een gesloten deur, met S1 als de sleutel die de deur ontgrendelt en S2 als de kracht die de deur openduwt.
Leren van eerdere epidemieën
De algemene structuur van het SARS-CoV-2 spike-eiwit lijkt sterk op die van SARS-CoV, een eerder coronavirus dat ernstig acuut respiratoir syndroom (SARS) veroorzaakte, en deze gelijkenis hielp het team bij het ontwikkelen van hun onderzoeksstrategie.
De S1-subeenheid is de focus van veel onderzoeken naar de ontwikkeling van geneesmiddelen, omdat is aangetoond dat dit deel van het spike-eiwit een immuunrespons in het menselijk lichaam oproept. Echter, eerdere SARS-CoV-onderzoeken hebben aangetoond dat de S1-subeenheid hoge mutatiesnelheden ervaart, waardoor het virus antilichaamgebaseerde behandelingen kan ontwijken terwijl het zijn vermogen om cellen te infecteren behoudt. "Dit is de les die we hebben geleerd van de oorspronkelijke SARS-epidemie, " zei Minh Phan, een postdoctoraal onderzoeksmedewerker bij ORNL en hoofdonderzoeker van dit project.
Phan en zijn collega's bestuderen de S2-subeenheid omdat dit onderdeel van het spike-eiwit niet zo snel muteert. Behandelingen die succesvol blijken te zijn in het remmen van de S2-functie kunnen langer werkzaam blijven.
Een kijk op het coronavirus op nanoschaal
Om de dynamiek van virale S2-subeenheden en gastheercelmembranen beter te begrijpen, de onderzoekers gebruiken de vloeistofreflectometer (LIQREF) bij ORNL's Spallation Neutron Source (SNS). Door te meten hoe neutronen onder verschillende hoeken reflecteren wanneer ze door verschillende soorten materie gaan, het instrument kan helpen licht te werpen op de structuur van biologische materialen op moleculaire schaal.
Het team synthetiseerde eerst een lipidemembraan dat het buitenmembraan nabootst van cellen die de oppervlakken in menselijke longen bekleden, waar deze virale infectie kan plaatsvinden. Ze identificeerden hoe de lipiden in het membraan waren georganiseerd en hoe deze opstelling verandert wanneer de membranen worden blootgesteld aan verschillende omstandigheden, zoals temperatuur, druk, en zuurgraad.
Bij het LIQREF-instrument, de onderzoekers verspreidden het lipidemembraan bovenop een dunne laag water in een apparaat dat een Langmuir-trog wordt genoemd. Vervolgens introduceren ze de S2-subeenheid in deze membranen om in detail te observeren hoe de S2- en lipidemembranen van vorm veranderen wanneer ze op elkaar inwerken.
Neutronen zijn ook ideaal voor dit onderzoek omdat ze gevoelig zijn voor het element waterstof, gemeenschappelijk voor alle biologische moleculen, en zijn isotopen. Door sommige waterstofatomen te vervangen door deuteriumatomen, wetenschappers kunnen contrast creëren in hun monsters en selectief inzoomen op verschillende structurele kenmerken. Deze techniek is handig voor het bestuderen van monsters met meerdere componenten met vergelijkbare dichtheden, zoals lipidemembranen.
"Over het algemeen, these membranes are not single-lipid membranes, " said John Ankner, an instrument scientist involved with this study. "They consist of lipids of a certain structure, lipids of another structure, cholesterol, eiwitten, and things that come in contact with them."
To capture this complexity, the research team is investigating multiple versions of the membrane, changing the contrast of the sample with deuterium each time.

Researchers at ORNL are using neutron scattering at the Spallation Neutron Source to better understand how spike proteins help the COVID-19 virus infect human cells and what drugs could be effective in stopping them. This research team includes John Ankner (left) and Minh Phan (right). Krediet:ORNL/Genevieve Martin
"By taking multiple measurements and assembling all of this information together, you can create a single picture of how these different components go together, " said Ankner.
The information derived from these experiments will then help steer the team's efforts in selecting and testing drug candidates that could block this interaction, such as fusion inhibitors that successfully blocked original SARS-CoV infections. If these inhibitors can stop the new coronavirus from invading healthy cells, existing drugs could potentially be repurposed to treat COVID-19 patients. The results may also help guide the design of new fusion inhibitors.
Capturing infection
While other studies have used protein crystallography to better understand the atomic structure of the coronavirus S2 subunit alone, this project is analyzing how S2 changes shape when interacting with a lipid membrane. A shape change could be important for inducing actions within a cell after the spike S1 subunit binds to the cell receptor. Phan also notes that the LIQREF instrument allows the team to measure these dynamics under physiological conditions, whereas protein crystallography only allows researchers to capture what the S2 subunit looks like in a crystallized form.
"At ORNL, we have the right tools to study the dynamics of the interaction under physiological conditions. This allows us to better understand how the S2 subunit moves and changes shape naturally in a wet environment, " said Phan. "Such information could complement what experts already know about the protein from crystallography. If we can help verify what this mechanism looks like, then we may have a clearer understanding to guide the development of drugs that block the fusion process.
Collaboration is key
Natuurlijk, learning more about the S2 subunit and its certain behaviors depends on the ability to grow quality samples, which involves synthesizing S2 subunit proteins, purifying them, and preparing them for experimentation.
Phan and Ankner note that this part of their research has been made possible only through collaboration with labs across ORNL and at outside institutions.
The S2 subunit protein was synthesized in mammalian cell cultures by Steve Foster, a biomedical researcher at the University of Tennessee Medical Center in Knoxville, Tennessee. Through this method, he can develop S2 proteins for research that retain several aspects of its natural structure and function.
"In our lab we routinely use mammalian cell cultures for protein production, so we hope we've produced an S2 protein best suited for this research analysis. Our proximity to ORNL also works well in that the sample doesn't have to travel far, meaning less risk of damaging the protein or distorting its original structure, which is critical for this work, " said Foster.
Following its synthesis, the sample was purified by Jessy Labbé and Michael Melesse Vergara from ORNL's Biosciences Division. Scientists from the ORNL Neutron Sciences Directorate then performed a series of tests to confirm the structure of the sample protein and check its purity. This effort was implemented by Yichong Fan and Wellington Leite from the Bio-Labs team, and Jacob Kinnun and Mary Odom from the SNS team.
"We put an enormous effort into making sure the protein has the right properties going into the experiment. If it does not, we could get spurious results and misinterpret what we're doing, " said Hugh O'Neill, director of ORNL's Center for Structural and Molecular Biology and lead researcher for the Bio-Labs team.
"This virus is extremely delicate in its components, and it's a big challenge to get these materials to the neutron instrument, " said Ankner. "That's why involving various ORNL labs and the University of Tennessee is so crucial. Each step that eventually gets the sample onto our instrument requires the expertise of lots of people."
This project also relied on efforts from the LIQREF instrument staff, who were instrumental in developing the systems, protocols, and modeling frameworks necessary to run the experiments and interpret the data.
"Experts across the division, across ORNL, and from partner institutes have come together for this project, " said Phan. "We couldn't have done this without their support, and it's greater motivation to fulfill our mission."
 Studie:Oceaanviskweek in tropen en subtropen die het meest worden getroffen door klimaatverandering
Studie:Oceaanviskweek in tropen en subtropen die het meest worden getroffen door klimaatverandering Naarmate de klimaatdoelstellingen van Parijs verdwijnen, geo-engineering doemt groter op
Naarmate de klimaatdoelstellingen van Parijs verdwijnen, geo-engineering doemt groter op Bangladesh plant miljoen bomen om bliksemtol te verminderen
Bangladesh plant miljoen bomen om bliksemtol te verminderen Big Muddy Missouri rivier heeft een plan nodig
Big Muddy Missouri rivier heeft een plan nodig Waarom het voorspellen van het weer en klimaat nog moeilijker is voor de regenachtige noorderburen van Australië
Waarom het voorspellen van het weer en klimaat nog moeilijker is voor de regenachtige noorderburen van Australië
Hoofdlijnen
- Hoe het Alien Hand Syndroom werkt
- Voedselketen: definitie, soorten, belang en voorbeelden (met diagram)
- Staan we aan het begin van het einde van de westerse beschaving?
- Verloren Australische duiker zwom mijlen naar kust gestalkt door haai
- IJsberen verdringen op Russisch eiland in teken van Arctische verandering
- Nieuwe studie biedt inzicht in een hondenleven in gezinnen met kinderen
- De voordelen en nadelen van het Bacillus-expressiesysteem
- Wat is een Mordant in de microbiologie?
- Wat betekent ##### in Excel?
- 5 manieren om te weten of er een chemische verandering heeft plaatsgevonden

- Een nieuw type chemische binding:de ladingsverschuivingsbinding

- Nieuw ontdekte hybride moleculen zouden kunnen dienen als een nieuwe categorie antikankermiddelen

- Nieuwe geleiders met een hoog protonengehalte met inherent zuurstofarme lagen openen een duurzame toekomst
- Microgolfplastic verlengt de levensduur van lithium-zwavelbatterijen

 Biomedische doorbraak:koolstofnanodeeltjes die je thuis kunt maken
Biomedische doorbraak:koolstofnanodeeltjes die je thuis kunt maken Locals maken zich zorgen dat wind en zonne-energie bossen en boerderijen zullen opslokken
Locals maken zich zorgen dat wind en zonne-energie bossen en boerderijen zullen opslokken NASA-NOAA-satelliet staart in het oog van Super Typhoon Mangkhut
NASA-NOAA-satelliet staart in het oog van Super Typhoon Mangkhut Hoe wordt water gevormd tijdens cellulaire ademhaling?
Hoe wordt water gevormd tijdens cellulaire ademhaling?  Partijdiscipline voor springgenen
Partijdiscipline voor springgenen Kometen droegen bij aan de atmosfeer van de aarde, zegt studie van 3 miljard jaar oude mineralen
Kometen droegen bij aan de atmosfeer van de aarde, zegt studie van 3 miljard jaar oude mineralen Supercomputers om de productie van keramische matrixcomposiet te stimuleren
Supercomputers om de productie van keramische matrixcomposiet te stimuleren Discovery haalt de druk van bloedmetingen
Discovery haalt de druk van bloedmetingen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway | French |

-
Wetenschap © https://nl.scienceaq.com

