
Wetenschap
Nieuwe RNA-gebaseerde tool kan hersencircuits verlichten, specifieke cellen bewerken
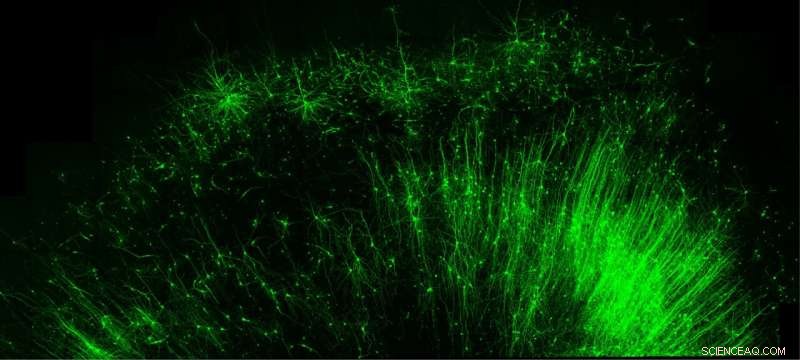
Het labelen en verlichten van alleen de remmende "rem"-cellen (groen) in menselijk hersenweefsel is slechts een van de vele dingen die de nieuwe tool van Duke University, CellREADR, kan doen. Krediet:Derek Southwell, Duke University
Onderzoekers van de Duke University hebben een op RNA gebaseerde bewerkingstool ontwikkeld die zich richt op individuele cellen in plaats van genen. Het is in staat om elk type cel nauwkeurig te targeten en selectief elk eiwit van belang toe te voegen.
Onderzoekers zeiden dat de tool het mogelijk zou kunnen maken om zeer specifieke cellen en celfuncties aan te passen om ziekten te beheersen.
Met behulp van een op RNA gebaseerde sonde, heeft een team onder leiding van neurobioloog Z. Josh Huang, Ph.D. en postdoctoraal onderzoeker Yongjun Qian, Ph.D. aangetoond dat ze fluorescerende tags in cellen kunnen introduceren om specifieke soorten hersenweefsel te labelen; een lichtgevoelige aan/uit-schakelaar om neuronen naar keuze uit te schakelen of te activeren; en zelfs een zelfvernietigend enzym om bepaalde cellen precies uit te wissen, maar andere niet. Het werk verschijnt 5 oktober in Nature .
Hun selectieve celbewakings- en controlesysteem is gebaseerd op het ADAR-enzym, dat in de cellen van elk dier wordt aangetroffen. Hoewel dit de eerste dagen zijn voor CellREADR (Cell Access through RNA sensing by Endogene ADAR), lijken de mogelijke toepassingen eindeloos, zei Huang, evenals het potentieel om in het hele dierenrijk te werken.
"We zijn enthousiast omdat dit een vereenvoudigde, schaalbare en generaliseerbare technologie biedt om alle celtypen in elk dier te volgen en te manipuleren," zei Huang. "We zouden specifieke soorten celfuncties kunnen aanpassen om ziekten te beheersen, ongeacht hun initiële genetische aanleg," zei hij. "Dat is niet mogelijk met de huidige therapieën of medicijnen."
CellREADR is een aanpasbare reeks RNA die bestaat uit drie hoofdsecties:een sensor, een stopbord en een set blauwdrukken.
Eerst beslist het onderzoeksteam welk specifiek celtype ze willen onderzoeken en identificeert het een doel-RNA dat op unieke wijze door dat celtype wordt geproduceerd. De opmerkelijke weefselspecificiteit van de tool is gebaseerd op het feit dat elk celtype kenmerkend RNA produceert dat niet wordt gezien in andere celtypen.
Een sensorsequentie wordt vervolgens ontworpen als de complementaire streng van het doel-RNA. Net zoals de sporten op DNA zijn samengesteld uit complementaire moleculen die inherent tot elkaar worden aangetrokken, heeft RNA hetzelfde magnetische potentieel om te koppelen met een ander stuk RNA als het bijpassende moleculen heeft.
Nadat een sensor een cel is binnengedrongen en zijn doel-RNA-sequentie heeft gevonden, klonteren beide stukken samen om een stuk dubbelstrengs RNA te creëren. Deze nieuwe RNA-mashup activeert het enzym ADAR om de nieuwe creatie te inspecteren en vervolgens een enkele nucleotide van zijn code te wijzigen.
The ADAR enzyme is a cell-defense mechanism designed to edit double-strand RNA when it occurs, and is believed to be found in all animal cells.
Knowing this, Qian designed CellREADR's stop sign using the same specific nucleotide ADAR edits in double-stranded RNA. The stop sign, which prevents the protein blueprints from being built, is only removed once CellREADR's sensor docks to its target RNA sequence, making it highly specific for a given cell type.
Once ADAR removes the stop sign, the blueprints can be read by cellular machinery that builds the new protein inside the target cell.
In their paper, Huang and his team put CellREADR through its paces. "I remember two years ago when Yongjun built the first iteration of CellREADR and tested it in a mouse brain," Huang said. "To my amazement, it worked spectacularly on his first try."
The team's careful planning and design paid off as they were then able to demonstrate CellREADR accurately labelled specific brain cell populations in living mice, as well as effectively added activity monitors and control switches where directed. It also worked well in rats, and in human brain tissue collected from epilepsy surgeries.
"With CellREADR, we can pick and choose populations to study and really begin to investigate the full range of cell types present in the human brain," said co-author Derek Southwell, M.D., Ph.D., a neurosurgeon and assistant professor in the department of neurosurgery at Duke.
Southwell hopes CellREADR will improve his and others' understanding of the wiring diagram for human brain circuits and the cells within them, and in doing so, help advance new therapies for neurological disorders, such as a promising new method to treat drug-resistant epilepsy he is piloting.
Huang and Qian are especially hopeful about CellREADR's potential as a "programmable RNA medicine" to possibly cure diseases—since that's what drew them both to science in the first place. They have applied for a patent on the technology.
"When I majored in pharmacology as an undergraduate, I was very naïve," Qian said. "I thought you could do a lot of things, like cure cancer, but actually it's very difficult. However, now I think, yes maybe we can do it." + Verder verkennen
New technology identifies molecular properties of cells and maps their location within tissues
 Tropische depressie 15E lijkt bijna vormloos op NASA-satellietbeelden
Tropische depressie 15E lijkt bijna vormloos op NASA-satellietbeelden Bossen herstellen met 1 boom tegelijk, om het klimaat te helpen herstellen
Bossen herstellen met 1 boom tegelijk, om het klimaat te helpen herstellen Vulkaan in het zuiden van Japan barst uit; geen verwondingen of schade
Vulkaan in het zuiden van Japan barst uit; geen verwondingen of schade Verbrande bomen en miljarden in contanten:hoe een klimaatprogramma in Californië ervoor zorgt dat bedrijven blijven vervuilen
Verbrande bomen en miljarden in contanten:hoe een klimaatprogramma in Californië ervoor zorgt dat bedrijven blijven vervuilen Licht onthult de oorsprong van een zandkorrel
Licht onthult de oorsprong van een zandkorrel
Hoofdlijnen
- De in 1995 opgegraven Plesiosaurus bleek een langnekkig zeereptiel te zijn
- 245 miljoen jaar oude fossielen bieden nieuwe inzichten in de evolutie en voedingsstrategieën van waterinsecten
- Daar is een studie over:het stenen tijdperk had net zoveel linkse mensen
- Studie onderzoekt langere levensduur door gebrekkige RNA-verwerking
- Nieuwe oude dolfijnsoort Urkudelphis chawpipacha ontdekt in Ecuador
- Maak een lijst van de stappen van de celcyclus in volgorde
- Een 3D-model voor celbiologische projecten bouwen Mitochondria en chloroplast
- Te veel charisma kan de effectiviteit van een leider schaden
- Het vinden van een dodelijke parasieten kwetsbaarheden
- Vrijgelaten zeeschildpad draagt as van oceanografen naar zee

- 245 miljoen jaar oude fossielen bieden nieuwe inzichten in de evolutie en voedingsstrategieën van waterinsecten

- Is Algae een Decomposer, Scavenger of Producer?

- Sluwe kraaien weten wat er nodig is om een goed stuk gereedschap te maken

- Gedachtenexperiment:wat is ons transhumane pad voorbij de aarde?

 Victoria verbiedt fracking voorgoed maar heft stilletjes het verbod op gasexploratie op land op
Victoria verbiedt fracking voorgoed maar heft stilletjes het verbod op gasexploratie op land op Vikingen arriveerden in Ierland toen de bevolking ernstig achteruitging
Vikingen arriveerden in Ierland toen de bevolking ernstig achteruitging NASA ziet post-tropische cycloon Helene Ierland treffen, Verenigd Koninkrijk
NASA ziet post-tropische cycloon Helene Ierland treffen, Verenigd Koninkrijk Afbeelding:onderdeel van de Europese servicemodule van het ruimtevaartuig Orion
Afbeelding:onderdeel van de Europese servicemodule van het ruimtevaartuig Orion Biologische drankenkartons
Biologische drankenkartons Diamanten zijn voor altijd:nieuwe basis voor nanostructuren
Diamanten zijn voor altijd:nieuwe basis voor nanostructuren Modelontwikkeling is cruciaal om klimaatverandering te begrijpen
Modelontwikkeling is cruciaal om klimaatverandering te begrijpen Een hybride materiaal om organische verontreinigingen in de atmosfeer op te sporen
Een hybride materiaal om organische verontreinigingen in de atmosfeer op te sporen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

