
Wetenschap
Wetenschappers stellen efficiëntieregels voor om het gebruik van nieuwe technologie voor het bewerken van genen te verbeteren
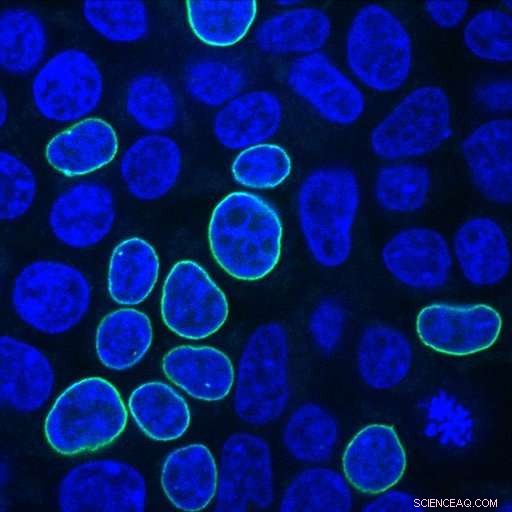
Menselijke embryonale niercellen gloeien groen na reparatie van een door CRISPR geïnduceerde DNA-breuk met een PCR-fragment dat codeert voor een fluorescerend eiwit en homologiearmen met 33 nucleotiden. Krediet:Alexandre Paix
Johns Hopkins-wetenschappers hebben een gestroomlijnde methode en bijbehorende efficiëntie-"regels" ontwikkeld voor het introduceren van nieuwe DNA-sequenties in cellen na gebruik van de gen-cutting tool die bekend staat als CRISPR. De wetenschappers zeggen dat de methode, die ze baseerden op tests met muizenembryo's en duizenden menselijke cellen, zou de consistentie en efficiëntie van genoombewerking kunnen verbeteren.
De nieuwe methode en de ontwikkeling ervan worden online beschreven in de 28 november in de Proceedings van de National Academy of Sciences .
"CRISPR is een hulpmiddel om wetenschappers te helpen het genoom te wijzigen, de uitkomst van bepaalde eigenschappen voorspellen en bestuderen, maar de tool zelf creëert alleen breuken in het genoom. Het bepaalt niet hoe een nieuwe DNA-sequentie in het genoom wordt ingebracht, " zegt Geraldine Seydoux, doctoraat, de Huntington Sheldon Professor in Medical Discovery in de afdeling Molecular Biology and Genetics en vice-decaan voor fundamenteel onderzoek aan de Johns Hopkins University School of Medicine, en een onderzoeker bij het Howard Hughes Medical Institute.
"We gingen onderzoeken hoe cellen door CRISPR veroorzaakte breuken herstellen met als doel het natuurlijke DNA-herstelproces van de cel te gebruiken om nieuwe sequenties in het genoom te introduceren. We waren verrast toen we ontdekten dat cellen gemakkelijk sequenties van vreemd DNA kopiëren om DNA-breuken te repareren , zolang de vreemde DNA's lineair zijn, Seydoux vult aan. "Door te bestuderen hoe vreemde DNA-fragmenten worden gekopieerd tijdens het reparatieproces, we bedachten enkele eenvoudige regels om genoombewerking zo efficiënt mogelijk te maken, het gereedschap optimaliseren, en doe dat met vertrouwen."
CRISPR, wat staat voor geclusterde regelmatige interspaced korte palindroomherhaling, is de afgelopen vijf jaar populair geworden onder wetenschappers als een hulpmiddel om DNA efficiënt te knippen. Het werd aangepast voor gebruik in zoogdiercellen van een natuurlijk viraal afweerproces in bacteriële cellen waarbij dodelijke sneden in viraal DNA worden gemaakt. Eigenlijk, het gereedschap is een gestroomlijnde set moleculaire "schaar".
Het heersende geloof, onder wetenschappers, is dat cellen DNA-breuken repareren door een willekeurige set nucleotiden in te voegen, de chemische bouwstenen van DNA. Dit vernietigt meestal elk gen dat zich bevindt op de plek waar het DNA is gebroken.
Het is ook bekend bij wetenschappers dat, zo nu en dan, cellen gebruiken een andere bron - een sequentie van een ander stuk DNA, of "donor"-DNA - om de breuk in het DNA te verzegelen. Echter, de nieuwe "donor"-sequentie kan niet vanzelf in een lege ruimte in het genoom worden ingevoegd.
In plaats daarvan, het nieuwe donor-DNA heeft aan elk uiteinde een soort tape nodig om het in de opening te houden die door de snede is gemaakt. Wetenschappers noemen deze tape de 'homologie'-armen van het donor-DNA.
De homologie-armen bestaan uit nucleotiden die de intacte delen van het DNA overlappen met overeenkomende genetische code. Dit helpt het donor-DNA aan het intacte DNA te "kleven".
Nog, wetenschappers beschouwden het gebruik van donor-DNA als een inefficiënte manier om het genoom te herstellen, ervan uitgaande dat er lange homologiearmen voor nodig waren, vooral bij het invoegen van een lange DNA-sequentie, en enkelstrengs of circulair DNA, die moeilijk te bereiden zijn in lange maten.
Naarmate wetenschappers meer ervaring opdeden met CRISPR, Seydoux zegt, "Er ontstonden vragen over de optimale ontwerpregels voor donor-DNA en de lengte van de homologiearmen."
Op zoek naar antwoorden op deze vragen, de Johns Hopkins-wetenschappers plaatsten verschillende combinaties van donor-DNA in menselijke embryonale niercellen, bekend om hun vermogen om goed te groeien en om hun frequente gebruik in kankeronderzoek. De wetenschappers gebruikten donor-DNA met een gen dat codeert voor een fluorescerend eiwit, die groen oplicht in het kernmembraan van de cel wanneer de geninsertie succesvol is.
Johns Hopkins-onderzoeksmedewerker Alexandre Paix ontdekte dat lineaire DNA-fragmenten heel goed functioneren als donor, en zijn twee tot vijf keer efficiënter dan circulaire DNA's (bekend als plasmiden) in menselijke cellen. "Lineair DNA is heel gemakkelijk te bereiden in het laboratorium, met behulp van PCR, " zegt Paix, verwijzend naar polymerasekettingreactiehulpmiddelen, die worden gebruikt om DNA te amplificeren.
Paix testte ook verschillende lengtes van homologiearmen. Hij ontdekte dat de goede plek voor homologie-armen ongeveer 35 nucleotiden lang is, veel korter dan wetenschappers doorgaans gebruiken.
specifiek, bleek dat homologiearmen van 33 tot 38 nucleotiden lang net zo succesvol waren als die met 518 nucleotiden, wat tussen de 10 en 20 procent succesvolle bewerkingen oplevert onder optimale omstandigheden. In tegenstelling tot, toen de wetenschappers homologiearmen van 15 en 16 nucleotiden lang testten, de slagingspercentages daalden met de helft. Ze herhaalden deze resultaten op drie verschillende locaties in het menselijk genoom.
Ze ontdekten ook dat de nieuw ingevoegde sequentie, de homologie-armen niet meegerekend, kan maximaal 1 zijn 000 nucleotiden lang.
Het team behaalde succespercentages tussen 10 en 50 procent met inserts variërend van 57 tot 993 nucleotiden lang. Kortere sequenties werden met meer succes ingevoegd dan langere. Bijvoorbeeld, nieuwe reeksen die 57 waren, 714 en 993 nucleotiden lang werden met succes ingevoegd 45,4, 23,5 en 17,9 procent van de tijd, respectievelijk. voorbij 1, 000 nucleotiden, nieuwe inzetstukken met 1, 122 en 2, 229 nucleotiden hadden weinig succes - ongeveer 0,5 procent van de tijd. "Op die maat het wordt erg moeilijk om de hoeveelheid donor-DNA in te voeren die nodig is voor bewerking. Cellen hebben de neiging om te 'stikken' in zoveel DNA, ', zegt Seydoux.
Eindelijk, het team ontdekte ook dat het succespercentage van het bewerken piekt wanneer de nieuwe sequentie zich binnen 30 nucleotiden van de CRISPR-cut-site bevindt. "Meer dan 30 nucleotiden, het inbrengen is niet werkbaar, ', zegt Seydoux.
"Deze parameters zouden geschikt moeten zijn voor de meeste genen die wetenschappers proberen te bewerken. In feite, de meeste experimenten omvatten het bewerken van slechts twee tot drie nucleotiden dicht bij de CRISPR-cut-site, ", voegt Seydoux toe.
Het onderzoeksteam testte ook of dezelfde aanpak zou kunnen werken in muizenembryo's. Met behulp van een PCR-fragment met 36-nucleotide homologie-armen, het team heeft met succes een 739 nucleotide-lange sequentie ingevoegd die codeert voor een fluorescerend eiwit in 27 van de 87 (31 procent) muizenembryo's.
Seydoux' onderzoeksteam gebruikt de reparatieregels al om DNA te bestuderen in Caenorhabditis elegans, een soort worm, en de onderzoekers onderzoeken of de reparatieregels ook gelden voor andere typen menselijke cellen.
Voordat de richtlijnen algemeen worden aangenomen, Seydoux zegt dat ze moeten worden getest in meer menselijke celtypen en andere organismen.
 Gluten in tarwe:wat is er veranderd in 120 jaar fokken?
Gluten in tarwe:wat is er veranderd in 120 jaar fokken? Wat gebeurt er wanneer een ionische verbinding oplost in water?
Wat gebeurt er wanneer een ionische verbinding oplost in water?  Van het maken van wijn tot het beheren van mijnafval, klei is belangrijk voor veel industrieën
Van het maken van wijn tot het beheren van mijnafval, klei is belangrijk voor veel industrieën Onthulling van de nare werking van transvetzuren in het bloed
Onthulling van de nare werking van transvetzuren in het bloed Waarom zijn elektronenmicroscopen belangrijk?
Waarom zijn elektronenmicroscopen belangrijk?
 Nieuwe studie helpt de nauwkeurigheid van toekomstige voorspellingen van klimaatverandering te verbeteren
Nieuwe studie helpt de nauwkeurigheid van toekomstige voorspellingen van klimaatverandering te verbeteren Abonnementen op satellietwaarschuwingen gekoppeld aan verminderde ontbossing in Afrika
Abonnementen op satellietwaarschuwingen gekoppeld aan verminderde ontbossing in Afrika Parijs verbiedt oude diesels om vervuiling aan te pakken
Parijs verbiedt oude diesels om vervuiling aan te pakken Zweden is geweldig in het omzetten van afval in energie
Zweden is geweldig in het omzetten van afval in energie  De Indonesische Merapi-vulkaan spuwt hete wolken, 500 evacueren
De Indonesische Merapi-vulkaan spuwt hete wolken, 500 evacueren
Hoofdlijnen
- De oudste kleur van de aarde was roze
- Een titerrapport lezen
- Genezing van verbrande poten met vissenhuid
- Is maretak meer dan alleen een excuus voor een kus?
- Hevig debat komt tot leven over annulering jacht op grizzlyberen
- Ja,
- Drie manieren waarop genetische diversiteit optreedt tijdens Meiosis
- Wat wordt er weergegeven als geen enkele kopie van een allel de expressie maskeert?
- Een omkeerbare hoofdschakelaar ontdekken voor ontwikkeling
- Droogteresistente plantengenen kunnen de evolutie van waterbesparende gewassen versnellen

- Amerikaanse biologische klok-genetici winnen Nobelprijs voor Geneeskunde 2017

- De biodiversiteit van de aarde verandert naarmate de planeet opwarmt. Maar hoe?
- Celsignalen die wondgenezing in gang zetten zijn verrassend complex
- Nieuwe sorghumcultivars kunnen duizenden liters ethanol produceren

 Science Fair Ideas With a Baseball
Science Fair Ideas With a Baseball  Hoe de kegelslak dodelijk gif ons kan helpen betere medicijnen te maken
Hoe de kegelslak dodelijk gif ons kan helpen betere medicijnen te maken Beruchte astrologische artsen 400 jaar oude aantekeningen getranscribeerd en online vrijgegeven
Beruchte astrologische artsen 400 jaar oude aantekeningen getranscribeerd en online vrijgegeven Het geheim van katalysatoren die de efficiëntie van brandstofcellen verhogen
Het geheim van katalysatoren die de efficiëntie van brandstofcellen verhogen Ecologische zorgen die de toendra beïnvloeden
Ecologische zorgen die de toendra beïnvloeden  Afbeelding:Hubble bekijkt melkweg uit beroemde catalogus
Afbeelding:Hubble bekijkt melkweg uit beroemde catalogus Een stad ontwerpen zonder auto's - in het belang van de kinderen
Een stad ontwerpen zonder auto's - in het belang van de kinderen Deze beeldsensor werkt op zonlicht
Deze beeldsensor werkt op zonlicht
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Spanish | Portuguese | Swedish | German | Dutch | Danish | Italian | Norway |

-
Wetenschap © https://nl.scienceaq.com

