
Wetenschap
Ontwikkeling van een zeer nauwkeurig computermodel van het menselijk metabolisme
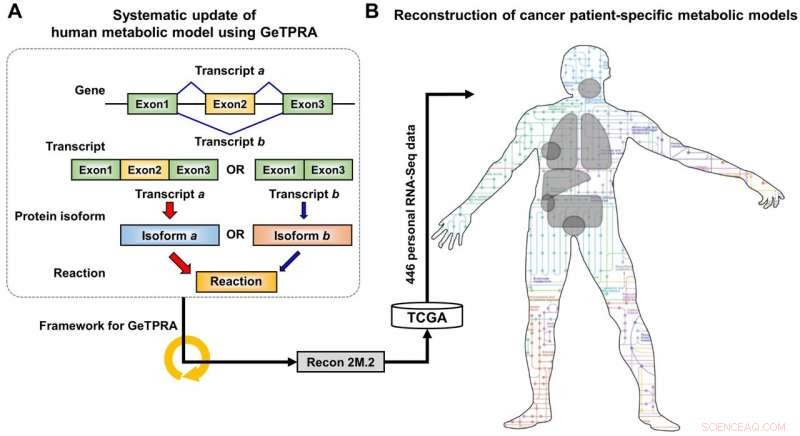
Schema van de ontwikkeling van Recon 2M.2 en het gebruik ervan bij het reconstrueren van persoonlijke metabole modellen op genoomschaal (GEM's). (A) Bij de ontwikkeling van Recon 2M.2 een concept van alternatieve splicing van menselijke genen werd overwogen, en opgenomen in modellering via het computationele raamwerk voor Gene-Transcript-Protein-Reaction Associations (GeTPRA). (B) Bij de ontwikkeling van Recon 2M.2, kankerpatiëntspecifieke GEM's kunnen worden gereconstrueerd met behulp van persoonlijke biologische gegevens (RNA-Seq-gegevens). In dit onderzoek, patiëntspecifieke RNA-Seq-gegevens werden verkregen uit The Cancer Genome Atlas (TCGA; https://cancergenome.nih.gov/). Krediet:KAIST
Een Koreaans onderzoeksteam van KAIST heeft een computationeel raamwerk ontwikkeld dat de reconstructie van een uitgebreid computermodel van het menselijk metabolisme mogelijk maakt, die een nauwkeurige voorspelling van persoonlijke metabolische kenmerken (of fenotypes) mogelijk maakt.
Door persoonlijke metabole fenotypes te begrijpen, kunnen we effectieve therapeutische strategieën ontwerpen voor verschillende chronische en infectieziekten. Een menselijk computermodel genaamd het genoomschaal metabolisch model (GEM) bevat informatie over duizenden metabole genen en hun overeenkomstige reacties en metabolieten, en heeft een belangrijke rol gespeeld bij het voorspellen van metabole fenotypes. Hoewel er verschillende versies van menselijke GEM's zijn uitgebracht, ze hadden ruimte voor verdere ontwikkeling, vooral wat betreft het opnemen van biologische informatie afkomstig van een menselijk genetisch mechanisme dat 'alternatieve splicing' wordt genoemd. Alternatieve splicing is een genetisch mechanisme waardoor een gen meerdere reacties kan veroorzaken, en wordt sterk geassocieerd met pathologie.
Om dit probleem aan te pakken, Jae Yong Ryu (een doctoraatsstudent), Dr. Hyun Uk Kim (onderzoeker), en Distinguished Professor Sang Yup Lee, allemaal van de afdeling Chemische en Biomoleculaire Engineering van KAIST, een computationeel raamwerk ontwikkeld dat systematisch metabolische reacties genereert, en voegt ze toe aan de menselijke GEM. De resulterende menselijke GEM bleek nauwkeurig metabole fenotypes te voorspellen onder verschillende omgevingsomstandigheden. De onderzoeksresultaten zijn online gepubliceerd in Proceedings van de National Academy of Sciences ( PNAS ) op 24 oktober, 2017, onder de titel "Framework and resource for more than 11, 000 gen-transcript-eiwit-reactie-associaties in het metabolisme van de mens."
Het onderzoeksteam heeft eerst de biologische inhoud van een eerdere versie van de menselijke GEM bijgewerkt. De bijgewerkte biologische inhoud omvat metabole genen en hun overeenkomstige metabolieten en reacties. Vooral, metabolische reacties gekatalyseerd door reeds bekende eiwit-isovormen werden bovendien opgenomen in de menselijke GEM; eiwit-isovormen zijn meerdere varianten van eiwitten die zijn gegenereerd uit individuele genen via het alternatieve splitsingsproces. Elke eiwit-isovorm is vaak verantwoordelijk voor de werking van een metabolische reactie. Hoewel meerdere eiwit-isovormen die zijn gegenereerd uit één gen verschillende functies kunnen spelen door verschillende sets eiwitdomeinen en/of subcellulaire lokalisaties te hebben, dergelijke informatie werd niet goed overwogen in eerdere versies van menselijke GEM's.
Bij de eerste update van de menselijke GEM, genaamd Recon 2M.1, het onderzoeksteam implementeerde vervolgens een computationeel raamwerk dat systematisch informatie genereert over Gene-Transcript-Protein-Reaction Associations (GeTPRA) om eiwit-isovormen te identificeren die voorheen niet werden geïdentificeerd. Dit raamwerk is in dit onderzoek ontwikkeld. Als gevolg van de implementatie van het raamwerk voor GeTPRA, meer dan 11, 000 GeTPRA werden automatisch voorspeld, en grondig gevalideerd. Aanvullende metabolische reacties werden vervolgens toegevoegd aan Recon 2M.1 op basis van de voorspelde GeTPRA voor de eerder niet-gekarakteriseerde eiwit-isovormen; Recon 2M.1 werd omgedoopt tot Recon 2M.2 van deze upgrade. Eindelijk, Recon 2M.2 werd geïntegreerd met 446 sets persoonlijke biologische gegevens (RNA-Seq-gegevens) om patiëntspecifieke kankermodellen te bouwen. Deze patiëntspecifieke kankermodellen werden gebruikt om de activiteiten van het kankermetabolisme en antikankerdoelen te voorspellen.
De ontwikkeling van een nieuwe versie van menselijke GEM's, samen met het computationele raamwerk voor GeTPRA, zal naar verwachting studies in fundamentele menselijke genetica en geneeskunde stimuleren. Modelbestanden van de menselijke GEM's Recon 2M.1 en 2M.2, een volledige lijst van de GeTPRA en de broncode voor het rekenraamwerk om de GeTPRA te voorspellen, zijn allemaal beschikbaar als onderdeel van de publicatie van deze studie.
Distinguished Professor Lee zei:"De voorspelde GeTPRA uit het computationele raamwerk zal naar verwachting dienen als richtlijn voor toekomstige experimenten met menselijke genetica en biochemie, terwijl de resulterende Recon 2M.2 kan worden gebruikt om medicijndoelen voor verschillende menselijke ziekten te voorspellen."
 Wetenschappers leggen moleculaire kaarten van dierlijk weefsel vast met ongekende details
Wetenschappers leggen moleculaire kaarten van dierlijk weefsel vast met ongekende details Starend in de vlammen van ionische winden
Starend in de vlammen van ionische winden Laserbehandeling toont potentieel voor vermindering van industriële chemische verwerking van voertuigen
Laserbehandeling toont potentieel voor vermindering van industriële chemische verwerking van voertuigen Wat als vliegtuigen hun eigen schade konden herstellen?
Wat als vliegtuigen hun eigen schade konden herstellen? Aanpasbaar synthetisch antibioticum verslaat resistente bacteriën
Aanpasbaar synthetisch antibioticum verslaat resistente bacteriën
 Covid-Lockdown, seizoensveranderingen beïnvloeden de uitstoot van Californië
Covid-Lockdown, seizoensveranderingen beïnvloeden de uitstoot van Californië Bij benaderingen voor bosbehoud moeten de rechten van de lokale bevolking worden erkend
Bij benaderingen voor bosbehoud moeten de rechten van de lokale bevolking worden erkend Patronen blootleggen in brandende bosbranden in Californië
Patronen blootleggen in brandende bosbranden in Californië White Island:een kijkje in de meest actieve vulkaan van Nieuw-Zeeland
White Island:een kijkje in de meest actieve vulkaan van Nieuw-Zeeland Doelstellingen van de viskwekerij
Doelstellingen van de viskwekerij
Hoofdlijnen
- Triest afscheid als de in Maleisië geboren panda naar China gaat
- Welke materialen kan ik gebruiken om een DNA-model te maken?
- Hoe een 3D-model voor celbiologieprojecten te bouwen Mitochondria & Chloroplast
- Onderzoekers ontwikkelen krachtige nieuwe methode voor microbioomanalyse
- Wat zijn energiegerelateerde organellen?
- Wat is troebelheid en wat staat er in de microbiologie?
- Ryk heeft een begeleider nodig
- Welke organismen voeren fotosynthese uit?
- Wilt u 41 procent van de zeer bedreigde gewervelde planeten redden? Werken op eilanden
- Otzi the Iceman at een vetrijke laatste maaltijd
- Theorie van de evolutie van geslachten getest met algen
- Herten geven de voorkeur aan inheemse planten die blijvende schade aan bossen achterlaten

- Trofeejacht kan uitsterven veroorzaken in een veranderende omgeving

- Caribische bidsprinkhanen hebben een oude Afrikaanse oorsprong

 De Amazone staat in brand:hier zijn 5 dingen die je moet weten
De Amazone staat in brand:hier zijn 5 dingen die je moet weten Inheemse talen moeten meer aan bod komen in wetenschapscommunicatie
Inheemse talen moeten meer aan bod komen in wetenschapscommunicatie Onderzoekers willen de darmgezondheid van vee verbeteren
Onderzoekers willen de darmgezondheid van vee verbeteren Onderzoekers ontdekken nieuwe klasse van chemische reacties
Onderzoekers ontdekken nieuwe klasse van chemische reacties Onderzoekers ontwerpen moleculen die enzymen remmen bij infectieziekten
Onderzoekers ontwerpen moleculen die enzymen remmen bij infectieziekten APS-upgrade vereist de eerste oefenrun bouwmodules voor de nieuwe opslagring
APS-upgrade vereist de eerste oefenrun bouwmodules voor de nieuwe opslagring Hoe lang duurt het voordat de zonnewind de aarde bereikt?
Hoe lang duurt het voordat de zonnewind de aarde bereikt?  Hoe maak je een model van Jupiter voor de derde rang
Hoe maak je een model van Jupiter voor de derde rang
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

