
Wetenschap
Top-down design brengt nieuwe DNA-structuren tot leven
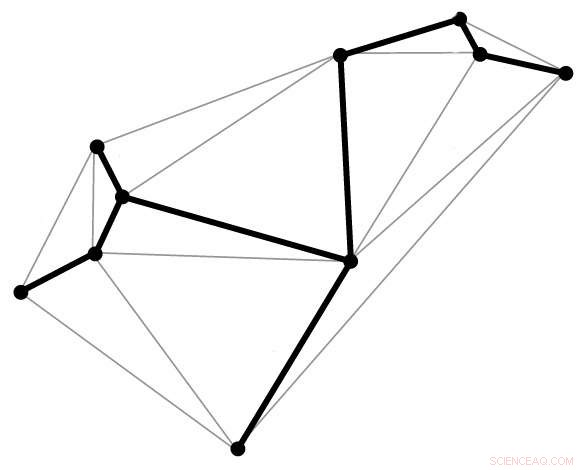
De dikgedrukte lijn, bekend als een opspannende boom, volgt de gewenste geometrische vorm, elk hoekpunt slechts één keer aanraken. In de nieuwe DNA-origamimethode wordt een spanning tree-algoritme gebruikt om het juiste routeringspad voor de DNA-streng in kaart te brengen. Krediet:publiek domein
Een van de waardevolle bezittingen in de Wellcome Library in Londen is een ruwe potloodschets gemaakt in 1953 door Francis Crick. De tekening is een van de eerste die de dubbele helixstructuur van DNA laat zien - de blauwdruk van de natuur voor het ontwerp van zeeslakken, mensen, en elke andere levende vorm op aarde.
Weinigen hadden echter kunnen voorspellen, dat DNA's eenvoudige eigenschappen van zelfassemblage, en zijn veelzijdige informatiedragende capaciteit, voor veel doeleinden kan worden gebruikt, nooit gedacht door Watson en Crick, (of inderdaad, door de natuur zelf).
In nieuw onderzoek dat verschijnt in de geavanceerde online-editie van het tijdschrift Wetenschap , Mark Baden, samen met collega's Hao Yan van ASU en Wah Chiu van Baylor College of Medicine beschrijven een nieuwe methode voor het ontwerpen van geometrische vormen die zijn opgebouwd uit DNA. Ze presenteren een nieuwe variant op een techniek die bekend staat als DNA-origami, waarin de basenparende eigenschappen van DNA worden benut voor de constructie van minuscule structuren in 2 en 3 dimensies.
"Een belangrijke uitdaging op het gebied van DNA-nanotechnologie is om alle gewenste structuren top-down te ontwerpen, zonder veel menselijke inbreng met betrekking tot details van de vouwpaden van de DNA-streng, "zegt Jan.
Zijn medewerkers aan het MIT, onder leiding van Mark Bathe, ontwikkelde een computeralgoritme om DNA-nanostructuren te ontwerpen door alleen een doelvorm in te voeren. Ze ontwierpen een softwareplatform dat de benodigde DNA-strengen kan berekenen en uitvoeren om designerarchitecturen te vormen. De vorming van deze structuren werd vervolgens systematisch gekarakteriseerd en experimenteel bevestigd in de drie instituten. "Dit toont echt interdisciplinaire collaboratieve wetenschap in het hele land, " zegt Yan
Yan leidt het Biodesign Center for Molecular Design and Biomimetics aan de Arizona State University.
andere werelden
Het team ontwerpt bruikbare constructies op een verbazingwekkend kleine schaal. (Eén nanometer is een miljardste van een meter of ongeveer de grootte van een suikermolecuul.) Gespecialiseerde beeldvormingstechnieken, inclusief atomaire kracht- en cryo-elektronenmicroscopie worden gebruikt om de resulterende vormen te visualiseren.
De beschreven vereenvoudigde techniek belooft het gebruik van DNA-origami aanzienlijk uit te breiden tot buiten de gespecialiseerde gemeenschap en het scala aan mogelijke toepassingen in de biomoleculaire wetenschap en nanotechnologie uit te breiden. Deze omvatten het gebruik van nanodeeltjes voor medicijnafgifte en celtargeting, constructie van robots op nanoschaal die verschillende activiteiten in de geneeskunde en de industrie kunnen uitvoeren en het ontwerpen van op maat gemaakte optische apparaten.
Een van de meest opwindende innovaties aan de horizon is het gebruik van DNA als opslagmedium - een met een retentietijd van miljoenen jaren. (Een enkele gram DNA kan ongeveer 700 terabyte aan informatie bevatten - een hoeveelheid die overeenkomt met 14, 000 Blu-ray-schijven van 50 gigabyte. Verder, een dergelijk nucleïnezuurgeheugen kan mogelijk worden gebruikt met een fractie van de energie die nodig is voor andere opties voor informatieopslag.)
Shape-shifting
De nieuwe ontwerpmethode, die vrijwel elke veelvlakkige vorm kan produceren, berust op een top-downstrategie, die begint met een schets van de gewenste vorm en in fasen achteruit werkt om de vereiste DNA-sequentie te definiëren die zich op de juiste manier zal vouwen om het eindproduct te vormen.
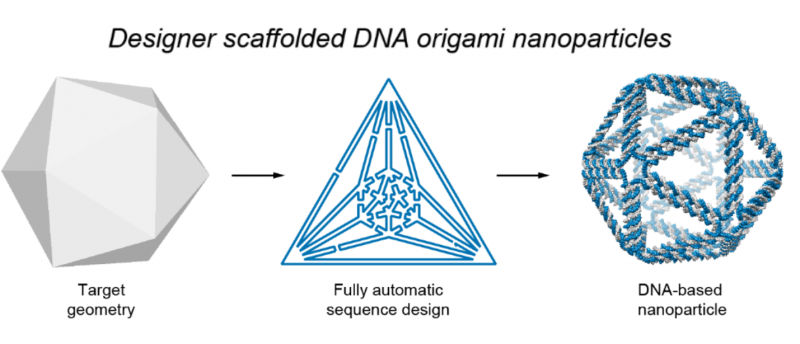
Afbeelding toont het basisproces dat wordt gebruikt om DNA-origami-nanostructuren te ontwerpen. Eerst, een wireframe van het beoogde doelontwerp wordt gemaakt. De software vertaalt dit vervolgens naar een plan voor de routering van DNA-steiger en stapelstrengen, die samenkomen om de gewenste vorm te vormen. Krediet:Biodesign Instituut
Het autonome proces wordt uitgevoerd met behulp van een door de auteurs ontworpen softwareprogramma. Bekend als DAEDALUS (voor DNA Origami Sequence Design Algorithm for User-Defined Structures), het programma voert inverse ontwerpen uit van willekeurige DNA-origami-nanovormen, gebaseerd op een input wireframe mesh, (een visuele weergave van de gesloten, 3-dimensionaal geometrisch oppervlak).
Het programma is niet alleen gebruiksvriendelijk, maar zeer veelzijdig, het produceren van vormen die niet beperkt zijn tot sferische topologie, (d.w.z., gesloten, tweezijdige structuren zonder grenzen of gaten). Zodra de doelvorm is beschreven als een netwerk van knooppunten en randen, DNA-steigerstrengen van aangepaste lengte en sequentie worden gegenereerd met behulp van technologie die bekend staat als asymmetrische polymerasekettingreactie.
De nieuwe studie beschrijft de fabricage van een verscheidenheid aan geometrische DNA-objecten, waaronder 35 veelvlakkige vormen (platonisch, Archimedisch, Johnson en Catalaanse vaste stoffen) 6 asymmetrische structuren, en vier veelvlakken met bolvormige topologie, met behulp van inverse ontwerpprincipes. De methode kan nanovormen met hoge betrouwbaarheid en stabiliteit produceren zonder het normale moeizame proces van het handmatig ontwerpen van basenparen om de beoogde doelstructuur te vormen.
De vouw betreden
DNA-origami brengt de oude Japanse methode van het vouwen van papier naar de moleculaire schaal. De basis is simpel:neem een stuk enkelstrengs DNA en leid het in een gewenste vorm, het aan elkaar bevestigen van de structuur met behulp van kortere zogenaamde stapelstrengen, die binden op strategische plaatsen langs de langere lengte van het DNA. De methode is gebaseerd op het feit dat de vier nucleotideletters van DNA - A, T, C, &G op een consistente manier bij elkaar blijven; Zoals altijd koppelen met Ts en Cs met Gs.
Het DNA-molecuul in zijn karakteristieke dubbelstrengige vorm is vrij stijf, vergeleken met enkelstrengs DNA, die flexibel is. Om deze reden, enkelstrengs DNA zorgt voor een ideaal veterachtig steigermateriaal. Verder, de koppelingseigenschappen zijn voorspelbaar en consistent, (in tegenstelling tot RNA, die als promiscue wordt beschouwd, vanwege basenparen die onverwacht kunnen zijn).
De techniek is enorm succesvol gebleken in het creëren van talloze vormen in 2- en 3-dimensies, die gemakkelijk zelf-assembleren wanneer de ontworpen DNA-sequenties met elkaar worden gemengd. Het lastige deel is het voorbereiden van de juiste DNA-sequentie en het routeringsontwerp voor steigers en stapelstrengen om de gewenste doelstructuur te bereiken. Typisch, dit is nauwgezet werk dat handmatig moet worden uitgevoerd.
Met de nieuwe techniek, de doelstructuur wordt eerst beschreven in termen van een gaas bestaande uit veelvlakken. Van dit, een spanning tree algoritme wordt gegenereerd. Dit is in feite een kaart die automatisch de route van de DNA-steigerstreng door de hele origami-structuur leidt, het aanraken van elk hoekpunt in de geometrische vorm, een keer. Complementaire stapelstrengen worden vervolgens toegewezen en de uiteindelijke vorm assembleert zichzelf.
Om de methode te testen, eenvoudigere vormen bekend als platonische lichamen werden voor het eerst gefabriceerd, gevolgd door steeds complexere structuren. Deze omvatten objecten met niet-sferische topologieën en ongebruikelijke interne details, die nooit eerder experimenteel was gerealiseerd.
De voltooide ontwerpen demonstreerden het vermogen van de top-down-techniek om automatisch steiger- en nietrouteringen te genereren voor een uitgebreide reeks nanovormen, uitsluitend gebaseerd op oppervlaktegeometrie. Cryo-EM werd gebruikt om de structurele betrouwbaarheid en stabiliteit van de geassembleerde origami-structuren te bevestigen.
Verdere experimenten bevestigden dat de geproduceerde DNA-structuren potentieel geschikt waren voor biologische toepassingen, aangezien ze stabiliteit op lange termijn vertoonden in serum en zoutarme omstandigheden.
Het onderzoek effent de weg voor de ontwikkeling van ontworpen systemen op nanoschaal die de eigenschappen van virussen nabootsen, fotosynthetische organismen en andere geavanceerde producten van natuurlijke evolutie.
Naast zijn aanstelling bij het Biodesign Institute, Hao Yan is de Milton D. Glick Distinguished Professor, College van Liberale Kunsten en Wetenschappen, School of Molecular Sciences aan de ASU.
 Interferentie leidt tot onnauwkeurige Raman-spectroscopische analyse van vitamine B12
Interferentie leidt tot onnauwkeurige Raman-spectroscopische analyse van vitamine B12 Ei-osmose-experimenten met gedestilleerd water en zout water
Ei-osmose-experimenten met gedestilleerd water en zout water Positieve of negatieve lading bepalen
Positieve of negatieve lading bepalen  De allereerste video met atomaire resolutie van zoutkristallen die zich in realtime vormen
De allereerste video met atomaire resolutie van zoutkristallen die zich in realtime vormen Wat zijn de gevaren van CO2-gas?
Wat zijn de gevaren van CO2-gas?
 Europa met dezelfde door lockdown veroorzaakte daling van de vervuiling als waargenomen in China
Europa met dezelfde door lockdown veroorzaakte daling van de vervuiling als waargenomen in China VS keuren landruil voor kopermijnen op Indiaans land goed
VS keuren landruil voor kopermijnen op Indiaans land goed Hebben bodems een zoutarm dieet nodig?
Hebben bodems een zoutarm dieet nodig? Effect van klimaatverandering op infectieziekten onbekend bij de helft van de bevolking
Effect van klimaatverandering op infectieziekten onbekend bij de helft van de bevolking Overstromingsschade Canada neemt toe, maar zware regenval neemt af
Overstromingsschade Canada neemt toe, maar zware regenval neemt af
Hoofdlijnen
- Vleermuizen anticiperen op optimale weersomstandigheden
- Wanneer dupliceren chromosomen tijdens een levenscyclus van cellen?
- Wat zijn endorfines?
- Stages of Mitosis (Cell Division)
- Technologie verhoogt melkgift met 9 procent
- Kijk hoe bacteriën muteren tot medicijnresistente superbacteriën
- Hoe kan een mutatie in DNA de eiwitsynthese beïnvloeden?
- De relatie tussen leeftijd en plasticiteit
- Japan doodt 177 walvissen in campagne in de Stille Oceaan:regering
- Biosensor-chip maakt zeer gevoelige eiwitanalyse mogelijk voor ziektediagnose
- Maak kennis met de krachtige diode met één molecuul

- Het kopiëren van het lock-and-key-systeem van de natuur kan de snelle medische diagnostiek verbeteren

- Nano- en micromotoren voor biologische en chemische toepassingen
- Wetenschappers creëren een voertuig op nanoschaal om kanker te bestrijden zonder gezonde cellen te beschadigen
 Simuleren van meteorietinslagen in het laboratorium
Simuleren van meteorietinslagen in het laboratorium Lokale emissies versterken regionale nevel en deeltjesgroei
Lokale emissies versterken regionale nevel en deeltjesgroei Gerichte evolutie van een designer-enzym met een onnatuurlijk katalytisch aminozuur
Gerichte evolutie van een designer-enzym met een onnatuurlijk katalytisch aminozuur Hoe goed is een wedstrijd? Statistieken in forensische vuurwapenidentificatie zetten
Hoe goed is een wedstrijd? Statistieken in forensische vuurwapenidentificatie zetten Drie componenten van ATP
Drie componenten van ATP Japanse ruimtevaartstartup wil concurreren met Amerikaanse rivalen
Japanse ruimtevaartstartup wil concurreren met Amerikaanse rivalen Indiase satellietvernietiging zorgde voor 400 stukken puin, ISS in gevaar brengen:NASA
Indiase satellietvernietiging zorgde voor 400 stukken puin, ISS in gevaar brengen:NASA Wetenschappers lossen 90 jaar oud meetkundeprobleem op
Wetenschappers lossen 90 jaar oud meetkundeprobleem op
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

