
Wetenschap
Onderzoekers ontwikkelen tools om 3D-weergave van grote RNA-moleculen te verscherpen
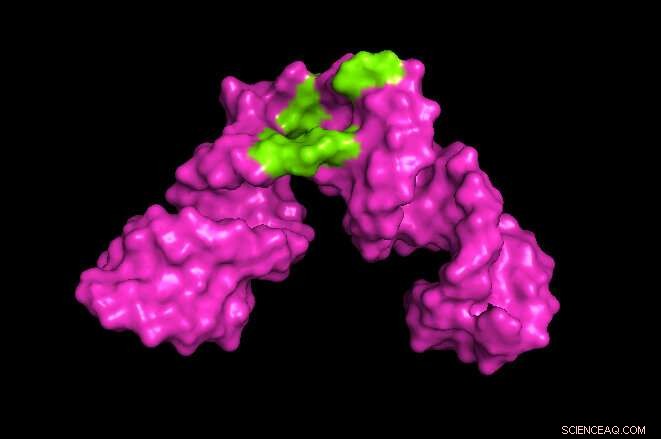
Onderzoekers van de Universiteit van Maryland ontwikkelden een methode om de reikwijdte van nucleaire magnetische resonantie (NMR) spectroscopie uit te breiden. In het bovenstaande voorbeeld de onderzoekers waren in staat om een 3D-beeld te maken dat de plaats op een stuk hepatitis-RNA onthulde waar kleine moleculen, zoals een medicijn, konden binden (in groen weergegeven). Krediet:Kwaku Dayie/Universiteit van Maryland
Wetenschappers van de Universiteit van Maryland hebben een methode ontwikkeld om de structuren van grote RNA-moleculen met hoge resolutie te bepalen. De methode overwint een uitdaging die de afgelopen 50 jaar 3D-analyse en beeldvorming van RNA heeft beperkt tot alleen kleine moleculen en stukjes RNA.
De nieuwe methode, die de reikwijdte van nucleaire magnetische resonantie (NMR) spectroscopie uitbreidt, zal onderzoekers in staat stellen de vorm en structuur van RNA-moleculen te begrijpen en te leren hoe ze interageren met andere moleculen. De inzichten die deze technologie oplevert, kunnen leiden tot gerichte RNA-therapeutische behandelingen voor ziekten. Het onderzoekspaper over dit werk is gepubliceerd in het tijdschrift wetenschappelijke vooruitgang op 7 oktober, 2020.
"Het veld van nucleaire magnetische resonantie spectroscopie is vastgelopen met kijken naar dingen die klein zijn, zeg 35 RNA-bouwstenen of nucleotiden. Maar de meeste interessante dingen die biologisch en medisch relevant zijn, zijn veel groter, 100 nucleotiden of meer, " zei Kwaku Dayie, een professor in de chemie en biochemie aan de UMD en senior auteur van de paper. "Dus, in staat zijn om de blokkade te doorbreken en naar dingen te kijken die groot zijn, is heel opwindend. Het zal ons in staat stellen om in deze moleculen te kijken en te zien wat er aan de hand is op een manier die we voorheen niet konden doen."
Bij NMR-spectroscopie, wetenschappers richten radiogolven op een molecuul, het prikkelen van de atomen en het "oplichten" van het molecuul. Door veranderingen in het magnetische veld rond de aangeslagen atomen te meten - de nucleaire magnetische resonantie - kunnen wetenschappers kenmerken reconstrueren zoals de vorm, structuur en beweging van het molecuul. De gegevens die dit oplevert, kunnen vervolgens worden gebruikt om afbeeldingen te genereren, net als MRI-beelden die in de geneeskunde worden gezien.
Gewoonlijk, NMR-signalen van de vele atomen in een biologisch molecuul zoals RNA overlappen elkaar, analyse erg moeilijk maken. Echter, In de jaren zeventig, wetenschappers leerden biochemisch RNA-moleculen te manipuleren om beter te werken met NMR door de waterstofatomen te vervangen door magnetisch actieve fluoratomen. In relatief kleine RNA-moleculen die uit 35 of minder nucleotiden bestaan, de fluoratomen lichten gemakkelijk op wanneer ze worden geraakt door radiogolven en blijven lang genoeg opgewonden voor analyse met hoge resolutie. Maar naarmate RNA-moleculen groter worden, de fluoratomen lichten slechts kort op, verliezen dan snel hun signaal. Dit heeft 3D-analyse met hoge resolutie van grotere RNA-moleculen voorkomen.
Eerder werk van anderen had aangetoond dat fluor een sterk signaal bleef produceren als het zich naast een koolstofatoom bevond dat zes protonen en zeven neutronen bevat (C-13). Dus, Dayie en zijn team ontwikkelden een relatief eenvoudige methode om het natuurlijk voorkomende C-12 in RNA (dat 6 protonen en 6 neutronen heeft) te veranderen in C-13 en er direct naast een fluoratoom (F-19) te installeren.
Dayie en zijn team toonden eerst aan dat hun methode gegevens en afbeeldingen kon produceren die gelijk zijn aan de huidige methoden door deze toe te passen op stukjes RNA van HIV die 30 nucleotiden bevatten. die eerder in beeld waren gebracht. Vervolgens pasten ze hun methode toe op stukjes Hepatitis B-RNA met 61 nucleotiden - bijna het dubbele van de eerdere NMR-spectroscopie die mogelijk was voor RNA.
Hun methode stelde de onderzoekers in staat om plaatsen op het hepatitis B-RNA te identificeren waar kleine moleculen binden en interageren met het RNA. Dat zou nuttig kunnen zijn om het effect van potentiële therapeutische medicijnen te begrijpen. De volgende stap voor de onderzoekers is het analyseren van nog grotere RNA-moleculen.
"Dit werk stelt ons in staat om uit te breiden wat in beeld kan worden gebracht, " zei Dayie. "Onze berekeningen vertellen ons dat, in theorie, we kunnen naar echt grote dingen kijken, als een deel van het ribosoom, dat is de moleculaire machine die eiwitten in cellen synthetiseert."
Door de vorm en structuur van een molecuul te begrijpen, wetenschappers kunnen de functie en de interactie met zijn omgeving beter begrijpen. Bovendien, deze technologie stelt wetenschappers in staat om de 3D-structuur te zien terwijl deze verandert, omdat met name RNA-moleculen vaak van vorm veranderen. Deze kennis is de sleutel tot het ontwikkelen van therapieën die zich specifiek richten op ziektespecifieke moleculen zonder de gezonde celfuncties aan te tasten.
"De hoop is dat als onderzoekers de hoeken en gaten kennen in een molecuul dat niet goed functioneert, dan kunnen ze medicijnen ontwerpen die alle hoeken en gaten vullen om het buiten werking te stellen, " zei Dayie. "En als we deze moleculen kunnen volgen terwijl ze van vorm en structuur veranderen, dan zal hun reactie op mogelijke medicijnen een beetje voorspelbaarder zijn, en het ontwerpen van medicijnen die effectief zijn, kan efficiënter zijn."
 Londen wordt 's werelds eerste nationale parkstad
Londen wordt 's werelds eerste nationale parkstad  Britse wijnbouwgebieden om champagne te evenaren onthuld
Britse wijnbouwgebieden om champagne te evenaren onthuld meer vee, meer kooldioxide, minder ijs:de vooruitgang van de klimaatverandering in de wereld sinds 2019
meer vee, meer kooldioxide, minder ijs:de vooruitgang van de klimaatverandering in de wereld sinds 2019 Droogte maakt Californische reservoirs zo heet, droge zomer weefgetouwen
Droogte maakt Californische reservoirs zo heet, droge zomer weefgetouwen Methode in de lucht om noordelijke meren en hun verband met klimaatverandering te begrijpen
Methode in de lucht om noordelijke meren en hun verband met klimaatverandering te begrijpen
Hoofdlijnen
- Hoe een 3D-model voor celbiologieprojecten te bouwen Mitochondria & Chloroplast
- Plasticity Cell Definition
- Een keep-fit gadget voor uw hond deze kerst - wie heeft er echt baat bij?
- Snelle papegaaien gefokt op roofdiervrije eilanden die met uitsterven worden bedreigd
- Een 3D-cel maken
- Oorsmeer zoals ijskernen - ontsluiten het verleden verborgen in oordopjes voor walvissen
- 'Welke dag is het ook alweer?' De psychologie van dinsdag
- Hoe de lengte van DNA-fragmenten te berekenen
- Beschrijving van gen-splitsing als een DNA-techniek
- Neutronen maken structurele veranderingen in moleculaire borstels zichtbaar

- Super sterk, biocompatibel materiaal kan kapotte vullingen tot het verleden maken
- Onderzoek levert nieuw ontwerpprincipe op voor watersplitsende katalysatoren

- Van ontziltingsafval een nuttige hulpbron maken

- Is stikstof brandbaar?

 Oud DNA uit Doggerland scheidt het VK van Europa
Oud DNA uit Doggerland scheidt het VK van Europa Onderzoekers maken in water afbreekbaar plastic dat zetmeel en cellulose combineert
Onderzoekers maken in water afbreekbaar plastic dat zetmeel en cellulose combineert Australië belooft te stoppen met het exporteren van zijn afval
Australië belooft te stoppen met het exporteren van zijn afval Paddenstoelenleer kan de sleutel zijn tot duurzame mode
Paddenstoelenleer kan de sleutel zijn tot duurzame mode Waarom technologie de mensenrechten in gevaar brengt
Waarom technologie de mensenrechten in gevaar brengt Het kan niet allemaal worden verzekerd:de verborgen economische impact van overstromingen en bosbranden tellen
Het kan niet allemaal worden verzekerd:de verborgen economische impact van overstromingen en bosbranden tellen VW in Canada veroordeeld tot betaling van 196,5 miljoen dollar voor emissieschandaal
VW in Canada veroordeeld tot betaling van 196,5 miljoen dollar voor emissieschandaal Onderzoekers theoretiseren de oorsprong van magnetars, de sterkste magneten in het universum
Onderzoekers theoretiseren de oorsprong van magnetars, de sterkste magneten in het universum
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

