
Wetenschap
Chemicus ontwikkelt 3D-simulaties van coronavirus-spike-eiwitten
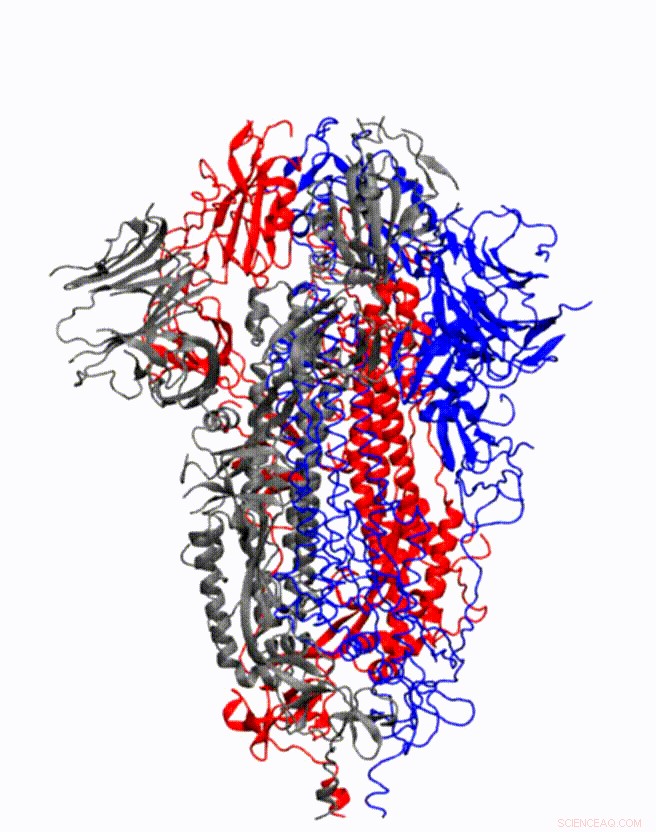
Animatie die laat zien hoe het coronavirus-spike-eiwit van vorm verandert vlak voordat het zich bindt aan de menselijke celreceptor. Credit:Illustratie geleverd door Mahmoud Moradi.
Computerchemicus Mahmoud Moradi zal verbeterde, 3D-simulaties van de moleculaire dynamica van coronavirus-piek-glycoproteïnen om beter te begrijpen hoe het virus zich bindt aan menselijke cellen.
Het in kaart brengen van hoe deze eiwitten conformationele veranderingen ondergaan om aan gastheercelreceptoren te binden, is van cruciaal belang voor de ontwikkeling van coronavirusvaccins en -therapieën. Simulaties zijn vooral belangrijk omdat een raamwerk voor het ontwerpen van geneesmiddelen dynamische, driedimensionale visualisaties van celstructuren en gedrag, in plaats van een statisch beeld.
"Net als bij andere virussen, een cruciale stap in het infectieproces van het coronavirus is het binnendringen van virussen, " zei Moradi, assistent-professor aan het J. William Fulbright College of Arts and Sciences. "Met coronavirussen, we weten dat deze spike-glycoproteïnen de toegang tot de menselijke cel bemiddelen. Zowel SARS-CoV-2, de oorzaak van COVID-19, en SARS-CoV, de oorzaak van de SARS-epidemie van 2002-2003, hebben spike-eiwitten die zich hechten aan dezelfde receptor in menselijke cellen."
Het werk van Moradi maakt deel uit van het COVID-19 High Performance Computing Consortium, een samenwerking van de overheid, industrie en academische partners gericht op computerbronnen voor COVID-19-onderzoek. Onder leiding van het Witte Huis Office of Science and Technology Policy, het Amerikaanse ministerie van Energie en IBM, het consortium stelt vrijwillig rekentijd en middelen vrij op enkele van 's werelds krachtigste supercomputers.
Om de simulaties uit te voeren, Moradi heeft toegang gekregen tot Frontera, een door de National Science Foundation gesponsorde supercomputer die is gehuisvest aan de Universiteit van Texas in Austin. Frontera is de grootste supercomputer op een universiteitscampus.
Moradi's project profiteert van verschillende recente, 3D-modellen met hoge resolutie van de spike-eiwitten van het coronavirus. Deze modellen kunnen worden gebruikt als initiële structuren om simulaties te starten die analyse van de gedetailleerde mechanismen van de eiwitten en hun gedrag bij virale binnenkomst mogelijk zullen maken. Versterkt, gedetailleerde simulaties van dergelijke moleculaire dynamica zullen een compleet beeld geven van de structurele veranderingen van eiwitten, en hoe ze binden aan angiotensine-converterend enzym 2, de specifieke menselijke celreceptor.
Moradi's onderzoek ligt op het snijvlak van biologie, natuurkunde, scheikunde, wiskunde, statistiek en informatica. Zijn biomoleculaire simulaties en computationele theorieën verklaren hoe eiwitten, de werkpaardmoleculen van cellen, functioneren op moleculair niveau. Zijn werk verbetert geometrische modellen om te beschrijven hoe eiwitten van vorm veranderen en hoe deze veranderingen het gedrag van een eiwit beïnvloeden. In februari, hij ontving een $ 650, 000 National Science Foundation Faculty Early Career Development-prijs voor dit werk.
 State-of-the-art klimaatmodel laat zien hoe we een crisis kunnen oplossen
State-of-the-art klimaatmodel laat zien hoe we een crisis kunnen oplossen Het boreale bos van Albertas zou tegen 2100 drastisch kunnen veranderen als gevolg van klimaatverandering, studie zegt:
Het boreale bos van Albertas zou tegen 2100 drastisch kunnen veranderen als gevolg van klimaatverandering, studie zegt: Het vet van het land:schatting van de ecologische kosten van te veel eten
Het vet van het land:schatting van de ecologische kosten van te veel eten Ozonafbrekende chemische alternatieven die in ons voedsel en water terechtkomen
Ozonafbrekende chemische alternatieven die in ons voedsel en water terechtkomen Wat is catastrofisme in de biologie?
Wat is catastrofisme in de biologie?
Hoofdlijnen
- Wat is de rol van ademhalingssystemen in homeostase?
- Pesticiden en slechte voeding schaden de diergezondheid
- Wat is sterker:nature of nurture?
- De gemiddelde levensduur van skeletspiercellen
- Wat zijn de voordelen van prokaryoten?
- Rangorde gebruiken om complexe genetische interacties te identificeren
- De genetische handtekening van de zeeschelp lezen
- Een bacterieel stroomdiagram maken
- Basiscelfuncties
- Op zoek naar een uit-schakelaar voor coeliakie
- Nieuw onderzoek creëert neutraliserende spons voor gevaarlijke chemicaliën

- Biostasis heeft tot doel de dood na traumatisch letsel te voorkomen door biochemische reacties in cellen te vertragen

- Materiaal voor veiligere voetbalhelmen kan hoofdletsel verminderen

- Bouwpakket voor op maat ontworpen producten

 NASA telt record regen in Australië op
NASA telt record regen in Australië op #MeToo:Onderzoek wijst uit dat seksuele intimidatie in wetenschappelijke bibliotheken vaker voorkomt dan gedacht
#MeToo:Onderzoek wijst uit dat seksuele intimidatie in wetenschappelijke bibliotheken vaker voorkomt dan gedacht Hoe sociale media het moeilijk maken om echt nieuws te identificeren
Hoe sociale media het moeilijk maken om echt nieuws te identificeren Wat maakt magneten sterk?
Wat maakt magneten sterk?  Nieuwe Canadese telescoop zal grootste volume ooit onderzochte ruimte in kaart brengen
Nieuwe Canadese telescoop zal grootste volume ooit onderzochte ruimte in kaart brengen Facebook plant loonsverhoging voor inhoudsmoderators
Facebook plant loonsverhoging voor inhoudsmoderators Over Bleach Neutralizers
Over Bleach Neutralizers  Orkaan Chriss oog staart naar NASA's Aqua-satelliet
Orkaan Chriss oog staart naar NASA's Aqua-satelliet
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

