
Wetenschap
Ontwikkeling decoderen:mRNA's spelen een rol bij de vorming van embryo's
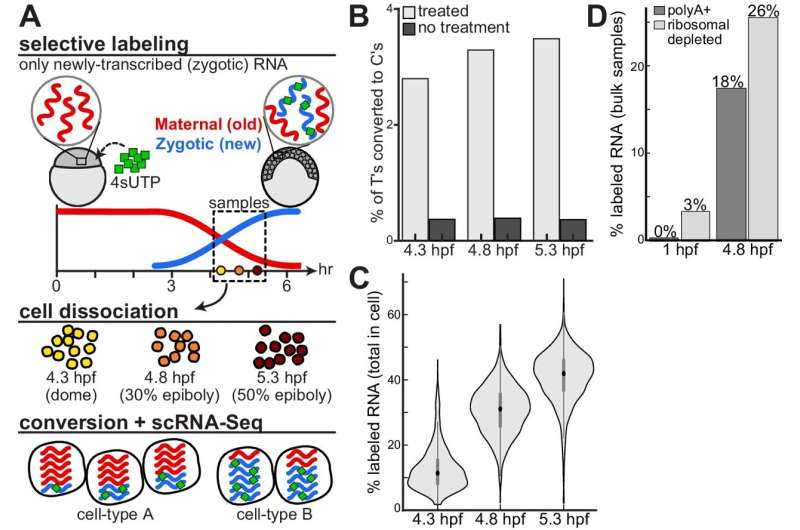
Een nieuwe studie aan de Hebreeuwse Universiteit onthult inzichten in de mRNA-regulatie tijdens de embryonale ontwikkeling. De studie werpt licht op het ingewikkelde proces van mRNA-regulatie tijdens de embryonale ontwikkeling en biedt nieuwe inzichten in de manier waarop pluripotente cellen gespecialiseerde identiteiten aannemen door middel van genexpressie.
Het werk is gepubliceerd in het tijdschrift Nature Communications Het onderzoek werd geleid door Ph.D. Student Lior Fishman en team onder leiding van onderzoeker Dr. Michal Rabani van het Alexander Silberman Institute of Life Science aan de Hebreeuwse Universiteit, en in samenwerking met onderzoekers van de National Institutes of Health in de VS
Embryonale ontwikkeling houdt in dat pluripotente cellen een gespecialiseerde identiteit aannemen door bepaalde genexpressieprofielen aan te nemen. Het begrijpen van de relatieve bijdragen van mRNA-transcriptie en -degradatie aan het vormgeven van deze profielen was echter een uitdaging, vooral bij embryo's met diverse cellulaire identiteiten.
In het onderzoek gebruikten onderzoekers een techniek genaamd single-cell RNA-sequencing, samen met metabolische labeling, om bij te houden hoe genen in de loop van de tijd worden in- en uitgeschakeld in zebravisembryo's. Ze konden het mRNA dat nieuw was gemaakt (van het embryo zelf) onderscheiden van het mRNA dat er al was (van de moeder). Met behulp van wiskundige modellen maten ze hoe snel genen in verschillende soorten cellen werden in- en uitgeschakeld terwijl ze zich ontwikkelden.
De resultaten van het onderzoek laten zeer uiteenlopende reguleringspercentages zien voor duizenden genen. De onderzoekers observeerden gecoördineerde transcriptie- en vernietigingssnelheden voor veel transcripten en koppelden verschillen in degradatie aan specifieke sequentie-elementen. Belangrijk is dat ze celtype-specifieke verschillen in afbraak identificeerden, waaronder selectieve retentie van maternale transcripten in primordiale kiemcellen en omhullende laagcellen, twee van de vroegst gespecificeerde celtypen.
Dr. Rabani, senior auteur van de studie, merkte op:"Onze studie biedt een kwantitatieve benadering voor het bestuderen van mRNA-regulatie tijdens een dynamische spatio-temporele respons. Dit werk opent nieuwe wegen voor het begrijpen van de moleculaire mechanismen die ten grondslag liggen aan het bepalen van het lot van cellen tijdens de embryonale ontwikkeling. "
De bevindingen van deze studie dragen bij aan een dieper begrip van mRNA-regulatie en de rol ervan bij het vormgeven van cellulaire identiteiten tijdens de embryonale ontwikkeling. Het onderzoeksteam hoopt dat hun werk de weg zal vrijmaken voor toekomstige studies gericht op het ontrafelen van de complexiteit van genexpressieregulatie in verschillende biologische contexten.