
Wetenschap
Onderzoekers verhelderen de ruimtelijke structuur en moleculaire mechanismen van Prime Editor, een nieuw hulpmiddel voor het bewerken van genen
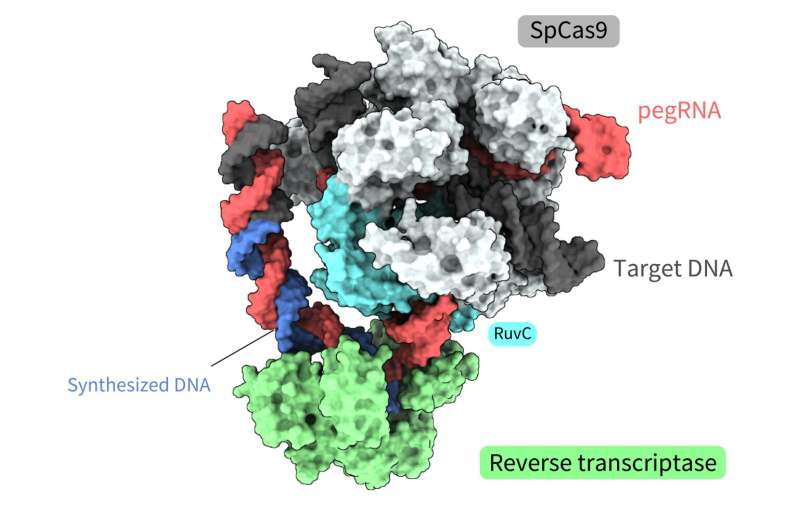
Gezamenlijk onderzoek onder leiding van Yutaro Shuto, Ryoya Nakagawa en Osamu Nureki van de Universiteit van Tokio bepaalde de ruimtelijke structuur van verschillende processen van een nieuw hulpmiddel voor het bewerken van genen, genaamd 'prime editor'. Functionele analyse op basis van deze structuren onthulde ook hoe een 'prime editor' omgekeerde transcriptie kon bereiken, waarbij DNA uit RNA werd gesynthetiseerd, zonder beide strengen van de dubbele helix te 'knippen'.
Het verduidelijken van deze moleculaire mechanismen draagt in grote mate bij aan het ontwerpen van hulpmiddelen voor genbewerking die nauwkeurig genoeg zijn voor gentherapiebehandelingen. De bevindingen worden gepubliceerd in het tijdschrift Nature .
De Nobelprijs voor de Scheikunde 2020 werd toegekend aan Jennifer Doudna en Emmanuelle Charpentier voor het ontwikkelen van een baanbrekende maar toch eenvoudige manier om DNA, de ‘blauwdruk’ van levende organismen, te bewerken. Hoewel hun ontdekking nieuwe wegen voor onderzoek opende, beperkten de nauwkeurigheid van de methode en de veiligheidsproblemen over het "knippen" van beide DNA-strengen het gebruik ervan voor gentherapiebehandelingen. Daarom wordt er onderzoek gedaan naar de ontwikkeling van tools die deze nadelen niet hebben.
Het Prime Editing-systeem is zo'n hulpmiddel, een molecuulcomplex dat uit twee componenten bestaat. Eén component is de hoofdeditor, die een SpCas9-eiwit combineert, gebruikt in de eerste CRISPR-Cas-genbewerkingstechnologie, en een reverse transcriptase, een enzym dat RNA in DNA transcribeert.
De tweede component is het primaire bewerkingsgids-RNA (pegRNA), een gemodificeerd gids-RNA dat de doelsequentie binnen het DNA identificeert en codeert voor de gewenste bewerking. In dit complex werkt de hoofdeditor als een 'tekstverwerker', die nauwkeurig genomische informatie vervangt. Het hulpmiddel is al met succes geïmplementeerd in levende cellen van organismen zoals planten, zebravissen en muizen. Hoe dit molecuulcomplex precies elke stap van het bewerkingsproces uitvoert, is echter nog niet duidelijk, vooral vanwege een gebrek aan informatie over de ruimtelijke structuur ervan.
"We werden nieuwsgierig naar hoe de onnatuurlijke combinatie van eiwitten Cas9 en reverse transcriptase samenwerken", zegt Shuto, de eerste auteur van het artikel.
Het onderzoeksteam maakte gebruik van cryogene elektronenmicroscopie, een beeldvormingstechniek die observaties op bijna atomaire schaal mogelijk maakt. De methode vereiste dat de monsters zich in glazig ijs bevonden om ze te beschermen tegen de mogelijke schade door de elektronenbundels, wat een aantal extra uitdagingen met zich meebracht.
"We ontdekten dat het hoofdredacteurcomplex onder experimentele omstandigheden onstabiel was", legt Shuto uit. "Het was dus een hele uitdaging om de omstandigheden voor het complex te optimaliseren om stabiel te blijven. Lange tijd konden we alleen de structuur van Cas9 bepalen."
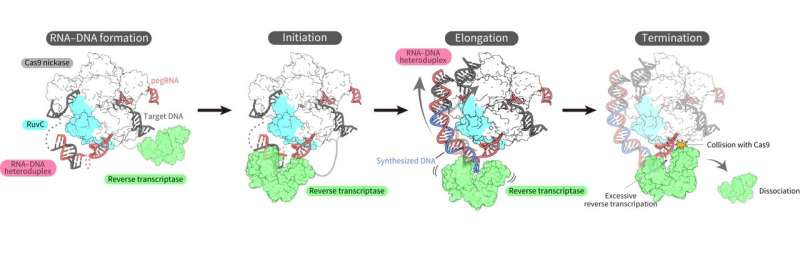
Nadat ze de uitdagingen eindelijk hadden overwonnen, slaagden de onderzoekers erin de driedimensionale structuur van het hoofdredacteurcomplex in meerdere toestanden te bepalen tijdens omgekeerde transcriptie op het doel-DNA.
Uit de structuren bleek dat de reverse transcriptase zich bond aan het RNA-DNA-complex dat zich vormde langs het "deel" van het Cas9-eiwit dat geassocieerd was met DNA-splitsing, de splitsing van een enkele streng van de dubbele helix. Tijdens het uitvoeren van de reverse transcriptie behield de reverse transcriptase zijn positie ten opzichte van het Cas9-eiwit. De structurele en biochemische analyses gaven ook aan dat de reverse transcriptase zou kunnen leiden tot extra, ongewenste inserties.
Deze bevindingen hebben nieuwe wegen geopend voor zowel fundamenteel als toegepast onderzoek. Daarom legt Shuto de volgende stappen uit.
"Onze structuurbepalingsstrategie in dit onderzoek kan ook worden toegepast op prime-editors die zijn samengesteld uit een ander Cas9-eiwit en reverse transcriptase. We willen de nieuw verkregen structurele informatie gebruiken om te leiden tot de ontwikkeling van verbeterde prime-editors."
Meer informatie: Structurele basis voor pegRNA-geleide reverse transcriptie door de hoofdredacteur, Nature (2024). DOI:10.1038/s41586-024-07497-8, https://www.nature.com/articles/s41586-024-07497-8
Journaalinformatie: Natuur
Aangeboden door Universiteit van Tokio
 Mini-centrifuge voor eenvoudigere studie van bloedcellen opent nieuwe organ-on-chip mogelijkheden
Mini-centrifuge voor eenvoudigere studie van bloedcellen opent nieuwe organ-on-chip mogelijkheden Wetenschappers ontwikkelen een composietmembraan voor op zink gebaseerde stroombatterijen met een lange levensduur
Wetenschappers ontwikkelen een composietmembraan voor op zink gebaseerde stroombatterijen met een lange levensduur De grenzen van kooldioxidefixatie verleggen
De grenzen van kooldioxidefixatie verleggen Onderzoekers ontwikkelen flexibel kristal, de weg vrijmaken voor efficiëntere buigbare elektronica
Onderzoekers ontwikkelen flexibel kristal, de weg vrijmaken voor efficiëntere buigbare elektronica Wetenschappers leggen de eerste cryo-EM-beelden vast van cellulair doelwit voor diabetes type 2 in actie
Wetenschappers leggen de eerste cryo-EM-beelden vast van cellulair doelwit voor diabetes type 2 in actie
 Onderzoekers noemen nieuwe oceaanzone:The rariphotic
Onderzoekers noemen nieuwe oceaanzone:The rariphotic De aarde verduistert door klimaatverandering
De aarde verduistert door klimaatverandering Arctisch zee-ijs bezwijkt voor Atlantificatie
Arctisch zee-ijs bezwijkt voor Atlantificatie Enge statistiek:90,5 procent van het plastic wordt niet gerecycled
Enge statistiek:90,5 procent van het plastic wordt niet gerecycled Hoe landen in conflictgebieden kunnen herstellen van overstromingen – lessen uit Pakistan
Hoe landen in conflictgebieden kunnen herstellen van overstromingen – lessen uit Pakistan
Hoofdlijnen
- Genen dicteren niet altijd dat jongens jongens zullen zijn
- Deze pijnbomen leunen bijna altijd naar de evenaar
- Voor het eerst identificeert onderzoek de belangrijkste trends in de biologische effecten van verstedelijking
- Wat zijn de rollen van chlorofyl A & B?
- Hoe celbrochures te maken
- Elandenpopulatie in Vermont worstelt ondanks bezuiniging op jacht
- De effecten van tornado's op mens en natuur
- Hoe is een Paramecium Digest Food?
- Forensische wetenschapsprojecten voor middelbare scholieren
 Hoe gevaarlijke vulkanen beter te identificeren?
Hoe gevaarlijke vulkanen beter te identificeren? Wat zal er gebeuren als je wordt blootgesteld aan paddenstoelenporen?
Wat zal er gebeuren als je wordt blootgesteld aan paddenstoelenporen?  Roofzuchtige bacteriën - de zoektocht naar een nieuwe klasse antibiotica
Roofzuchtige bacteriën - de zoektocht naar een nieuwe klasse antibiotica Onderzoek verklaart resistentie van het publiek tegen vaccinatie
Onderzoek verklaart resistentie van het publiek tegen vaccinatie Het Zwitserse zakmes onder de rookgordijnen
Het Zwitserse zakmes onder de rookgordijnen Foxconn-fabrieksbanen aangeprezen door Trump zullen niet doorgaan
Foxconn-fabrieksbanen aangeprezen door Trump zullen niet doorgaan Australië-gebonden asteroïde-monster kan de oorsprong van levens onthullen
Australië-gebonden asteroïde-monster kan de oorsprong van levens onthullen Wetenschappers lossen puzzel op om grafiet in diamant te veranderen
Wetenschappers lossen puzzel op om grafiet in diamant te veranderen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com






