
Wetenschap
Weten eiwitten het nog?
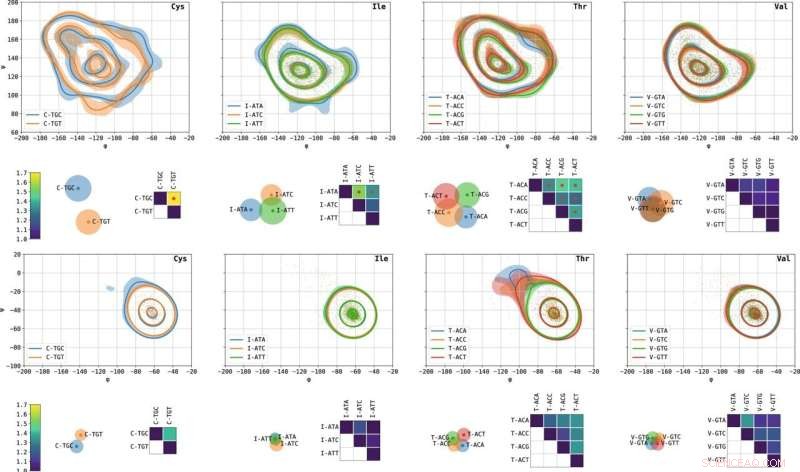
Codon-specifieke Ramachandran-plots van geselecteerde aminozuren en afstanden daartussen. Van links naar rechts worden cysteïne, isoleucine, threonine en valine weergegeven. Contourplots geven de niveaulijnen weer die 10, 50 en 90% van de waarschijnlijkheidsmassa bevatten. Gearceerde regio's vertegenwoordigen 10%-90% betrouwbaarheidsintervallen berekend op 1000 willekeurige bootstraps. De β- (boven) en α- (onder) modi worden afgebeeld. De matrices tonen L1 afstanden tussen paren van codonspecifieke Ramachandran-plots, genormaliseerd zodat de zelfafstand 1 is. Rode stippen geven paren aan met significant verschillende dihedrale hoekverdelingen op basis van hun p-waarde. De spreidingsgrafieken die de afstandsmatrices visualiseren, werden verkregen door een variant van multidimensionale schaling (MDS). Elk punt vertegenwoordigt een codon; paarsgewijze Euclidische afstanden tussen de punten benadert de L1 afstand tussen de corresponderende codons. Cirkels benaderen de onzekerheidsstralen. Hoe meer twee cirkels elkaar overlappen, des te minder zijn de overeenkomstige codonspecifieke Ramachandran-plots te onderscheiden. Krediet:Natuurcommunicatie (2022). DOI:10.1038/s41467-022-30390-9
Een studie waarin biologische ideeën en nieuwe computerwetenschappelijke hulpmiddelen zijn geïntegreerd, heeft nieuwe associaties blootgelegd tussen genetische codering en eiwitstructuur, die mogelijk de manier kunnen veranderen waarop we denken over eiwitproductie in het ribosoom - de 'eiwitassemblagelijn' van de cel. Het onderzoek, geleid door professor Alex Bronstein, Dr. Ailie Marx, en Ph.D. student Aviv Rosenberg, werd gepubliceerd in Nature Communications .
Eiwitten, de complexe moleculen die een cruciale rol spelen in vrijwel elk biologisch mechanisme, worden geproduceerd door ribosomen in een proces dat translatie wordt genoemd. Het ribosoom decodeert binnenkomende "genetische instructies" om ketens van aminozuren te synthetiseren - de bouwstenen van eiwitten. Wanneer aminozuren achtereenvolgens aan elkaar worden gebonden tot een lange keten, vouwen ze zich op tot een unieke driedimensionale structuur die het eiwit zijn biologische eigenschappen en functionaliteit verleent. Fouten in de vertaling kunnen leiden tot verkeerd vouwen en vervolgens tot fysiologische stoornissen, zowel milde als ernstige.
Instructies voor eiwitproductie worden aan het ribosoom geleverd als codons, sequenties van drie "letters" van de genetische nucleotidecode, die de identiteit en volgorde specificeren van aminozuren die door het ribosoom aan de eiwitketen moeten worden toegevoegd. Het codon UUU signaleert bijvoorbeeld voor toevoeging van het aminozuur fenylalanine, terwijl codon UAC instrueert voor de toevoeging van tyrosine. Op deze manier codeert de codonsequentie voor de unieke sequentie van aminozuren die kenmerkend is voor elk eiwit. Deze toewijzing van genetische codons aan aminozuren die bij de vertaling worden gebruikt, is gebruikelijk voor alle levende wezens op de planeet en wordt als een oermechanisme beschouwd.
Alsof dit alles nog niet ingewikkeld genoeg was, is het belangrijk erop te wijzen dat er 61 codons zijn die worden gedecodeerd in slechts 20 aminozuren. Met andere woorden, alle aminozuren op twee na worden gecodeerd door meerdere codons.
Hier komt het huidige onderzoek in beeld. Op basis van experimenten die in de jaren zestig en zeventig werden uitgevoerd, stelt het geaccepteerde dogma dat eiwitten geen "geheugen" dragen van het specifieke codon waaruit elk aminozuur is vertaald zolang de aminozuuridentiteit onveranderd blijft. Deze vroege experimenten met eiwitvouwing gebruikten chemische denaturatiemiddelen om volledig gevormde eiwitten te ontvouwen en toonden vervolgens aan dat na verwijdering van deze chemicaliën de eiwitketen spontaan opnieuw kon vouwen om zijn oorspronkelijke structuur en functie terug te krijgen. Deze experimenten suggereerden dat alleen de aminozuursequentie, en niet de specifieke codonsequentie, de structuur van een eiwit bepaalt. Met het oog op dit dogma worden mutaties die de genetische codering veranderen zonder het aminozuur te veranderen, algemeen als "stil" genoemd en als onbelangrijk beschouwd voor de eiwitstructuur en -functie.
Het Technion-onderzoeksteam heeft een verband ontdekt tussen de identiteit van het codon en de lokale structuur van het getranslateerde eiwit, wat suggereert dat dit misschien niet het algemene geval is en dat eiwitten zich inderdaad de specifieke instructies kunnen "herinneren" waaruit ze zijn gesynthetiseerd. Het onderzoeksteam analyseerde duizenden driedimensionale eiwitstructuren met behulp van speciale tools die ze ontwikkelden, die geavanceerde computerwetenschappelijke methoden, machine learning en statistieken integreren. Op deze manier vergeleken ze nauwkeurig de verdelingen van hoeken gevormd in deze structuren onder verschillende synonieme genetische codes. Hun bevindingen tonen aan dat er voor bepaalde codons een significante statistische afhankelijkheid bestaat tussen de identiteit van het codon en de lokale structuur van het eiwit, op de positie van het aminozuur dat door dat codon wordt gecodeerd.
De onderzoekers benadrukken dat de bevindingen nog geen licht kunnen werpen op de richting van het causale verband, waardoor het nog niet mogelijk is om te zeggen of een verandering in genetische codering een verandering in de lokale eiwitstructuur kan veroorzaken of dat structurele veranderingen kunnen leiden tot verschillende codering, bijvoorbeeld door evolutionaire processen. This question is the foundation for a subsequent research study now being carried out by the group. According to Dr. Marx, a biologist by training and education, "If we find in subsequent research that the codon indeed has a causal effect on protein folding, this is likely to have a huge impact on our understanding of protein folding, as well as on future applications, such as engineering new proteins."
Dr. Marx emphasizes that the discovery presented in the article would not have been possible without Prof. Bronstein's computer and analysis skills. "This research is truly interdisciplinary, because biology alone cannot cope with such vast quantities of data without the help of data science, and computer scientists cannot themselves perform research of this kind, since they lack familiarity with the complex biological processes being probed. Therefore, our research highlights the huge advantage of interdisciplinary research that integrates skills from different fields to create a whole that is greater than the sum of its parts."
 Onderzoekers ontdekken dat traditionele waarnemingen van vloeistofstromen het grote geheel kunnen missen
Onderzoekers ontdekken dat traditionele waarnemingen van vloeistofstromen het grote geheel kunnen missen Video:Brengen menselijke begrafenispraktijken de ecosystemen van de aarde in de war?
Video:Brengen menselijke begrafenispraktijken de ecosystemen van de aarde in de war? Neutronen optimaliseren hoogrendementskatalysator voor groenere benadering van biobrandstofsynthese
Neutronen optimaliseren hoogrendementskatalysator voor groenere benadering van biobrandstofsynthese Kleine robot kan game-changer zijn in de strijd tegen tuberculose
Kleine robot kan game-changer zijn in de strijd tegen tuberculose Nieuwe generatie kooldioxidegasscheidingssysteem met adsorbentia van het gate-type
Nieuwe generatie kooldioxidegasscheidingssysteem met adsorbentia van het gate-type
 Een groeiende klimaatniche voor gigantische bomen
Een groeiende klimaatniche voor gigantische bomen Oudste Amerikanen waren het meest gericht op het verminderen van voedselverspilling
Oudste Amerikanen waren het meest gericht op het verminderen van voedselverspilling Modelmanier om bomen te beschermen
Modelmanier om bomen te beschermen Berlijnse volkstuinders graven zich in tegen projectontwikkelaars
Berlijnse volkstuinders graven zich in tegen projectontwikkelaars SimRadar:een polarimetrische radar-tijdreekssimulator voor tornadisch puinonderzoek
SimRadar:een polarimetrische radar-tijdreekssimulator voor tornadisch puinonderzoek
Hoofdlijnen
- Verspilde vogelveren veranderd in voedsel
- Productietijden kunnen het illegaal witwassen van wilde dieren tegengaan
- Wat beïnvloedt een Trait Expression de meeste, genetica of de omgeving?
- Een nieuw gen bij zoogdieren dat een nieuwe structuur in zenuwcellen aanstuurt
- De controversiële handel in bushmeat herkaderen:wie bepaalt welke voedingsmiddelen geschikt zijn voor consumptie?
- Kaarten uit de Eerste Wereldoorlog helpen de geschiedenis van kelpbossen in Pacific Northwest te volgen
- Tips voor bedwantsen voor vakantiereizigers
- Zenuwen controleren de bacteriële gemeenschap van het lichaam
- Kunnen algen de geheimen van fotosynthese ontsluiten?
- Onderzoek naar roodrugspinnen vindt dat schijnbaar weerzinwekkende paringsstrategie zowel mannen als vrouwen ten goede komt

- Nest van bedreigde reuzenweekschildpad gevonden in Cambodja

- Vijf stappen om Agar te bereiden Slants

- Welke materialen kan ik gebruiken om een DNA-model te maken?

- Nieuwe studie onthult mechanisme voor hoe ziekteverspreidende prionen van de ene soort naar de andere kunnen springen
 Kleine technologie kan grote vondsten opleveren - en mogelijke gevaren
Kleine technologie kan grote vondsten opleveren - en mogelijke gevaren Privacyboete braaklegging deukt Facebooks winst
Privacyboete braaklegging deukt Facebooks winst Q&A:Hoe klimaatverandering, andere factoren veroorzaken branden in Australië
Q&A:Hoe klimaatverandering, andere factoren veroorzaken branden in Australië Welke statistische analyse voer ik uit bij het vergelijken van drie dingen met elkaar?
Welke statistische analyse voer ik uit bij het vergelijken van drie dingen met elkaar?  ALMA vindt enorme oerstelsels die zwemmen in een enorme oceaan van donkere materie
ALMA vindt enorme oerstelsels die zwemmen in een enorme oceaan van donkere materie Oude interventie zou de slinkende waterreserves in de kust van Peru kunnen stimuleren
Oude interventie zou de slinkende waterreserves in de kust van Peru kunnen stimuleren Waterlek apparatuur verkort ruimtewandeling door twee Amerikaanse astronauten
Waterlek apparatuur verkort ruimtewandeling door twee Amerikaanse astronauten Energie besparen bij de productie van chemicaliën
Energie besparen bij de productie van chemicaliën
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

