
Wetenschap
Wetenschappers combineren disciplines om kleine structuren in ongelabelde moleculen te lokaliseren
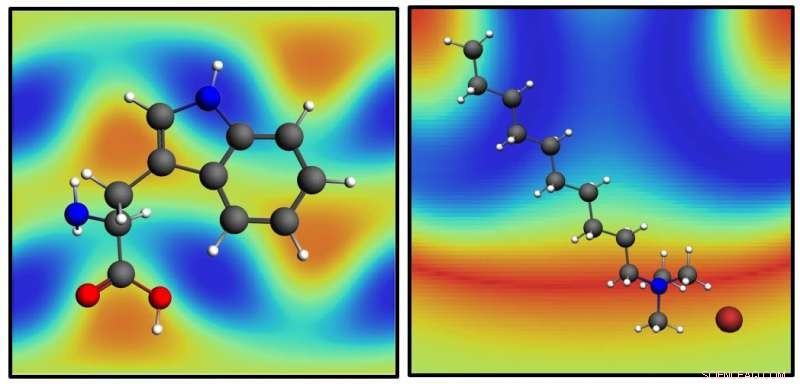
De moleculen tryptofaan, links, en decyltrimethylammoniumbromide, Rechtsaf, over hun SABERS-kaarten. SABELS, een nieuwe analysemethode ontwikkeld aan de Rice University, is in staat om structurele details van moleculen in lipidemembranen in de buurt van gouden nanodeeltjes te verkrijgen zonder moleculaire tags. Krediet:Hafner Lab/Rice University
Vijf jaar hard werken en een beetje "kosmisch geluk" leidden de Rice University-onderzoekers naar een nieuwe methode om structurele details van moleculen in biomembranen te verkrijgen.
De methode van het Rice-lab van natuurkundige Jason Hafner combineert experimentele en computationele technieken en is gebaseerd op de plasmonische eigenschappen van gouden nanodeeltjes. Het maakt gebruik van het unieke vermogen van de nanodeeltjes om licht te focussen op zeer kleine doelen.
De onderzoekers noemen hun protocol SABERS, voor structurele analyse door verbeterde Raman-verstrooiing, en zeggen dat het wetenschappers kan helpen die amyloïde-interacties bestuderen die betrokken zijn bij neurodegeneratieve ziekten, de neuroprotectieve werking van vetzuren en de functie van chemotherapiemiddelen.
De details verschijnen deze maand in het tijdschrift American Chemical Society Nano-letters .
Hun methode extraheert de locatie van specifieke chemische groepen in de moleculen door hun karakteristieke trillingen te lokaliseren. Wanneer een laser plasmonen in de nanodeeltjes activeert, het versterkt vibrerend verstrooid licht van nabijgelegen moleculen, een fenomeen dat oppervlakteversterkte Raman-verstrooiing (SERS) wordt genoemd. De verbetering is gevoelig voor precies waar het molecuul zit ten opzichte van het nanodeeltje.
"Moleculen kunnen op veel verschillende manieren trillen, dus we moeten aan elk een 'trillingscentrum' toewijzen, "Zei Hafner. "Als je een deel van een molecuul ziet trillen, je kunt visualiseren waar het zich voordoet, maar we moesten ook een wiskundige manier vinden om het te beschrijven."
SERS-spectra zijn notoir moeilijk te ontwarren, dus de volledige SABERS-methode vereist ook niet-versterkte spectrale metingen en theoretische berekeningen van zowel de nanorod-optiek als de moleculaire eigenschappen, hij zei.
Hafner en zijn team testten hun techniek op drie structuren:oppervlakteactieve moleculen die worden geleverd met gouden nanostaafjes, lipidemoleculen die membranen vormen op gouden nanostaafjes en tryptofaan, een aminozuur dat zich in het membraan nestelt.
"We ontdekten dat de oppervlakteactieve laag 25 graden is gekanteld, wat interessant is omdat het verklaart waarom uit andere metingen bleek dat de laag dunner lijkt dan verwacht, ’ zei Hafner.
Lipiden vervangen gemakkelijk oppervlakteactieve stoffen op nanostaafjes omdat ze in dezelfde chemische structuur eindigen. Door trillingen van die structuur in de lipidekopgroep te vergelijken met een dubbele binding in de staart, SABERS vond de juiste oriëntatie en dikte van het lipide dubbellaags membraan. "Het is gewoon kosmisch geluk dat een lipide eindigt in een perfect symmetrische structuur die trilt en Raman-actief is en graag op een nanostaafje zit, ’ zei Hafner.
De onderzoekers gebruikten ook SABERS om tryptofaan in de lipidedubbellaag te lokaliseren. "Het is heel helder, spectroscopisch, en gemakkelijk te zien, " zei hij. "In echte biologische structuren, tryptofaan is slechts een klein residu gehecht aan een veel groter eiwit. Echter, tryptofaan helpt het eiwit aan het membraan te verankeren, dus onderzoekers willen weten waar hij het liefst zit."
Volgende, Hafner wil grotere moleculen analyseren. "In principe, door middel van spectroscopische trucs, we zouden dit naar grotere structuren kunnen brengen, en misschien zelfs elk residu in een eiwit vinden om de hele structuur te krijgen. Dat is futuristisch, maar het is waar we denken dat we ermee kunnen gaan, " hij zei.
Hoofdlijnen
- Trofisch niveau (voedselketen en web): definitie en voorbeelden (met diagram)
- Onderzoekers lokaliseren genetische mutatie bij chronische nachtbrakers
- Wat doet veel van de activiteiten van een cel?
- De taal van cellulaire communicatie begrijpen
- Geurende reinigingsproducten: The New Smoking?
- Waarom verhuizen chloroplasten in Elodea?
- Waarschijnlijkheden in de genetica: waarom is het belangrijk?
- De oudste kleur van de aarde was roze
- Hypermutators zorgen ervoor dat pathogene schimmels sneller evolueren
- Aan een zijden draadje hangen:beeldvorming en sondeerketens van enkele atomen
- Eenvoudig printen van biosensoren gemaakt van grafeen

- Berekeningen laten zien hoe mengsels van verschillende elementen de thermische eigenschappen van nanodraden kunnen regelen
- Project heeft tot doel nanopetals in massa te produceren voor sensoren, batterijen
- Magnetische nanodeeltjes kunnen de sleutel zijn tot effectieve immunotherapie
 Magische hoek grafeen en het creëren van onverwachte topologische kwantumtoestanden
Magische hoek grafeen en het creëren van onverwachte topologische kwantumtoestanden Kaketoes matchen objecten in corresponderende frames in een taak met gereedschap
Kaketoes matchen objecten in corresponderende frames in een taak met gereedschap De hoogte van een driehoek vinden
De hoogte van een driehoek vinden Inzicht in voorspelbaarheid en willekeur door in de modder te graven
Inzicht in voorspelbaarheid en willekeur door in de modder te graven Mannelijke en vrouwelijke reproductieve delen van een bloem
Mannelijke en vrouwelijke reproductieve delen van een bloem  Hoe kan recycling een gemeenschap beïnvloeden?
Hoe kan recycling een gemeenschap beïnvloeden?  Deze nieuwe technieken stellen je browsegeschiedenis bloot aan aanvallers
Deze nieuwe technieken stellen je browsegeschiedenis bloot aan aanvallers Onopgeloste puzzels in exotische kernen
Onopgeloste puzzels in exotische kernen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com


