
Wetenschap
Nieuwe genomische instrumenten voor drie moderne katoenvariëteiten zouden toekomstige veredelingsinspanningen kunnen sturen
We leven in een steeds veranderende en groeiende wereld. Veranderende klimaten, opkomende plagen en andere omgevingsstressoren zetten de marktgewassen onder druk die de wereld voeden en van brandstof voorzien. Terwijl we racen om tegemoet te komen aan de groeiende vraag naar duurzame en hoogwaardige voedsel- en vezelgewassen, komt genomics naar voren als een krachtig hulpmiddel in de strijd. Door de genetische codes van planten te begrijpen, kunnen onderzoekers en veredelaars gewassen ontwikkelen met hogere opbrengsten, verbeterde weerstand tegen plagen en ziekten en een groter aanpassingsvermogen aan milieu-uitdagingen.
Genoom-geïnformeerde veredeling komt vooral ten goede aan gewassen met bestaande hoogwaardige genomische hulpbronnen, zoals rijst en tarwe. Gewassen met minder volwassen genomische hulpbronnen moeten echter blijven vertrouwen op traditionele veredelingsmethoden, die soms te lijden hebben onder een gebrek aan genomische diversiteit binnen de voortplantingspopulaties.
Katoen, wereldwijd een essentieel marktgewas, ontbeert robuuste genomische hulpbronnen. De katoenindustrie is een big business, met een mondiale economische impact van 600 miljard dollar en werkgelegenheid voor meer dan 250 miljoen mensen. Succesvolle katoenproductie is afhankelijk van katoenvariëteiten met wenselijke eigenschappen zoals hoge opbrengst, goede vezelkwaliteit, resistentie tegen plagen en ziekten, en droogtetolerantie.
"Katoenveredelaars hebben de vezelopbrengst en -kwaliteit door de jaren heen verbeterd met behulp van traditionele kweekmethoden", zegt Jeremy Schmutz, mededirecteur van het HudsonAlpha Genome Sequencing Center, die al meer dan tien jaar aan katoengenomics werkt. "Het bereiken van aanvullende verbeteringen kan voor hen moeilijk zijn vanwege het gebrek aan genetische variatie binnen het moderne gedomesticeerde katoen. Het creëren van nieuwe genomische instrumenten voor de industrie zal de katoenverbeteringen naar een hoger niveau tillen."
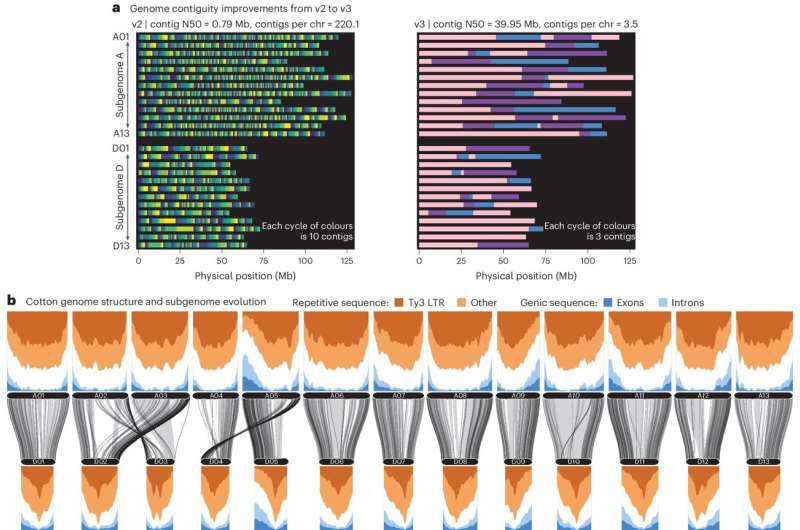
Wetenschappers van het HudsonAlpha Institute for Biotechnology Genome Sequencing Center (GSC) en andere medewerkers wilden hoogwaardige genoomsequenties creëren voor drie belangrijke katoenvariëteiten, en daarmee de noodzakelijke genoombronnen voor katoenveredelaars verschaffen. De resultaten zijn onlangs gepubliceerd in Nature Plants .
"Katoenonderzoek is sterk afhankelijk geweest van één referentiegenoom, 'TM1', een katoensoort die niet langer veel wordt gebruikt in fokprogramma's", zegt Avinash Sreedasyam, Ph.D., eerste auteur van het manuscript. "Om ervoor te zorgen dat moleculaire veredeling de katoenindustrie ten goede komt, moeten er veel, gevarieerde genomen bestaan om de diversiteit van katoenvariëteiten te vertegenwoordigen. Deze studie genereerde referentiegenomen van hoge kwaliteit voor drie moderne katoencultivars in het hoogland en actualiseerde de 'TM-1'-katoengenetica. standaardreferentie."
De eerste analyse van de nieuwe referentiegenomen leverde belangrijke informatie op over de vezelkwaliteit. De zeer nauwkeurige en complete genoomassemblages werden gebruikt om genetisch materiaal van Pima-katoen (bekend om zijn superieure vezelkwaliteit) binnen moderne katoenvariëteiten te identificeren. Kleine segmenten van elk genoom werden vergeleken met zowel Pima als het referentiekatoengenoom.
Segmenten die beter bij Pima leken dan het referentiekatoen, werden geclassificeerd als potentiële introgressies, wat suggereert dat Pima-DNA was opgenomen in de genetische samenstelling van het moderne katoen. Kennis van deze Pima-introgressies zal fokkers helpen om op efficiënte wijze nakomelingen te selecteren met deze aan vezelkwaliteit gekoppelde genetische markers in hun fokprogramma's.
"Door gebruik te maken van relatief goedkope low-pass sequencing naast deze genomen kunnen fokkers snel nakomelingen selecteren", zegt Sreedasyam. "Dit zal niet alleen tijd besparen, maar ook de kosten verlagen die gepaard gaan met traditionele vezelfenotypering, een moeizaam proces dat gewoonlijk honderden tot duizenden monsters per kweekcyclus vereist."
Deze bevindingen benadrukken het belang van het gebruik van gedetailleerde genoomassemblages om genetische variaties bloot te leggen die katoenveredelingsprogramma's kunnen verbeteren. Hoe meer deze nieuwe, hoogwaardige genomen worden gebruikt voor vergelijkende studies, hoe meer informatie over economisch belangrijke katoeneigenschappen naar voren zal komen. De genomische bronnen die in dit onderzoek worden beschreven, vormen een waardevolle aanvulling op de gereedschapskist voor katoenveredeling en zullen daar nog jaren de vruchten van plukken.
Medewerkers aan dit project zijn onder meer Don C. Jones, Cotton Incorporated, NC; Peng W. Chee, Universiteit van Georgia, Tifton, GA; Warwick N. Stiller, CSIRO, Cotton Research Unit, Australië; en Fred Bourland, Universiteit van Arkansas, Keizer, AR.