
Wetenschap
Minimalistische algoritmen voor machine learning analyseren afbeeldingen van zeer weinig gegevens
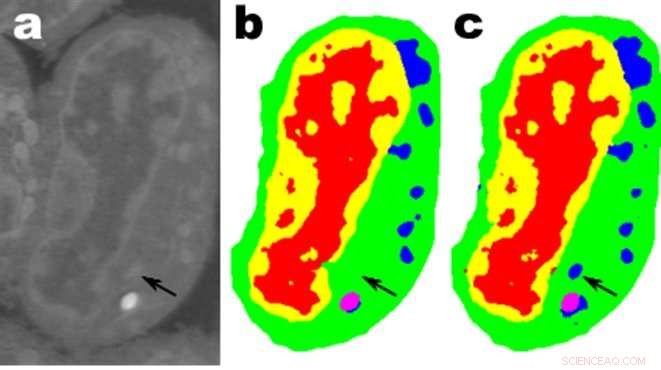
Afbeeldingen van een plakje lymfblastoïde cellen van de muis; A. zijn de ruwe gegevens, b is de overeenkomstige handmatige segmentatie en c is de uitvoer van een MS-D-netwerk met 100 lagen. Credit:gegevens van A. Ekman en C. Larabell, Nationaal centrum voor röntgentomografie.
Wiskundigen van het Lawrence Berkeley National Laboratory (Berkeley Lab) van het Department of Energy hebben een nieuwe benadering van machine learning ontwikkeld, gericht op experimentele beeldgegevens. In plaats van te vertrouwen op de tien- of honderdduizenden afbeeldingen die worden gebruikt door typische machine learning-methoden, deze nieuwe aanpak "leert" veel sneller en vereist veel minder afbeeldingen.
Daniël Pelt en James Sethian van Berkeley Lab's Center for Advanced Mathematics for Energy Research Applications (CAMERA) hebben het gebruikelijke machine learning-perspectief op zijn kop gezet door het ontwikkelen van wat zij een "Mixed-Scale Dense Convolution Neural Network (MS-D)" noemen dat vereist veel minder parameters dan traditionele methoden, convergeert snel, en heeft het vermogen om te "leren" van een opmerkelijk kleine trainingsset. Hun aanpak wordt al gebruikt om biologische structuur uit celbeelden te extraheren, en staat klaar om een belangrijk nieuw computerhulpmiddel te bieden om gegevens over een breed scala aan onderzoeksgebieden te analyseren.
Omdat experimentele faciliteiten bij hogere snelheden beelden met een hogere resolutie genereren, wetenschappers kunnen moeite hebben om de resulterende gegevens te beheren en te analyseren, wat vaak minutieus met de hand wordt gedaan. In 2014, Sethian richtte CAMERA op in Berkeley Lab als een geïntegreerd, interdisciplinair centrum voor het ontwikkelen en leveren van fundamentele nieuwe wiskunde die nodig is om te profiteren van experimenteel onderzoek bij gebruikersfaciliteiten van het DOE Office of Science. CAMERA is onderdeel van de Computational Research Division van het lab.
"In veel wetenschappelijke toepassingen, er is enorm veel handwerk nodig om afbeeldingen te annoteren en te labelen - het kan weken duren om een handvol zorgvuldig afgebakende afbeeldingen te produceren, " zei Sethian, die ook een wiskundeprofessor is aan de Universiteit van Californië, Berkeley. "Ons doel was om een techniek te ontwikkelen die leert van een zeer kleine dataset."
Details van het algoritme werden op 26 december gepubliceerd, 2017 in een krant in de Proceedings van de National Academy of Sciences .
"De doorbraak vloeide voort uit het besef dat de gebruikelijke schaalvergroting en schaalvergroting die functies op verschillende beeldschalen vastlegt, zou kunnen worden vervangen door wiskundige convoluties die meerdere schalen binnen een enkele laag verwerken, " zei Pelt, die ook lid is van de Computational Imaging Group van het Centrum Wiskunde &Informatica, het nationale onderzoeksinstituut voor wiskunde en informatica in Nederland.
Om het algoritme toegankelijk te maken voor een brede groep onderzoekers, een Berkeley-team onder leiding van Olivia Jain en Simon Mo bouwde een webportaal "Segmenting Labeled Image Data Engine (SlideCAM)" als onderdeel van de CAMERA-suite met tools voor DOE-experimentele faciliteiten.

Tomografische beelden van een vezelversterkte minicomposiet, gereconstrueerd met 1024 projecties (a) en 120 projecties (b). Bij (c), de output van een MS-D netwerk met afbeelding (b) als input wordt getoond. Een klein gebied aangegeven door een rood vierkant wordt vergroot weergegeven in de rechterbenedenhoek van elke afbeelding. Credit:Daniël Pelt en James Sethian, Berkeley Lab
Een veelbelovende toepassing is het begrijpen van de interne structuur van biologische cellen en een project waarbij de MS-D-methode van Pelt en Sethian alleen gegevens van zeven cellen nodig had om de celstructuur te bepalen.
"In ons laboratorium we werken eraan om te begrijpen hoe celstructuur en morfologie het celgedrag beïnvloeden of controleren. We besteden talloze uren aan het met de hand segmenteren van cellen om structuur te extraheren, en identificeren, bijvoorbeeld, verschillen tussen gezonde versus zieke cellen, " zei Carolyn Larabell, Directeur van het National Center for X-ray Tomography en professor aan de University of California San Francisco School of Medicine. "Deze nieuwe benadering heeft het potentieel om ons vermogen om ziekte te begrijpen radicaal te veranderen, en is een belangrijk hulpmiddel in ons nieuwe door Chan-Zuckerberg gesponsorde project om een menselijke celatlas op te zetten, een wereldwijde samenwerking om alle cellen in een gezond menselijk lichaam in kaart te brengen en te karakteriseren."
Meer wetenschap halen uit minder gegevens
Beelden zijn overal. Smartphones en sensoren hebben een schat aan foto's opgeleverd, veel getagd met relevante informatie die de inhoud identificeert. Met behulp van deze enorme database van afbeeldingen met kruisverwijzingen, convolutionele neurale netwerken en andere machine learning-methoden hebben een revolutie teweeggebracht in ons vermogen om snel natuurlijke afbeeldingen te identificeren die eruitzien als eerder gezien en gecatalogiseerd.
Deze methoden "leren" door een verbluffend grote reeks verborgen interne parameters af te stemmen, geleid door miljoenen getagde afbeeldingen, en waarvoor grote hoeveelheden supercomputertijd nodig zijn. Maar wat als je niet zoveel getagde afbeeldingen hebt? Op veel gebieden, zo'n database is een onbereikbare luxe. Biologen nemen celbeelden op en schetsen nauwgezet de grenzen en structuur met de hand:het is niet ongebruikelijk dat één persoon wekenlang bezig is met het bedenken van één volledig driedimensionaal beeld. Materiaalwetenschappers gebruiken tomografische reconstructie om in rotsen en materialen te kijken, en dan de mouwen opstropen om verschillende regio's te labelen, het identificeren van scheuren, breuken, en holtes met de hand. Contrasten tussen verschillende maar belangrijke structuren zijn vaak erg klein en "ruis" in de gegevens kan kenmerken maskeren en de beste algoritmen (en mensen) verwarren.
Deze kostbare, met de hand samengestelde afbeeldingen zijn lang niet genoeg voor traditionele methoden voor machinaal leren. Om deze uitdaging aan te gaan, wiskundigen bij CAMERA vielen het probleem van machine learning aan op basis van zeer beperkte hoeveelheden gegevens. Proberen te doen "meer met minder, " hun doel was om erachter te komen hoe ze een efficiënte set wiskundige "operators" konden bouwen die het aantal parameters aanzienlijk konden verminderen. Deze wiskundige operatoren zouden natuurlijk belangrijke beperkingen kunnen opnemen om te helpen bij identificatie, bijvoorbeeld door eisen op te nemen aan wetenschappelijk aannemelijke vormen en patronen.
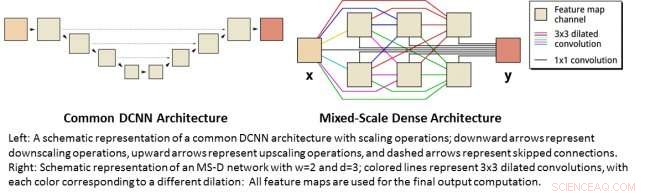
Links:een schematische weergave van een gemeenschappelijke DCNN-architectuur met schaalbewerkingen; neerwaartse pijlen vertegenwoordigen schaalverkleining, opwaartse pijlen vertegenwoordigen opschalingsbewerkingen en gestippelde pijlen vertegenwoordigen overgeslagen verbindingen. Rechts:Schematische weergave van een MS-D-netwerk met w=2 en d=3; gekleurde lijnen vertegenwoordigen 3x3 verwijde windingen, waarbij elke kleur overeenkomt met een andere dilatatie:alle functiekaarten worden gebruikt voor de uiteindelijke uitvoerberekening. Credit:Daniël Pelt en James Sethian, Berkeley Lab
Neurale netwerken op gemengde schaal met dichte convolutie
Veel toepassingen van machine learning voor beeldvormingsproblemen maken gebruik van diepe convolutionele neurale netwerken (DCNN's), waarbij het invoerbeeld en tussenbeelden zijn geconvolueerd in een groot aantal opeenvolgende lagen, waardoor het netwerk zeer niet-lineaire functies kan leren. Om nauwkeurige resultaten te verkrijgen voor moeilijke beeldverwerkingsproblemen, DCNN's zijn doorgaans afhankelijk van combinaties van aanvullende bewerkingen en verbindingen, waaronder:bijvoorbeeld, downscaling- en upscaling-bewerkingen om functies op verschillende beeldschalen vast te leggen. Om diepere en krachtigere netwerken te trainen, er zijn vaak extra laagtypen en verbindingen nodig. Eindelijk, DCNN's gebruiken doorgaans een groot aantal tussenliggende beelden en trainbare parameters, vaak meer dan 100 miljoen, om resultaten te boeken voor moeilijke problemen.
In plaats daarvan, de nieuwe "Mixed-Scale Dense" netwerkarchitectuur vermijdt veel van deze complicaties en berekent verwijde windingen als vervanging voor schaaloperaties om kenmerken op verschillende ruimtelijke bereiken vast te leggen, het gebruik van meerdere schalen binnen een enkele laag, en dicht alle tussenliggende beelden met elkaar verbinden. Het nieuwe algoritme behaalt nauwkeurige resultaten met weinig tussenliggende beelden en parameters, het elimineren van zowel de noodzaak om hyperparameters af te stemmen als extra lagen of verbindingen om training mogelijk te maken.
Wetenschap met een hoge resolutie halen uit gegevens met een lage resolutie
Een andere uitdaging is het produceren van afbeeldingen met een hoge resolutie van invoer met een lage resolutie. Zoals iedereen die heeft geprobeerd een kleine foto te vergroten en ontdekte dat het alleen maar erger wordt naarmate het groter wordt, dit klinkt bijna onmogelijk. Maar een kleine set trainingsbeelden die zijn verwerkt met een Mixed-Scale Dense-netwerk kan echte vooruitgang opleveren. Als voorbeeld, stel je voor dat je tomografische reconstructies van een vezelversterkt mini-composietmateriaal probeert ongedaan te maken. In een experiment beschreven in de krant, afbeeldingen werden gereconstrueerd met behulp van 1, 024 verwierf röntgenprojecties om beelden met relatief weinig ruis te verkrijgen. Vervolgens werden luidruchtige beelden van hetzelfde object verkregen door te reconstrueren met 128 projecties. Trainingsinputs waren beelden met ruis, met bijbehorende geruisloze beelden die tijdens de training als doeluitvoer worden gebruikt. Het getrainde netwerk was vervolgens in staat om ruisige invoergegevens effectief op te nemen en afbeeldingen met een hogere resolutie te reconstrueren.
Nieuwe toepassingen
Pelt en Sethian benaderen een groot aantal nieuwe gebieden, zoals snelle real-time analyse van beelden die uit synchrotron-lichtbronnen komen en reconstructieproblemen bij biologische reconstructie zoals voor cellen en hersenkartering.
"Deze nieuwe benaderingen zijn echt opwindend, omdat ze de toepassing van machine learning op een veel grotere verscheidenheid aan beeldvormingsproblemen mogelijk maken dan momenteel mogelijk is, "Zei Pelt. "Door het aantal vereiste trainingsbeelden te verminderen en de grootte van de beelden die kunnen worden verwerkt, te vergroten, de nieuwe architectuur kan worden gebruikt om belangrijke vragen op veel onderzoeksgebieden te beantwoorden."
Hoofdlijnen
- Ambtenaren:walvissen, na een dodelijk jaar, zou kunnen uitsterven
- Hoe deelbaar door uit te drukken in Excel
- Voorbeelden van archaebacteriën met hun wetenschappelijke naam en classificatie
- Hoe een hart voor een wetenschapsproject te bouwen
- Horror als Noorse goederentreinen meer dan 100 rendieren neermaaien
- Je wandeling is zo duidelijk dat het diepe persoonlijkheidskenmerken kan onthullen
- "Recessive Allele: What is it?", 3, [[& Waarom gebeurt het? (met eigenschappenkaart)
- Europese zeebaars vertoont chronische verslechtering na blootstelling aan ruwe olie
- Hoeveel onontdekte wezens zijn er in de oceaan?
- Octrooiaanvraag kijkt naar slimme deurbelsnuiven, ACLU reageert

- Tesla bevestigt crimineel onderzoek naar Musk over privé gaan

- Hongaarse luchtvaartmaatschappij Wizz Air schrapt 1, 000 banen

- Inkjet-zonnepanelen klaar om een revolutie teweeg te brengen in groene energie

- banken, Bitcoin, obligatiefondsen:waar is uw geld veilig in een tijdperk van cyberaanvallen?

 Definitie van celoppervlakte-eiwitten
Definitie van celoppervlakte-eiwitten Waarom gebruiken mensen nog steeds faxmachines?
Waarom gebruiken mensen nog steeds faxmachines? Wetenschappers ontwikkelen proces om brandstof van hogere kwaliteit te produceren uit bioafval
Wetenschappers ontwikkelen proces om brandstof van hogere kwaliteit te produceren uit bioafval De politiek van pandemieën:waarom sommige landen beter reageren dan andere
De politiek van pandemieën:waarom sommige landen beter reageren dan andere Nieuwe aanval kan captcha's voor websitebeveiliging overbodig maken
Nieuwe aanval kan captcha's voor websitebeveiliging overbodig maken Onderzoekers ontwikkelen een eenvoudige techniek om structuren op atomaire schaal te visualiseren
Onderzoekers ontwikkelen een eenvoudige techniek om structuren op atomaire schaal te visualiseren Welk dier is de aaseter in een voedselketen?
Welk dier is de aaseter in een voedselketen?  Vijf jaar durende studie onthult hoeveel koolstof de ecologische hulpbronnen van China vastleggen
Vijf jaar durende studie onthult hoeveel koolstof de ecologische hulpbronnen van China vastleggen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com


