Wetenschap
Enzymatische photocaging voor de studie van genregulatie door middel van DNA-methylatie
Krediet:Wiley
De toevoeging en verwijdering van methylgroepen op DNA speelt een belangrijke rol bij genregulatie. Om deze mechanismen nauwkeuriger te bestuderen, een Duits team heeft een nieuwe methode ontwikkeld waarmee specifieke methyleringsplaatsen kunnen worden geblokkeerd en vervolgens op een nauwkeurig tijdstip kunnen worden gedeblokkeerd door middel van bestraling met licht (photocaging). Zoals gerapporteerd in het journaal Angewandte Chemie , de benodigde regent wordt enzymatisch geproduceerd, ter plaatse.
Hoewel ze er heel anders uitzien en totaal verschillende functies hebben, alle cellen in ons lichaam hebben identiek DNA. Echter, ze gebruiken niet dezelfde genen. Bepaalde genen zijn ingeschakeld en andere uitgeschakeld, afhankelijk van het type cel en het moment in de tijd. De "schakelaars" zijn chemische veranderingen in de bouwstenen van het DNA. Deze veranderingen worden epigenetische modificaties genoemd. Een belangrijk regulatiemechanisme is methylering en demethylering, wat betekent de aanhechting en verwijdering van een methylgroep (-CH 3 ). De methylatiepatronen van kankercellen, bijvoorbeeld, verschillen van gezonde cellen. Tijdens een methylering, enzymen die bekend staan als methyltransferasen (MTasen) dragen een methylgroep over van S-adenosyl- L -methionine (AdoMet) aan het doelmolecuul.
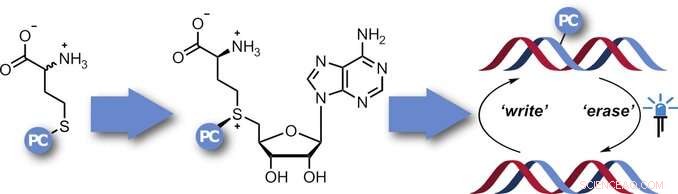
Om het doel en de functie van deze regulatie nader te bestuderen en methyleringspatronen te bepalen, het zou nuttig zijn om "tools" te hebben om specifiek methylering op gerichte locaties te remmen en vervolgens de remming op een bepaald tijdstip op te heffen. Hiertoe, een team onder leiding van Andrea Rentmeister koos voor een methode die bekend staat als photocaging. Bij deze methode, een "fotokooi" is een molecuul dat uit elkaar valt bij bestraling, zoals een 2-nitrobenzylgroep. De kooi blokkeert eerst de doellocatie, dan werkt gerichte bestraling met licht als 'schakelaar' om de blokkade op te heffen.
Het idee was om AdoMet-analogen uit te rusten met een fotokooi die vervolgens wordt overgebracht naar de methyleringsplaatsen. Echter, AdoMet-analogen ontleden in waterige oplossingen en kunnen geen cellen binnendringen. Daarom, het team van de Universiteit van Münster wilde ze in situ produceren. In het lichaam, AdoMet wordt geproduceerd uit het aminozuur methionine door de werking van het enzym, methionine-adenosyltransferase (MAT). Synthese van de AdoMet-analogen vereist methionine met een aangehechte nitrobenzyl-fotokooi en een MAT die een dergelijk gewijzigd substraat kan gebruiken. Beginnend met een MAT-enzym uit een eencellig organisme (Cryptosporidium hominis), de onderzoekers waren in staat om specifieke aminozuren in het enzym zorgvuldig te veranderen om de grootte van de hydrofobe bindingsholte te vergroten, zodat het de nitrobenzylgroep kon bevatten. Een kristalstructuuranalyse toonde aan dat de ADoMet-analoog is gebonden in de holte van deze fotokooiende MAT (PC-MAT). Op basis van deze informatie, het team produceerde ook een tweede PC-MAT op basis van een thermostabiel MAT-enzym van de archaeon Methanocaldococcus jannaschii.
Beide PC-MAT's zijn compatibel met DNA- en RNA-MTasen en maakten het mogelijk om fotokooien te hechten aan alle natuurlijke methyleringsplaatsen van een plasmide-DNA. Bestraling met licht verwijderde de blokkade.
 Nieuwe tool analyseert ziekte- en medicijneffecten met ongekende nauwkeurigheid en consistentie
Nieuwe tool analyseert ziekte- en medicijneffecten met ongekende nauwkeurigheid en consistentie Splits het op:inzicht in de vorming van chemische bijproducten tijdens waterbehandeling
Splits het op:inzicht in de vorming van chemische bijproducten tijdens waterbehandeling Een nieuwe methode om de efficiëntie van kristalhalfgeleiders te kwantificeren
Een nieuwe methode om de efficiëntie van kristalhalfgeleiders te kwantificeren Draagbaar apparaat verlicht de weg naar betere detectie van door voedsel overgedragen ziekten
Draagbaar apparaat verlicht de weg naar betere detectie van door voedsel overgedragen ziekten Titaanatoom dat op twee plaatsen tegelijk in kristal bestaat en de oorzaak is van een ongewoon fenomeen
Titaanatoom dat op twee plaatsen tegelijk in kristal bestaat en de oorzaak is van een ongewoon fenomeen
 Openbaarmaking van klimaatgerelateerde financiële risico's niet voldoende om actie te stimuleren
Openbaarmaking van klimaatgerelateerde financiële risico's niet voldoende om actie te stimuleren Hoe kennis te integreren voor het beheer van toekomstige klimaatextremen
Hoe kennis te integreren voor het beheer van toekomstige klimaatextremen Droogte in Californië neemt af door stormen die sneeuw en regen dumpen
Droogte in Californië neemt af door stormen die sneeuw en regen dumpen Stijgende oceaantemperatuur bedreigt ijskap Groenlands
Stijgende oceaantemperatuur bedreigt ijskap Groenlands Om tropische bossen te behouden, empowerment van lokale gemeenschappen
Om tropische bossen te behouden, empowerment van lokale gemeenschappen
Hoofdlijnen
- Nieuwe studie laat zien hoe vogels samenwerken om samen te zingen
- Wat is TA Cloning?
- De voordelen van over jezelf praten in de derde persoon
- Het is goed om zeldzaam te zijn, voor sommige soorten
- Recordaantal walvissen geteld in Patagonië in Argentinië
- De vliegsnelheid van vogels is complexer dan eerder werd gedacht
- Wat gebeurt er als er sprake is van mitose?
- Waarom krijgen demonen de schuld van slaapverlamming?
- Fenotype: definitie, types, voorbeelden
 Roemenië zou de Russische greep op de energie van Europa kunnen uitdagen
Roemenië zou de Russische greep op de energie van Europa kunnen uitdagen Big Techs grote winstweek overschaduwd door politiek verzet
Big Techs grote winstweek overschaduwd door politiek verzet Verbetering van de foto-elektrische efficiëntie
Verbetering van de foto-elektrische efficiëntie Waarom de Middellandse Zee een hotspot voor klimaatverandering is
Waarom de Middellandse Zee een hotspot voor klimaatverandering is Materialen op bestelling:ingenieurs richten zich op de nano om sterke, lichtgewicht materialen
Materialen op bestelling:ingenieurs richten zich op de nano om sterke, lichtgewicht materialen Dit bazige bureau bepaalt wanneer je opstaat en gaat zitten
Dit bazige bureau bepaalt wanneer je opstaat en gaat zitten  Video-app Zoom schiet naar roem, met wat haperingen, te midden van pandemie
Video-app Zoom schiet naar roem, met wat haperingen, te midden van pandemie Gebruik maken van de slagvastheid van 's werelds hardste beton voor rampenpreventie
Gebruik maken van de slagvastheid van 's werelds hardste beton voor rampenpreventie
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Swedish | German | Dutch | Danish | Norway | Portuguese |

-
Wetenschap © https://nl.scienceaq.com