
Wetenschap
Onderzoeksgroep gebruikt supercomputing om zich te richten op de meest veelbelovende kandidaat-geneesmiddelen uit een enorm aantal mogelijkheden
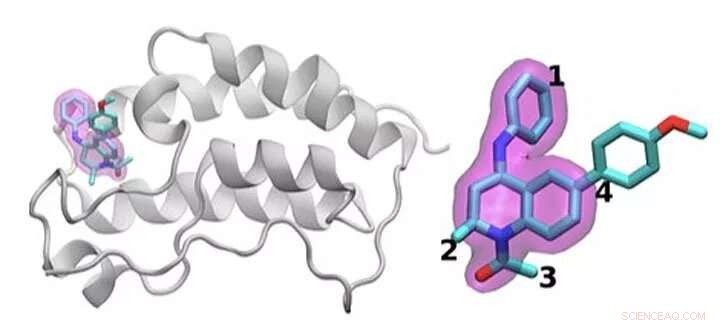
Een schema van het BRD4-eiwit gebonden aan een van de 16 geneesmiddelen op basis van dezelfde tetrahydrochinoline-steiger (gemarkeerd in magenta). Regio's die chemisch gemodificeerd zijn tussen de geneesmiddelen die in deze studie zijn onderzocht, zijn gelabeld 1 tot 4. Gewoonlijk worden er wordt slechts een kleine verandering aangebracht in de chemische structuur van het ene medicijn naar het andere. Deze conservatieve benadering stelt onderzoekers in staat om te onderzoeken waarom het ene medicijn effectief is en het andere niet. Krediet:Brookhaven National Laboratory
Het identificeren van de optimale medicamenteuze behandeling is als het raken van een bewegend doelwit. Om ziekte te stoppen, medicijnen met kleine moleculen binden stevig aan een belangrijk eiwit, het blokkeren van de effecten ervan in het lichaam. Zelfs goedgekeurde medicijnen werken meestal niet bij alle patiënten. En na verloop van tijd, infectieuze agentia of kankercellen kunnen muteren, een eenmaal effectief medicijn onbruikbaar maken.
Aan al deze problemen ligt een fysiek kernprobleem ten grondslag:het optimaliseren van de interactie tussen het medicijnmolecuul en zijn eiwitdoelwit. De variaties in kandidaat-geneesmiddelen, het mutatiebereik in eiwitten en de algehele complexiteit van deze fysieke interacties maken dit werk moeilijk.
Shantenu Jha van het Brookhaven National Laboratory en de Rutgers University van het Department of Energy (DOE) leidt een team dat probeert de rekenmethoden te stroomlijnen, zodat supercomputers een deel van deze immense werklast op zich kunnen nemen. Ze hebben een nieuwe strategie gevonden om één onderdeel aan te pakken:differentiëren hoe kandidaat-geneesmiddelen interageren en binden met een gericht eiwit.
Voor hun werk, Jha en zijn collega's wonnen vorig jaar de IEEE International Scalable Computing Challenge (SCALE) award, die schaalbare computeroplossingen herkent voor echte wetenschappelijke en technische problemen.
Om een nieuw medicijn te ontwerpen, een farmaceutisch bedrijf zou kunnen beginnen met een bibliotheek van miljoenen kandidaat-moleculen die ze beperken tot de duizenden die enige initiële binding aan een doeleiwit vertonen. Het verfijnen van deze opties tot een bruikbaar medicijn dat op mensen kan worden getest, kan uitgebreide experimenten omvatten om atoomgroepen op belangrijke locaties op het molecuul toe te voegen of af te trekken en te testen hoe elk van deze veranderingen de interactie tussen het kleine molecuul en het eiwit verandert.
Simulaties kunnen hierbij helpen. groter, snellere supercomputers en steeds geavanceerdere algoritmen kunnen realistische fysica bevatten en de bindingsenergieën tussen verschillende kleine moleculen en eiwitten berekenen. Dergelijke methoden kunnen aanzienlijke rekenkracht verbruiken, echter, om de benodigde nauwkeurigheid te bereiken. Ook industrie-nuttige simulaties moeten snelle antwoorden geven. Door het getouwtrek tussen nauwkeurigheid en snelheid, onderzoekers zijn voortdurend aan het innoveren, het ontwikkelen van efficiëntere algoritmen en het verbeteren van de prestaties, zegt Jha.
Dit probleem vereist ook een ander beheer van computerbronnen dan voor veel andere grootschalige problemen. In plaats van één enkele simulatie te ontwerpen die kan worden geschaald om een hele supercomputer te gebruiken, onderzoekers draaien tegelijkertijd veel kleinere modellen die elkaar en het traject van toekomstige berekeningen vormen, een strategie die bekend staat als ensemble-based computing, of complexe workflows.
"Zie dit als proberen een heel groot open landschap te verkennen om te proberen te vinden waar je misschien de beste kandidaat-medicijn kunt krijgen, " zegt Jha. In het verleden, onderzoekers hebben computers gevraagd om door dit landschap te navigeren door willekeurige statistische keuzes te maken. Op een beslissingsmoment, de helft van de berekeningen zou één pad kunnen volgen, de andere helft een andere.
Jha en zijn team zoeken naar manieren om deze simulaties te helpen leren van het landschap. Realtime gegevens opnemen en vervolgens delen is niet eenvoudig, Jha zegt, "en dat was wat een deel van de technologische innovatie op grote schaal vereiste." Hij en zijn team uit Rutgers werken aan dit werk samen met de groep van Peter Coveney aan het University College London.
Om dit idee te testen, ze hebben algoritmen gebruikt die bindingsaffiniteit voorspellen en hebben gestroomlijnde versies geïntroduceerd in een HTBAC-framework, voor bindingsaffiniteitscalculator met hoge doorvoer. Een dergelijke rekenmachine, bekend als ESMACS, helpt hen moleculen te elimineren die slecht binden aan een doeleiwit. De andere, STROPDAS, is nauwkeuriger maar beperkter in omvang en vereist 2,5 keer meer rekenkracht. Niettemin, het kan de onderzoekers helpen een veelbelovende interactie tussen een medicijn en een eiwit te optimaliseren. Het HTBAC-raamwerk helpt hen deze algoritmen efficiënt te implementeren, het intensievere algoritme opslaan voor situaties waar het nodig is.
Het team demonstreerde het idee door 16 kandidaat-geneesmiddelen uit een moleculebibliotheek bij GlaxoSmithKline (GSK) met hun doelwit te onderzoeken, BRD4-BD1 - een eiwit dat belangrijk is bij borstkanker en ontstekingsziekten. De kandidaat-geneesmiddelen hadden dezelfde kernstructuur, maar verschilden op vier verschillende gebieden rond de randen van het molecuul.
In deze eerste studie voerde het team duizenden processen tegelijkertijd uit op 32, 000 kernen op Blue Waters, een supercomputer van de National Science Foundation (NSF) aan de Universiteit van Illinois in Urbana-Champaign. Ze hebben soortgelijke berekeningen uitgevoerd op Titan, de Cray XK7-supercomputer bij de Oak Ridge Leadership Computing Facility, een DOE Office of Science gebruikersfaciliteit. Het team maakte met succes onderscheid tussen de binding van deze 16 kandidaat-geneesmiddelen, de grootste dergelijke simulatie tot nu toe. "We hebben niet alleen een ongekende schaal bereikt, Jha zegt. "Onze aanpak toont het vermogen om te differentiëren."
Ze wonnen hun SCALE-award voor deze eerste proof of concept. De uitdaging nu, Jha zegt, zorgt ervoor dat het niet alleen werkt voor BRD4, maar ook voor andere combinaties van medicijnmoleculen en eiwitdoelen.
Als de onderzoekers hun aanpak kunnen blijven uitbreiden, dergelijke technieken zouden uiteindelijk de ontdekking van geneesmiddelen kunnen versnellen en gepersonaliseerde geneeskunde mogelijk maken. Maar om meer realistische problemen te onderzoeken, ze hebben meer rekenkracht nodig. "We zitten midden in deze spanning tussen een zeer grote chemische ruimte die we, in principe, moet verkennen, en, helaas beperkte computerbronnen." zegt Jha.
Zelfs als supercomputing zich uitbreidt naar de exaschaal, computationele wetenschappers kunnen de leemte meer dan opvullen door meer realistische fysica aan hun modellen toe te voegen. Voor de nabije toekomst, onderzoekers zullen vindingrijk moeten zijn om deze berekeningen op te schalen. Noodzaak is de moeder van innovatie, Jha zegt, juist omdat de moleculaire wetenschap niet over de ideale hoeveelheid rekenkracht zal beschikken om simulaties uit te voeren.
Maar exascale computing kan hen helpen dichter bij hun doelen te komen. Naast het werken met University College London en GSK, Jha en zijn collega's werken samen met Rick Stevens van Argonne National Laboratory en het CANcer Distributed Learning Environment (CANDLE) team. Dit co-designproject binnen het DOE's Exascale Computing Project bouwt diepe neurale netwerken en algemene machine learning-technieken om kanker te bestuderen. De algoritmen en software binnen HTBAC zouden de focus van CANDLE op die benaderingen kunnen aanvullen.
Deze bredere samenwerking tussen Jha's groep, het CANDLE-team en John Chodera's Lab in het Memorial Sloan-Kettering Cancer Center hebben geleid tot het project Integrated and Scalable Prediction of Resistance (INSPIRE). Dit team heeft al simulaties uitgevoerd op DOE's Summit-supercomputer in het Oak Ridge National Laboratory. Het zal binnenkort doorgaan met dit werk aan Frontera - de leiderschapsmachine van de NSF aan de Universiteit van Texas in Austin's Texas Advanced Computing Center.
"We snakken naar meer vooruitgang en grotere methodologische verbeteringen, " zegt Jha. "We zouden graag willen zien hoe deze mooie complementaire benaderingen integrerend kunnen werken in de richting van deze grootse visie."
 Azië Grote Rivieren:Klimaatcrisis, vervuiling brengt miljarden in gevaar
Azië Grote Rivieren:Klimaatcrisis, vervuiling brengt miljarden in gevaar Indonesië verhoogt alarmniveau voor vulkaan Bali
Indonesië verhoogt alarmniveau voor vulkaan Bali Mierenaanpassing
Mierenaanpassing  Het bepalen van de timing van de evolutie van methanogeen
Het bepalen van de timing van de evolutie van methanogeen 11% van de vernietigde vochtige tropische bossen kan worden hersteld om het klimaat te verbeteren, omgeving
11% van de vernietigde vochtige tropische bossen kan worden hersteld om het klimaat te verbeteren, omgeving
Hoofdlijnen
- Veel dierenartsen praten niet graag over dikke katten
- Plasmamembraan: definitie, structuur en functie (met diagram)
- Natuurlijke selectie: definitie, Darwins-theorie, voorbeelden en feiten
- Klimaatverandering maakt baardagamen mogelijk minder intelligent
- Onderzoekers onthullen een faalveilige structuur van een enzym dat verband houdt met de ziekte van Alzheimer, kanker
- Enzymen: wat is het? & Hoe werkt het?
- Hoe hoge bomen suikers verplaatsen
- Gevaren van het inademen van olierook
- Onderzoekers ontwikkelen een gel voor het kweken van grote hoeveelheden neurale stamcellen
- Nieuwe methode zet methaan in aardgas bij kamertemperatuur om in methanol

- Ontdekking van natuurlijk chirale oppervlakken voor veiligere geneesmiddelen

- Spinchemie heroverwegen vanuit een kwantumperspectief
- Hoe verleng je de levensduur van smeermiddelen?

- Onderzoeksteam ontdekt gebruik van elasticiteit om microplaten op gebogen 2D-vloeistoffen te positioneren
 Hoge verwachtingen nu de nieuwe nachttrein van Oostenrijk vertrekt naar Brussel
Hoge verwachtingen nu de nieuwe nachttrein van Oostenrijk vertrekt naar Brussel Armoede en eerlijkheid zijn geen tegenstellingen
Armoede en eerlijkheid zijn geen tegenstellingen Met behulp van virtuele realiteit, Onderzoekers van het Amerikaanse leger in Orlando proberen soldaten te trainen in IED-detectie
Met behulp van virtuele realiteit, Onderzoekers van het Amerikaanse leger in Orlando proberen soldaten te trainen in IED-detectie Warm ijs in 's werelds hoogste gletsjer
Warm ijs in 's werelds hoogste gletsjer Erosie van de Himalaya geregeerd door tektonische bewegingen, de effecten van klimaatverandering op landschapsvorming beperken
Erosie van de Himalaya geregeerd door tektonische bewegingen, de effecten van klimaatverandering op landschapsvorming beperken Verschil tussen lineaire en vertakte polymeren
Verschil tussen lineaire en vertakte polymeren Beeldvorming met superresolutie onthult het mechanisme van GLUT1-clustering
Beeldvorming met superresolutie onthult het mechanisme van GLUT1-clustering Nieuwe studie onderzoekt de tijd en moeite die het kost om in elke staat te stemmen
Nieuwe studie onderzoekt de tijd en moeite die het kost om in elke staat te stemmen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

