
Wetenschap
AI-tool maakt synthetische afbeeldingen van cellen voor verbeterde microscopieanalyse
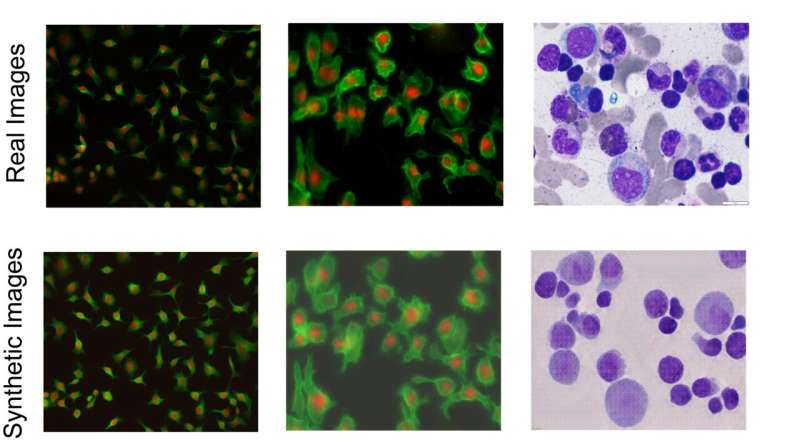
Het observeren van individuele cellen door microscopen kan een reeks belangrijke celbiologische verschijnselen aan het licht brengen die vaak een rol spelen bij ziekten bij de mens, maar het proces om afzonderlijke cellen van elkaar en hun achtergrond te onderscheiden is uiterst tijdrovend – en een taak die zeer geschikt is voor AI-hulp.
AI-modellen leren hoe ze dergelijke taken kunnen uitvoeren door een reeks gegevens te gebruiken die door mensen zijn geannoteerd, maar het proces om cellen van hun achtergrond te onderscheiden, genaamd 'single-cell segmentation', is zowel tijdrovend als moeizaam. Als gevolg hiervan is er een beperkte hoeveelheid geannoteerde gegevens die kunnen worden gebruikt in AI-trainingssets. Onderzoekers van UC Santa Cruz hebben een methode ontwikkeld om dit op te lossen door een AI-model voor het genereren van microscopiebeelden te bouwen om realistische beelden van afzonderlijke cellen te creëren, die vervolgens worden gebruikt als "synthetische gegevens" om een AI-model te trainen om de segmentatie van afzonderlijke cellen beter uit te voeren.
De nieuwe software wordt beschreven in een nieuw artikel gepubliceerd in het tijdschrift iScience . Het project werd geleid door universitair docent biomoleculaire technologie Ali Shariati en zijn afgestudeerde student Abolfazl Zargari. Het model, genaamd cGAN-Seg, is gratis beschikbaar op GitHub.
"De beelden die uit ons model komen, zijn klaar om te worden gebruikt om segmentatiemodellen te trainen", zei Shariati. "In zekere zin doen we microscopie zonder microscoop, in die zin dat we beelden kunnen genereren die heel dicht bij echte beelden van cellen liggen wat betreft de morfologische details van de enkele cel. Het mooie ervan is dat wanneer ze naar buiten komen van het model zijn ze al geannoteerd en gelabeld. De afbeeldingen vertonen veel overeenkomsten met echte afbeeldingen, waardoor we nieuwe scenario's kunnen genereren die tijdens de training niet door ons model zijn gezien.
Afbeeldingen van individuele cellen, gezien door een microscoop, kunnen wetenschappers helpen meer te leren over celgedrag en -dynamiek in de loop van de tijd, de detectie van ziekten te verbeteren en nieuwe medicijnen te vinden. Subcellulaire details zoals textuur kunnen onderzoekers helpen belangrijke vragen te beantwoorden, zoals of een cel kankerachtig is of niet.
Het handmatig vinden en labelen van de grenzen van cellen vanaf hun achtergrond is echter uiterst moeilijk, vooral in weefselmonsters waarbij er veel cellen in een beeld staan. Het kan onderzoekers meerdere dagen kosten om handmatig celsegmentatie uit te voeren op slechts 100 microscopiebeelden.
Deep learning kan dit proces versnellen, maar er is een eerste dataset van geannoteerde afbeeldingen nodig om de modellen te trainen. Er zijn minstens duizenden afbeeldingen nodig als basis om een accuraat deep learning-model te trainen. Zelfs als de onderzoekers 1000 afbeeldingen kunnen vinden en annoteren, bevatten die afbeeldingen mogelijk niet de variatie aan kenmerken die onder verschillende experimentele omstandigheden voorkomen.
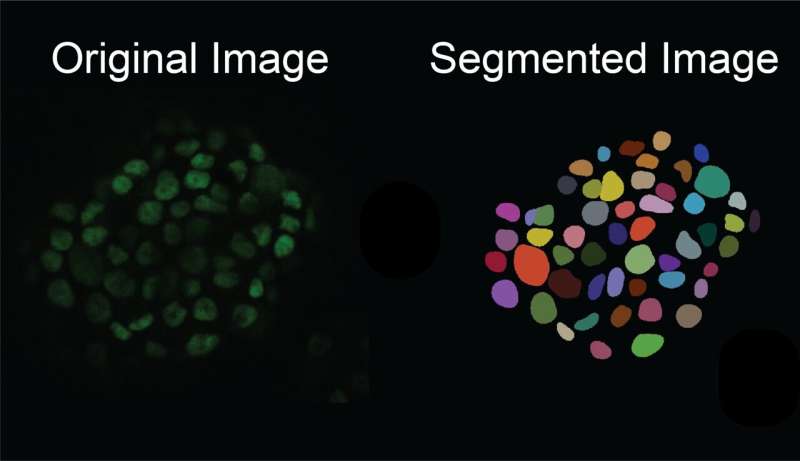
"Je wilt laten zien dat je deep learning-model werkt in verschillende monsters met verschillende celtypen en verschillende beeldkwaliteiten", zei Zargari. "Als je je model bijvoorbeeld traint met afbeeldingen van hoge kwaliteit, zal het de celafbeeldingen van lage kwaliteit niet kunnen segmenteren. We kunnen zelden zo'n goede dataset vinden op microscopiegebied."
Om dit probleem aan te pakken, creëerden de onderzoekers een beeld-naar-beeld generatief AI-model dat een beperkte reeks geannoteerde, gelabelde celafbeeldingen gebruikt en er meer genereert, waarbij meer ingewikkelde en gevarieerde subcellulaire kenmerken en structuren worden geïntroduceerd om een gevarieerde reeks ‘synthetische’ beelden te creëren. afbeeldingen. Ze kunnen met name geannoteerde afbeeldingen genereren met een hoge celdichtheid, die bijzonder moeilijk met de hand te annoteren zijn en vooral relevant zijn voor het bestuderen van weefsels. Deze techniek verwerkt en genereert afbeeldingen van verschillende celtypen, evenals verschillende beeldvormingsmodaliteiten, zoals opnames gemaakt met behulp van fluorescentie of histologische kleuring.
Zargari, die leiding gaf aan de ontwikkeling van het generatieve model, gebruikte voor het creëren van realistische beelden een veelgebruikt AI-algoritme, een 'cycle generatief adversarial network' genaamd. Het generatieve model is verbeterd met zogenaamde 'augmentatiefuncties' en een 'stijlinjectienetwerk', waarmee de generator een grote verscheidenheid aan hoogwaardige synthetische beelden kan creëren die verschillende mogelijkheden laten zien voor hoe de cellen eruit zouden kunnen zien. Voor zover de onderzoekers weten is dit de eerste keer dat stijlinjectietechnieken in deze context zijn gebruikt.
Vervolgens wordt deze diverse reeks synthetische beelden, gemaakt door de generator, gebruikt om een model te trainen om nauwkeurig celsegmentatie uit te voeren op nieuwe, echte beelden die tijdens experimenten zijn gemaakt.
"Met behulp van een beperkte dataset kunnen we een goed generatief model trainen. Met behulp van dat generatieve model kunnen we een meer diverse en grotere reeks geannoteerde, synthetische afbeeldingen genereren. Met behulp van de gegenereerde synthetische afbeeldingen kunnen we een goed segmentatiemodel trainen - dat is het hoofdidee', zei Zagari.
De onderzoekers vergeleken de resultaten van hun model met behulp van synthetische trainingsgegevens met meer traditionele methoden om AI te trainen om celsegmentatie over verschillende soorten cellen uit te voeren. Ze ontdekten dat hun model een aanzienlijk verbeterde segmentatie oplevert in vergelijking met modellen die zijn getraind met conventionele, beperkte trainingsgegevens. Dit bevestigt voor de onderzoekers dat het aanbieden van een meer diverse dataset tijdens het trainen van het segmentatiemodel de prestaties verbetert.
Door deze verbeterde segmentatiemogelijkheden zullen de onderzoekers cellen beter kunnen detecteren en de variabiliteit tussen individuele cellen kunnen bestuderen, vooral onder stamcellen. In de toekomst hopen de onderzoekers de technologie die ze hebben ontwikkeld te gebruiken om verder te gaan dan alleen stilstaande beelden en video's te genereren, die hen kunnen helpen vast te stellen welke factoren het lot van een cel vroeg in zijn leven beïnvloeden en hun toekomst kunnen voorspellen.
"We genereren synthetische beelden die ook kunnen worden omgezet in een time-lapse-film, waarin we de onzichtbare toekomst van cellen kunnen genereren", zei Shariati. "Daarmee willen we zien of we de toekomstige toestanden van een cel kunnen voorspellen, bijvoorbeeld of de cel gaat groeien, migreren, differentiëren of delen."