
Wetenschap
Zoete aardappelgenetica:een uitgebreide update van de Taizhong 6-genoomannotatie
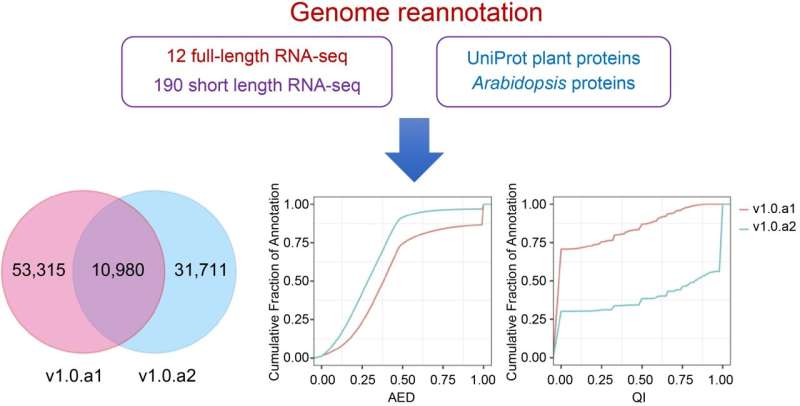
Een onderzoeksteam heeft de annotatie van het zoete aardappelgenoom "Taizhong 6" aanzienlijk verbeterd, door een uitgebreidere en gedetailleerdere versie te introduceren, v1.0.a2. Deze update maakt gebruik van 12 Nanopore-RNA-bibliotheken van volledige lengte en 190 Illumina RNA-seq-bibliotheken, resulterend in de identificatie van 360 nieuwe genen, wijziging of toevoeging van 31.771 genmodellen en verfijnde gennomenclatuur.
De verbeterde annotatie omvat gedetailleerde miRNA-expressieprofielen, wat ten goede komt aan functionele genstudies bij zoete aardappelen en het bevorderen van genomische analyses in de Convolvulaceae-familie, waardoor een cruciale bron wordt geboden voor toekomstig landbouw- en genetisch onderzoek.
Zoete aardappel (Ipomoea batatas) is een essentieel gewas in ontwikkelingslanden, dat als hexaploïde soort essentiële voedingsstoffen levert en vitamine A-tekort in Afrika bestrijdt. De eerste genoomsequentie, cultivar Taizhong 6, maakte gebruik van Illumina-sequencing, later uitgebreid met sequencing-technologieën van de derde generatie, wat leidde tot een hoogwaardige, op chromosoom geschaalde genoomassemblage. Er blijven echter uitdagingen bestaan bij het verkrijgen van nauwkeurige genoomannotaties vanwege de beperkingen van short-read sequencing.
Recentelijk hebben ontwikkelingen op het gebied van long-read sequencing, zoals Oxford Nanopore Technologies, nauwkeurigere annotaties mogelijk gemaakt en diepere inzichten in de genstructuur en alternatieve splitsing mogelijk gemaakt.
Een onderzoek gepubliceerd in Tropical Plants op 21 maart 2024, bouwt voort op deze vooruitgang met behulp van een verfijnde annotatiepijplijn die zowel Nanopore-datasets van volledige lengte als uitgebreide RNA-seq-datasets omvatte, waardoor de huidige genoomannotatie voor zoete aardappel wordt verbeterd.
In deze studie hebben onderzoekers de annotatie van het I. batata-genoom verbeterd naar versie 1.0.a2, met behulp van een alomvattende aanpak waarbij Nanopore transcriptomen van volledige lengte en Illumina RNA-seq-gegevens over verschillende ontwikkelingsstadia en weefsels van zoete aardappel worden geïntegreerd.
Hun methode maakte gebruik van BRAKER voor initiële genvoorspellingen, verrijkt met verschillende genomische hints, gevolgd door het genereren van consensusmodellen via EVidenceModeler (EVM). Opvallend is dat de bijgewerkte annotatie nu 42.751 eiwitcoderende genen bevat, waardoor het model wordt uitgebreid met 3'- en 5'-UTR's en het gemiddelde aantal exonen per gen toeneemt.
Het is veelbetekenend dat deze herziening 31.771 genmodellen heeft toegevoegd of gewijzigd, met 8.736 alternatieve splicing-isovormen, en een nieuwe gennomenclatuur heeft geïntroduceerd voor duidelijkere referentie. Deze meer gedetailleerde annotatie helpt bij nauwkeurige genomische studies en ondersteunt geavanceerde functionele genomica bij zoete aardappel.
Bovendien biedt de integratie van miRNA-gegevens en hun doelwitten nieuwe inzichten in genregulatie, vooral tijdens de verschillende ontwikkelingsstadia van opslagwortels, waardoor ons begrip van de zoete aardappelbiologie wordt vergroot en gerichte veredelingsinspanningen worden ondersteund. De uitgebreide voorspellingen van de genfunctie zijn uitgevoerd met behulp van InterProScan en eggNOG mapper, wat een rijkere annotatie oplevert die cruciaal is voor lopend onderzoek en veredelingsprogramma's gericht op het verbeteren van zoete aardappelcultivars voor de mondiale landbouw.
Volgens de hoofdonderzoeker van de studie, prof. Guopeng Zhu, "draagt onze studie bij aan een bijgewerkte genoomannotatie voor het zoete aardappelgenoom, wat genfunctionele studies bij zoete aardappelen aanzienlijk zal vergemakkelijken en genomische analyses in de hele Convolvulaceae-familie zal bevorderen."
Over het geheel genomen faciliteert dit verbeterde genomische raamwerk diepere functionele genomica in zoete aardappelen en ondersteunt het geavanceerde veredelingsprogramma's door gedetailleerde miRNA-gegevens en genfunctievoorspellingen te integreren om cultivareigenschappen te verbeteren.