
Wetenschap
Beyond AlphaFold:AI blinkt uit in het creëren van nieuwe eiwitten

Eiwitten die zijn ontworpen met een ultrasnelle softwaretool genaamd ProteinMPNN, vouwen veel eerder op zoals bedoeld. Credit:Ian Haydon, UW Medicine Institute for Protein Design
In de afgelopen twee jaar heeft machinaal leren een revolutie teweeggebracht in de voorspelling van de eiwitstructuur. Nu, drie artikelen in Science beschrijven een soortgelijke revolutie in eiwitontwerp.
In de nieuwe artikelen laten biologen van de University of Washington School of Medicine zien dat machine learning kan worden gebruikt om eiwitmoleculen veel nauwkeuriger en sneller te maken dan voorheen mogelijk was. De wetenschappers hopen dat deze vooruitgang zal leiden tot veel nieuwe vaccins, behandelingen, hulpmiddelen voor het afvangen van koolstof en duurzame biomaterialen.
"Eiwitten zijn fundamenteel in de hele biologie, maar we weten dat alle eiwitten die in elke plant, elk dier en elke microbe worden aangetroffen, veel minder dan één procent uitmaken van wat mogelijk is. Met deze nieuwe softwaretools zouden onderzoekers in staat moeten zijn om oplossingen te vinden voor langdurige -staande uitdagingen op het gebied van geneeskunde, energie en technologie", zegt senior auteur David Baker, hoogleraar biochemie aan de University of Washington School of Medicine en ontvanger van een Breakthrough Prize in Life Sciences 2021.
Eiwitten worden vaak de "bouwstenen van het leven" genoemd omdat ze essentieel zijn voor de structuur en functie van alle levende wezens. Ze zijn betrokken bij vrijwel elk proces dat plaatsvindt in cellen, inclusief groei, deling en herstel. Eiwitten zijn opgebouwd uit lange ketens van chemicaliën die aminozuren worden genoemd. De volgorde van aminozuren in een eiwit bepaalt de driedimensionale vorm. Deze ingewikkelde vorm is cruciaal voor het functioneren van het eiwit.
Onlangs zijn krachtige algoritmen voor machine learning, waaronder AlphaFold en RoseTTAFold, getraind om de gedetailleerde vormen van natuurlijke eiwitten te voorspellen, uitsluitend op basis van hun aminozuursequenties. Machine learning is een vorm van kunstmatige intelligentie waarmee computers kunnen leren van gegevens zonder expliciet te worden geprogrammeerd. Machine learning kan worden gebruikt om complexe wetenschappelijke problemen te modelleren die voor mensen te moeilijk te begrijpen zijn.
Om verder te gaan dan de eiwitten die in de natuur worden aangetroffen, hebben de teamleden van Baker de uitdaging van eiwitontwerp in drie delen opgesplitst en voor elk daarvan nieuwe softwareoplossingen gebruikt.
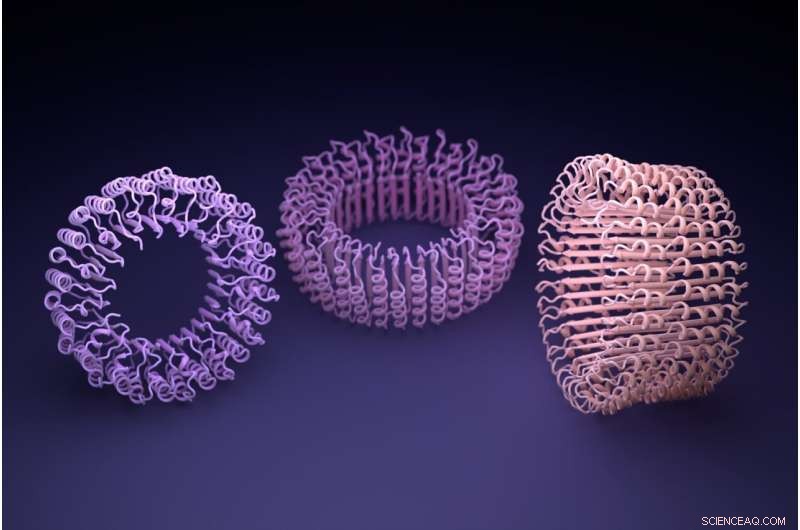
Kunstmatige intelligentie hallucineerde deze symmetrische eiwitassemblages, op een manier die vergelijkbaar is met andere A.!. generatieve tools die output produceren op basis van eenvoudige prompts. Credit:Ian Haydon, UW Medicine Institute for Protein Design
Eerst moet een nieuwe eiwitvorm worden gegenereerd. In een paper gepubliceerd op 21 juli in het tijdschrift Science , toonde het team aan dat kunstmatige intelligentie op twee manieren nieuwe eiwitvormen kan genereren. De eerste, genaamd "hallucinatie", is verwant aan DALL-E of andere generatieve A.I. tools die output produceren op basis van eenvoudige prompts. De tweede, 'inpainting' genoemd, is analoog aan de functie voor automatisch aanvullen die in moderne zoekbalken wordt gevonden.
Ten tweede bedacht het team om het proces te versnellen een nieuw algoritme voor het genereren van aminozuursequenties. Beschreven in het nummer van 15 september van Science , deze softwaretool, genaamd ProteinMPNN, draait in ongeveer een seconde. Dat is meer dan 200 keer sneller dan de vorige beste software. De resultaten zijn superieur aan eerdere tools en de software vereist geen deskundige aanpassingen om te worden uitgevoerd.
"Neurale netwerken zijn gemakkelijk te trainen als je een hoop gegevens hebt, maar met eiwitten hebben we niet zoveel voorbeelden als we zouden willen. We moesten naar binnen gaan en vaststellen welke kenmerken in deze moleculen het belangrijkst zijn. was een beetje vallen en opstaan", zegt projectwetenschapper Justas Dauparas, een postdoctoraal onderzoeker aan het Institute for Protein Design
Ten derde gebruikte het team AlphaFold, een tool ontwikkeld door Alphabet's DeepMind, om onafhankelijk te beoordelen of de aminozuursequenties die ze bedachten waarschijnlijk in de beoogde vormen zouden vouwen.
"Software voor het voorspellen van eiwitstructuren is een deel van de oplossing, maar kan zelf niets nieuws bedenken", legt Dauparas uit.
"ProteinMPNN is voor eiwitontwerp wat AlphaFold was voor het voorspellen van de eiwitstructuur," voegde Baker toe.

Detail van een eiwit dat is ontworpen met behulp van een snel hulpmiddel genaamd ProteinMPNN, een andere vooruitgang in het gebruik van kunstmatige intelligentie en machinaal leren bij het ontwerpen van eiwitten. Credit:Ian Haydon, UW Medicine Institute for Protein Design
In een ander artikel dat verschijnt in Science Op 15 september bevestigde een team van het Baker-lab dat de combinatie van nieuwe machine learning-tools op betrouwbare wijze nieuwe eiwitten zou kunnen genereren die in het laboratorium functioneerden.
"We ontdekten dat eiwitten gemaakt met behulp van ProteinMPNN veel meer kans hadden om op te vouwen zoals bedoeld, en we konden zeer complexe eiwitassemblages maken met deze methoden", zegt projectwetenschapper Basile Wicky, een postdoctoraal onderzoeker aan het Institute for Protein Design.
Onder de nieuwe eiwitten die werden gemaakt, bevonden zich ringen op nanoschaal waarvan de onderzoekers denken dat ze onderdelen kunnen worden voor aangepaste nanomachines. Elektronenmicroscopen werden gebruikt om de ringen te observeren, die een diameter hebben die ongeveer een miljard keer kleiner is dan die van een maanzaad.
"Dit is het allereerste begin van machinaal leren in eiwitontwerp. De komende maanden zullen we werken aan het verbeteren van deze tools om nog meer dynamische en functionele eiwitten te creëren", aldus Baker.
Computerbronnen voor dit werk zijn geschonken door Microsoft en Amazon Web Services. + Verder verkennen
Biologen trainen AI om medicijnen en vaccins te maken
 Het vermogen van SIRT6 om de groei van kankercellen te onderdrukken wordt uitgelegd
Het vermogen van SIRT6 om de groei van kankercellen te onderdrukken wordt uitgelegd Chemici verbeteren waterstofsensoren
Chemici verbeteren waterstofsensoren Nieuwe methode kan leiden tot betere in vivo medicijnafgifte
Nieuwe methode kan leiden tot betere in vivo medicijnafgifte Een veiligere manier om aziden te maken voor gebruik in klikchemie
Een veiligere manier om aziden te maken voor gebruik in klikchemie Nieuwe studie onderzoekt de effectiviteit van nucleair afvalfilter op nanoschaal
Nieuwe studie onderzoekt de effectiviteit van nucleair afvalfilter op nanoschaal
 Wetenschappers vinden eerste bewijs dat microplastics van insecten naar roofdieren in rivieren gaan
Wetenschappers vinden eerste bewijs dat microplastics van insecten naar roofdieren in rivieren gaan Tropische storm Isaias zorgt voor wild weer op de Interstate 95
Tropische storm Isaias zorgt voor wild weer op de Interstate 95 Hogere resolutie in oceaanmodellen:is het het waard?
Hogere resolutie in oceaanmodellen:is het het waard? Een belangrijke scorecard geeft de gezondheid van de Australische omgeving minder dan 1 op 10
Een belangrijke scorecard geeft de gezondheid van de Australische omgeving minder dan 1 op 10 Gecombineerde lokale en mondiale acties kunnen de gevolgen van veranderingen in het mariene milieu verminderen
Gecombineerde lokale en mondiale acties kunnen de gevolgen van veranderingen in het mariene milieu verminderen
Hoofdlijnen
- Planten combineren kleur en geur om bestuivers te verkrijgen
- Continentale controles nodig om de strijd tegen boomziekten te handhaven
- Een 3D-model plantencel maken zonder voedsel
- Celsignalen die wondgenezing in gang zetten zijn verrassend complex
- Meiosis 2: Definition, Stages, Meiosis 1 vs Meiosis 2
- Hoe dieren stemmen om groepsbeslissingen te nemen
- 3 soorten mutaties die kunnen optreden in het DNA Molecuul
- Hoe een cel te splitsen in Two
- 195 manieren om de geschilderde dames van Californië te helpen:nieuwe voedselbronnen bieden hoop voor bedreigde vlinders
- Telofase: wat gebeurt er in deze fase van mitose en meiose?
- Australische honden getraind om bedreigde diersoorten op te sporen

- Cellulaire ademhaling: definitie, vergelijking en stappen
- Een betere manier om RNA-virusnaalden te vinden in hooibergen van databases
- Eekhoorns en hamsters die in winterslaap zijn, zijn geëvolueerd om het minder koud te hebben

 Supersnelle lichtpulsen beeldhouwen:Nanopillars vormen licht precies voor praktische toepassingen
Supersnelle lichtpulsen beeldhouwen:Nanopillars vormen licht precies voor praktische toepassingen Geavanceerde wiskunde verdicht de complexiteit van COVID-19
Geavanceerde wiskunde verdicht de complexiteit van COVID-19 Nieuwe methode voor het bestuderen van sediment kan de impact van klimaatverandering voorspellen
Nieuwe methode voor het bestuderen van sediment kan de impact van klimaatverandering voorspellen Nieuwe methodologie voorspelt coronavirus en andere infectieziektebedreigingen voor dieren in het wild
Nieuwe methodologie voorspelt coronavirus en andere infectieziektebedreigingen voor dieren in het wild NASA vindt vervagende subtropische storm Beryl zonder centrumneerslag
NASA vindt vervagende subtropische storm Beryl zonder centrumneerslag EU-vrouwen verdienen 16 procent minder dan mannen:Eurostat
EU-vrouwen verdienen 16 procent minder dan mannen:Eurostat Majorana Photons:Nieuwe superklasse fotonen kan reizen met verschillende golffronten
Majorana Photons:Nieuwe superklasse fotonen kan reizen met verschillende golffronten Bouwden een 1, 300 km lang ondergronds wetenschappelijk experiment om 's werelds meest ongrijpbare deeltjes te bestuderen
Bouwden een 1, 300 km lang ondergronds wetenschappelijk experiment om 's werelds meest ongrijpbare deeltjes te bestuderen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

