Wetenschap
Nieuwe technologie verlicht microben die niet in een laboratorium kunnen worden gekweekt

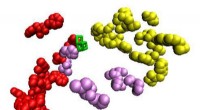
Wetenschappers gebruikten dit geïntegreerde microfluïdische circuit om mini-metagenomische microbiële celpartitionering en genomische DNA-amplificatie uit te voeren. Krediet:Brian Yu
Aan de Stanford-universiteit, onderzoekers hebben een nieuw microfluïdisch analysesysteem gebruikt om 29 nieuwe microbiële genomen (de complete set genetisch materiaal) te extraheren uit monsters van twee warmwaterbronnen in Yellowstone National Park. Ze extraheren de genomen met behoud van eencellige resolutie, wat betekent dat ze wisten uit welke cellen het genetische materiaal kwam. Dit werk werd mogelijk gemaakt door een nieuwe technologie die het monster verdeelt om nauwkeurige analyse van het genetische materiaal van een microbe mogelijk te maken. specifiek, het biedt details over de genoomfunctie en overvloed. Het werk werd mogelijk gemaakt door het Emerging Technology Opportunity Program, een onderdeel van het US Department of Energy Joint Genome Institute (DOE JGI), een DOE Office of Science gebruikersfaciliteit.
Deze nieuwe technologie verlicht microbiële "donkere materie, " genetische informatie van het grootste deel van de microbiële diversiteit van de planeet die niet in een laboratorium is gekweekt. Deze microben leven op locaties zo divers als warmwaterbronnen en woestijnen, onder Antarctisch ijs en in de afvoer van zuurmijnen van Superfund-locaties. Instrumenten die de genetica en het metabolisme van microben kunnen bepalen, zullen toepassingen hebben op gebieden variërend van bio-energie tot biotechnologie tot milieuonderzoek.
Er zijn meer dan 50, 000 microbiële genoomsequenties in de DOE JGI's Integrated Microbial Genomes openbaar toegankelijke database, en veel van hen zijn ontdekt met behulp van metagenomische sequencing en eencellige genomica. Ondanks hun nut, deze sequencing- en genomics-technieken hebben grenzen:amplificaties van het eencellige genoom zijn tijdrovend, vaak onvolledig, en metagenomische sequencing werkt over het algemeen het beste als het omgevingsmonster niet te complex is. In eLife, een team van onderzoekers van Stanford University rapporteert de ontwikkeling van een op microfluïdica gebaseerde, mini-metagenomics-benadering om deze uitdagingen te verminderen. De techniek begint met het verminderen van de complexiteit van het omgevingsmonster door het te scheiden, met behulp van microfluïdica, in 96 submonsters met elk 5 tot 10 cellen. Vervolgens, de genomen in de cellen in elk deelmonster worden geamplificeerd en bibliotheken worden gecreëerd voor het sequensen van deze mini-metanomen. De kleinere submonsters kunnen voor statistische analyses op eencellige resolutie worden gehouden. Gelijktijdige patronen van veel submonsters kunnen ook worden gebruikt om sequentie-onafhankelijke genoombinning uit te voeren. De technologie is ontwikkeld met behulp van middelen die worden geleverd door het DOE JGI's Emerging Technologies Opportunity Program, die in 2013 werd gelanceerd.
Het doel van JGI's Emerging Technologies Opportunity Program is om deze nieuwe technologieën te gebruiken om energie- en milieutoepassingen aan te pakken, waarde toevoegen aan de high-throughput sequencing en analyse die wordt gedaan voor DOE JGI-gebruikers. Het team valideerde de techniek met behulp van een synthetische microbiële gemeenschap, en vervolgens toegepast op monsters van de Bijah en Mound warmwaterbronnen in Yellowstone National Park. Een van hun bevindingen was dat de microben in Mound Spring een groter potentieel hadden om methaan te produceren dan de microben van Bijah Spring. Ze identificeerden ook een microbieel genoom van Bijah Spring dat nitriet zou kunnen reduceren tot stikstof. Het toepassen van deze nieuwe technologie op extra monsterlocaties zal bijdragen aan het bereik van tot nu toe niet-gekarakteriseerde microbiële mogelijkheden met potentiële DOE-missie-toepasbaarheid.
 El Nino-gekoppelde afname van bodemvocht kan leiden tot massale afsterving van tropische planten
El Nino-gekoppelde afname van bodemvocht kan leiden tot massale afsterving van tropische planten Nieuwe kaart onthult verloren habitat in estuarium aan de westkust
Nieuwe kaart onthult verloren habitat in estuarium aan de westkust Bergwolken - van regenmakers tot sneeuwmakers
Bergwolken - van regenmakers tot sneeuwmakers Wat zijn de oplossingen voor door klimaatverandering beïnvloede druiven?
Wat zijn de oplossingen voor door klimaatverandering beïnvloede druiven? Devoon integratieve stratigrafie en tijdschaal van China
Devoon integratieve stratigrafie en tijdschaal van China
Hoofdlijnen
- Moleculaire genetica (biologie): een overzicht
- De chemische stof die planten vertelt wanneer het tijd is om te slapen
- Welke organellen zijn membraansusjes die worden gebruikt om moleculen te transporteren?
- Nieuwe bevindingen over de biomechanica en evolutie van zuigvallen in vleesetende blaasjes
- Iraakse dierenliefhebbers gaan online om zwerfdieren in Bagdad te redden
- Wat is een koolhydraat gevonden in een celwand van plantencellen?
- Wat is sterker:nature of nurture?
- Zooplankton Vs. fytoplankton
- Het leefgebied van de panda krimpt, steeds meer gefragmenteerd worden
 Winst Samsung overtreft verwachtingen dankzij geheugenchips
Winst Samsung overtreft verwachtingen dankzij geheugenchips Onderzoekers bevestigen dat mensen hele groepen beoordelen op basis van de prestaties van het eerste lid
Onderzoekers bevestigen dat mensen hele groepen beoordelen op basis van de prestaties van het eerste lid Rangschikking van steden over de hele wereld op toegankelijkheid van vervoer
Rangschikking van steden over de hele wereld op toegankelijkheid van vervoer Hoe een binomiale tabel te gebruiken
Hoe een binomiale tabel te gebruiken SoftBank Group nettowinst over negen maanden stijgt met meer dan 50%
SoftBank Group nettowinst over negen maanden stijgt met meer dan 50% Waarom de oproepen voor de defunding van de politie?
Waarom de oproepen voor de defunding van de politie? Hoe beïnvloeden landforms mensen?
Hoe beïnvloeden landforms mensen?  NASA ziet ex-tropische cycloon Debbie boven Queensland
NASA ziet ex-tropische cycloon Debbie boven Queensland
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com