
Wetenschap
Onderzoekers brengen het menselijk genoom in 4-D in kaart terwijl het vouwt
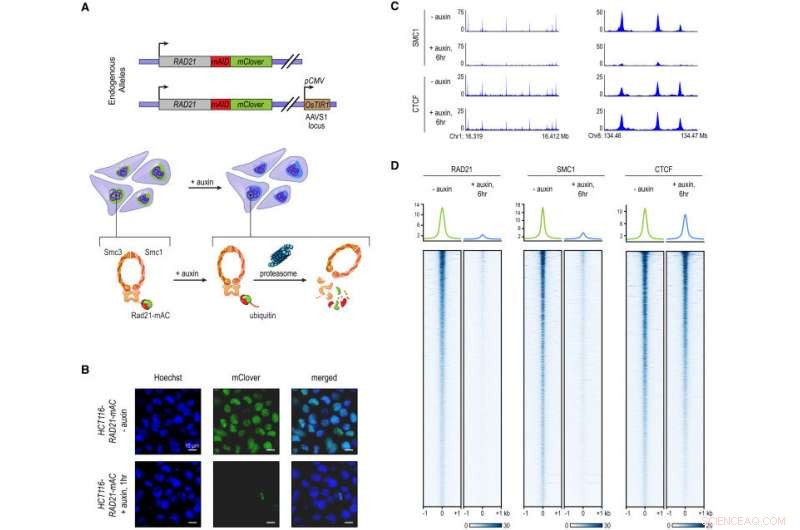
Tagging van endogene RAD21 met een auxine-induceerbare Degron zorgt voor snelle, Bijna volledig cohesineverlies. (A) In HCT-116-RAD21-mAC-cellen, beide RAD21-allelen zijn gelabeld met auxine-induceerbare degrons en een mClover-reporter, en het OsTIR1-gen is geïntegreerd op de AAVS1-locus. Behandeling met auxine leidt tot proteasomale afbraak van RAD21. (B) Live-celbeeldvorming na Hoechst 33342-kleuring om kernen te labelen. Nucleaire mClover-fluorescentie die overeenkomt met gelabeld RAD21 ging verloren na 1 uur auxinebehandeling. (C) SMC1 en CTCF ChIP-seq signaal met en zonder auxine behandeling. (D) RAD21, SMC1, en CTCF ChIP-seq-signaal (links, midden, rechts) over alle pieken die werden opgeroepen voor elk van de eiwitten in onbehandelde RAD21-mAC-cellen. Boven:Gemiddelde verrijkingen voor elk eiwit. Na degradatie van RAD21, het cohesinecomplex bindt niet langer aan chromatine. CTCF-binding wordt niet beïnvloed. Credit: Cel . DOI:10.1016/j.cell.2017.09.026
Een multi-institutioneel team van Baylor College of Medicine, Rijst Universiteit, Stanford University en het Broad Institute of MIT en Harvard hebben de eerste 4-D-kaart met hoge resolutie van genoomvouwen gemaakt, het volgen van een volledig menselijk genoom terwijl het in de loop van de tijd vouwt. Het verslag, die kunnen leiden tot nieuwe manieren om genetische ziekten te begrijpen, verschijnt op de omslag van Cel .
Connecties maken
Al decenia, onderzoekers vermoedden dat wanneer een menselijke cel op een stimulus reageert, DNA-elementen die ver uit elkaar liggen in het genoom vinden elkaar snel, vormen lussen langs het chromosoom. Door deze DNA-elementen in de ruimte te herschikken, de cel kan veranderen welke genen actief zijn.
In 2014, hetzelfde team van wetenschappers toonde aan dat het mogelijk was om deze lussen in kaart te brengen. Maar de eerste kaarten waren statisch, zonder de mogelijkheid om de loops te zien veranderen. Het was onduidelijk of, in de overvolle ruimte van de kern, DNA-elementen zouden elkaar snel genoeg kunnen vinden om cellulaire reacties te beheersen.
"Voordat, we konden kaarten maken van hoe het genoom vouwde toen het zich in een bepaalde staat bevond, maar het probleem met een statisch beeld is dat als er nooit iets verandert, het is moeilijk om erachter te komen hoe dingen werken, " zei Suhas Rao, eerste auteur van de nieuwe studie. "Onze huidige aanpak lijkt meer op het maken van een film; we kunnen kijken hoe plooien verdwijnen en weer verschijnen."
Een ring om ze allemaal te regeren
Om het vouwproces in de loop van de tijd te volgen, het onderzoeksteam begon met het verstoren van cohesin, een ringvormig eiwitcomplex dat zich op de grenzen van bijna alle bekende lussen bevond. in 2015, het team stelde voor dat cohesine DNA-lussen in de celkern creëert door een proces van extrusie.
"Extrusie werkt als de riemlengteversteller op een rugzak, " verklaarde Dr. Erez Lieberman Aiden, directeur van het Center for Genome Architecture aan het Baylor College of Medicine en senior auteur van de nieuwe studie. "Als je de riem door een van beide kanten voert, het vormt een lus. DNA lijkt hetzelfde te doen - behalve dat cohesineringen de rol van de regelaar lijken te spelen."
Aiden zei dat een cruciale voorspelling van het 2015-model is dat alle lussen zouden moeten verdwijnen bij afwezigheid van cohesine. In het nieuwe onderzoek Aiden, Rao en collega's testten die veronderstelling.
"We ontdekten dat toen we cohesin verstoorden, duizenden lussen verdwenen, " zei Rao, een medische student aan de Stanford University, Hertz Fellow en lid van het Aiden-lab. "Vervolgens, als we cohesin terugplaatsen, al die lussen kwamen terug - vaak in een kwestie van minuten. Dit is precies wat je zou voorspellen uit het extrusiemodel, en het suggereert dat de snelheid waarmee cohesine langs het DNA beweegt, een van de snelste is voor alle bekende menselijke eiwitten."
Lussen versus groepen
Maar niet alles gebeurde zoals de onderzoekers hadden verwacht. In sommige gevallen, loops deed precies het tegenovergestelde van wat de onderzoekers verwachtten.
"Toen we zagen hoe duizenden lussen in het genoom zwakker werden, we zagen een grappig patroon, " zei Aiden, ook een McNair-geleerde, Hertz Fellow en senior onderzoeker aan het Centre for Theoretical Biological Physics van de Rice University. "Er waren een paar vreemde lussen die eigenlijk sterker werden. Toen, als we cohesin terugzetten, de meeste lussen herstelden volledig - maar deze vreemde lussen deden opnieuw het tegenovergestelde - ze verdwenen!"
Door nauwkeurig te onderzoeken hoe de kaarten in de loop van de tijd veranderden, het team realiseerde zich dat extrusie niet het enige mechanisme was dat DNA-elementen samenbracht. Een tweede mechanisme, compartimentering genoemd, cohesine was niet betrokken.
"Het tweede mechanisme dat we hebben waargenomen is heel anders dan extrusie, " legde Rao uit. "Extrusie heeft de neiging om DNA-elementen met twee tegelijk samen te brengen, en alleen als ze op hetzelfde chromosoom liggen. Dit andere mechanisme kan grote groepen elementen met elkaar verbinden, zelfs als ze op verschillende chromosomen liggen. En het lijkt net zo snel te gaan als extrusie."
Directeur van het brede instituut Eric Lander, een studie co-auteur, zei, "We beginnen de regels te begrijpen waarmee DNA-elementen in de kern samenkomen. Nu we de elementen kunnen volgen terwijl ze in de loop van de tijd bewegen, de onderliggende mechanismen beginnen duidelijker te worden."
Hoofdlijnen
- Your Brain On: Exam Stress
- Wat u moet weten over mitose voor een test
- Hoe gender en stereotypen onze relatie met honden kunnen bepalen
- Hoe duplicaten in twee rijen in OpenOffice te verwijderen
- Prozac in oceaanwater een mogelijke bedreiging voor het zeeleven, studie vondsten
- Wat is Herschikking in Meiose?
- Zit moraliteit in de hersenen?
- Zelfvernietiging van muggen
- Waarom zijn we gewelddadig?
- Wetenschappers creëren op verzoek endocytose door cellen warm te bedraden
- Waarom was frenologie een rage in de Victoriaanse tijd?

- Nieuwsgierige grote witte haai speelt met camera

- Zal de Hayflick-limiet ons ervan weerhouden om voor altijd te leven?
- Entomoloog ontdekt ongewervelde dieren die in meer kleurencombinaties voorkomen dan alle andere

 Apple neemt de kroon op de verkoop van smartphones van Samsung
Apple neemt de kroon op de verkoop van smartphones van Samsung Hoe de ontbrekende kant van een juiste driehoek te vinden
Hoe de ontbrekende kant van een juiste driehoek te vinden  Zeldzaam Byzantijns mozaïek onthuld in de oude stad van Jeruzalem
Zeldzaam Byzantijns mozaïek onthuld in de oude stad van Jeruzalem Wat is de beste manier om onderzoeksinstituten te rangschikken?
Wat is de beste manier om onderzoeksinstituten te rangschikken? Ruimtewandeling een succes voor Frans, Amerikaanse astronauten
Ruimtewandeling een succes voor Frans, Amerikaanse astronauten AAAS-panel richt zich op routekaart naar radicale transformatie van de AI-onderzoeksonderneming
AAAS-panel richt zich op routekaart naar radicale transformatie van de AI-onderzoeksonderneming Nieuwe methode kan scheuren in metaal opsporen lang voordat ze catastrofes veroorzaken
Nieuwe methode kan scheuren in metaal opsporen lang voordat ze catastrofes veroorzaken Een studie van microbiële bewoners van Monterey Canyons kan inzichten bieden over evolutie
Een studie van microbiële bewoners van Monterey Canyons kan inzichten bieden over evolutie
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com


