
Wetenschap
Informatie opslaan in DNA:DNA-opslag verbeteren met elektrodeputjes op nanoschaal
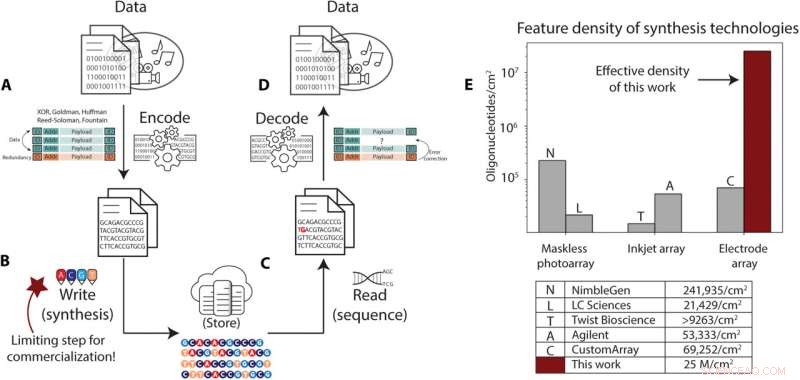
De opslag van DNA-gegevens vereist een hogere synthesecapaciteit dan met de huidige technieken mogelijk is. (A tot D) Overzicht van de pijplijn voor DNA-gegevensopslag. (A) Digitale gegevens worden vanuit hun binaire representatie gecodeerd in sequenties van DNA-basen, met een identifier die ze correleert met een data-object, informatie adresseert die wordt gebruikt om de gegevens opnieuw te ordenen bij het lezen, en redundante informatie die wordt gebruikt voor foutcorrectie. (B) Deze sequenties worden gesynthetiseerd in DNA-oligonucleotiden en opgeslagen. (C) Tijdens het ophalen worden de DNA-moleculen geselecteerd en gekopieerd via PCR of andere methoden en terug gesequenced in elektronische representaties van de basen in deze sequenties. (D) Het decoderingsproces neemt deze luidruchtige en soms onvolledige reeks sequentielezingen, corrigeert voor fouten en ontbrekende sequenties en decodeert de informatie om de gegevens te herstellen. (E) Samenvatting van de commerciële syntheseprocessen en bijbehorende geschatte dichtheden van oligonucleotiden, zoals gerapporteerd in de literatuur of door de bedrijven zelf. De dichtheid van onze elektrochemische methode is donkerrood gemarkeerd. Krediet:Wetenschappelijke vooruitgang , 10.1126/sciadv.abi6714
Genetici kunnen gegevens in synthetisch DNA opslaan als medium voor langdurige opslag vanwege de dichtheid, het kopieergemak, de lange levensduur en de duurzaamheid. Onderzoek in het veld was onlangs gevorderd met nieuwe coderingsalgoritmen, automatisering, bewaring en sequencing. Desalniettemin blijft de meest uitdagende hindernis bij de implementatie van DNA-opslag de schrijfdoorvoer, die de gegevensopslagcapaciteit kan beperken. In een nieuw rapport hebben Bichlien H. Nguyen en een team van wetenschappers in Microsoft Research en computerwetenschappen en engineering aan de Universiteit van Washington, Seattle, VS, de eerste schrijver voor DNA-opslag op nanoschaal ontwikkeld. Het team was van plan de DNA-schrijfdichtheid te schalen naar 25 x 10 6 sequenties per vierkante centimeter, een verbeterde opslagcapaciteit in vergelijking met bestaande DNA-synthese-arrays. De wetenschappers hebben met succes een bericht in DNA geschreven en gedecodeerd om een praktisch DNA-gegevensopslagsysteem op te zetten. De resultaten zijn nu gepubliceerd in Science Advances .
Lange termijn DNA-archieven
Het huidige tempo van datageneratie overtreft de bestaande opslagcapaciteiten, DNA is een veelbelovende oplossing voor dit probleem bij een verwachte praktische dichtheid van meer dan 60 petabyte per kubieke centimeter. Het materiaal is duurzaam onder verschillende omstandigheden, relevant en gemakkelijk te kopiëren, met de belofte om duurzamer of groener te zijn dan commerciële media. Tijdens het proces kunnen digitale gegevens in de vorm van sequenties van bits worden gecodeerd in sequenties van de vier natuurlijke DNA-basen - guanine, adenine, thiamine en cytosine, hoewel aanvullende basen ook mogelijk zijn. Het team kan vervolgens de sequenties in moleculaire vorm schrijven via de novo DNA-oligonucleotidesynthese om specifieke moleculen te creëren op basis van een reeks herhalende chemische stappen. De resulterende oligonucleotiden kunnen na synthese worden bewaard en bewaard. Om toegang te krijgen tot de gegevens, kan de DNA-opslag worden geamplificeerd met behulp van polymerasekettingreacties en kan de sequentie worden bepaald om de DNA-basesequenties terug te brengen naar het digitale domein, waarna de DNA-basesequenties kunnen worden gedecodeerd om de oorspronkelijke sequentie van bits te herstellen.
Overzicht van 650-nm-array met een pitch van 2 m. (A) Eindige elementenanalyse van anodische zuurgeneratie en diffusie bij een elektrode met een diameter van 650 nm en een putje van 200 nm wordt afgebeeld met een dwarsdoorsnede langs het y =x-vlak en (B) bovenaanzicht op de z =0 vlak. De kleuren blauw en geel vertegenwoordigen gebieden met respectievelijk relatief lage en hoge zuurconcentraties. (C) Een overzicht van de DNA-synthese-array op nanoschaal met scanning-elektronenmicroscopiebeelden van de 650-nm-elektrodearray en vergrote weergave van één elektrode. (D) Een fluorescerend beeld waarin de put rond elke geactiveerde anode een patroon heeft met AAA-fluoresceïne. Het cartoondiagram laat zien welke elektroden in de lay-out werden geactiveerd. (E) Illustratie van de putjes met AAA-fluoresceïne en AAA-AquaPhluor en (F) overeenkomstige beeldoverlay van de twee fluoroforen aan het einde van DNA gesynthetiseerd op dezelfde 650 nm elektrode-array. Krediet:Wetenschappelijke vooruitgang , 10.1126/sciadv.abi6714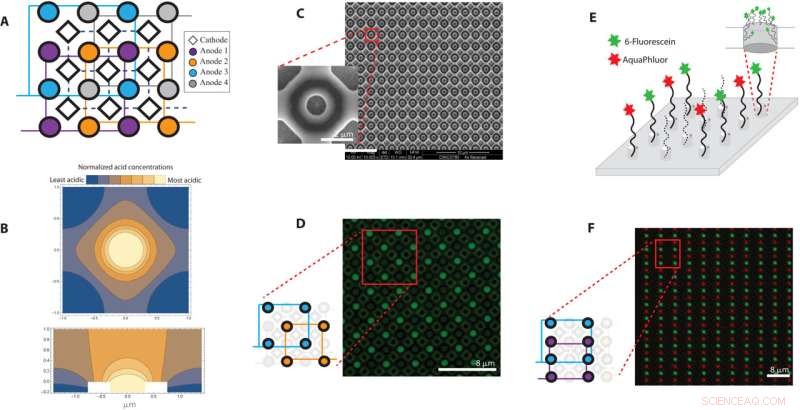
In deze studie hebben Nguyen et al. produceerde een elektrode-array die onafhankelijke elektrode-specifieke controle van DNA-synthese demonstreerde met elektrodegroottes en pitches om de synthesedichtheid van 25 miljoen oligonucleotiden per cm te bepalen 2 . Deze waarde wordt geschat als de elektrodedichtheid die nodig is om het minimale doel van kilobytes per seconde aan gegevensopslag in DNA te bereiken. Het team pushte de state-of-the-art in elektronisch-chemische controle en leverde experimenteel bewijs voor de schrijfbandbreedte die nodig is voor DNA-gegevensopslag.
Het team introduceerde een proof-of-concept moleculaire controller in de vorm van een klein schrijfmechanisme voor DNA-opslag op een chip. De chip kan de DNA-synthese stevig inpakken met een grootte van 3 orden hoger dan voorheen om een grotere DNA-schrijfcapaciteit te bereiken. Om informatie in DNA op te slaan op de schaal die nodig is voor commercieel gebruik waren twee cruciale processen nodig. Eerst moest het team digitale bits (enen en nullen) vertalen in strengen synthetisch DNA die bits vertegenwoordigen met coderende software en een DNA-synthesizer. Vervolgens moeten ze de informatie kunnen lezen en decoderen tot op de bits om die informatie weer in digitale vorm te herstellen met een DNA-sequencer en decoderingssoftware.
Ontwikkelen van elektrochemische arrays voor functies op nanoschaal
Tijdens de traditionele synthese van DNA-ketens gebruiken wetenschappers een meerstapsmethode die bekend staat als fosforamidietchemie, waarbij een DNA-keten achtereenvolgens kan worden gekweekt door toevoeging van DNA-basen. Elke DNA-base bevat een blokkerende groep om meerdere toevoegingen van DNA-basen aan de groeiende keten te voorkomen. Bij hechting aan een DNA-keten kan zuur in de opstelling worden afgeleverd om de blokkerende groep te splitsen en de DNA-keten te primen om de volgende base toe te voegen. Tijdens elektrochemische DNA-synthese bevat elke plek in de array een elektrode en wanneer een spanning wordt aangelegd, wordt zuur gegenereerd op de werkelektrode (anode) om de groeiende DNA-ketens te deblokkeren, terwijl een equivalente base wordt gegenereerd op de tegenelektrode (kathode) . Het team voorkwam zuurdiffusie in de opstelling door een elektrode-array te ontwerpen, waarbij elke werkende elektrode waaromheen zuurvorming tijdens de DNA-synthese plaatsvond, in een putje werd verzonken en omringd door vier gemeenschappelijke tegenelektroden, d.w.z. kathoden die de basisvorming aanstuurden, om het zuur naar specifieke regio's. Nguyen et al. verifieerde de effectiviteit van het ontwerp met behulp van eindige elementenanalyse. Tijdens de experimenten deblokkeerde het zuur, wanneer het in voldoende concentratie werd gepresenteerd, de oppervlaktegebonden nucleotiden om het volgende nucleotide te laten koppelen. Met behulp van chips met kenmerkende vlekken om zuren in te sluiten, ontwikkelden ze elektrochemische arrays met vier individuele elektroden om de DNA-synthese te reguleren. Het team voerde vervolgens experimenten uit met twee fluorescerend gelabelde basen in groen en rood. Als proof of concept toonden ze het vermogen van het apparaat om gegevens te schrijven door vier unieke DNA-strengen, elk 100 basen lang, te synthetiseren met een gecodeerd bericht, zonder fouten.
-
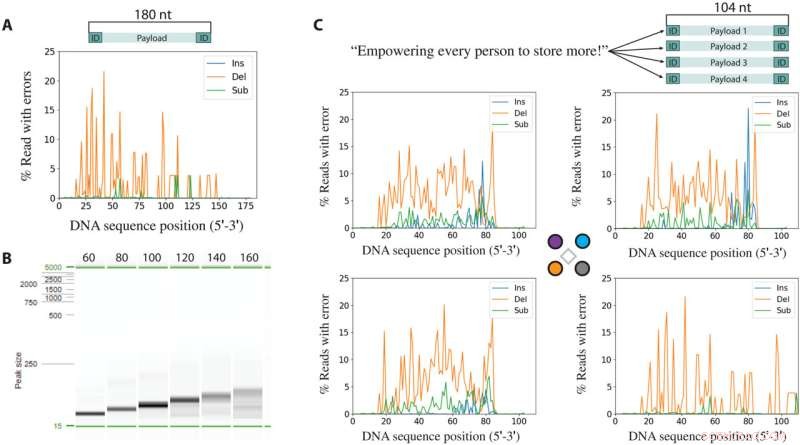
Fouten die voortkomen uit synthese gevolgd door sequencing. (A) Inserties (Ins), deleties (Del) en vervangingen (Sub) per positie voor een gesynthetiseerde en PCR-versterkte 180-base sequentie. (B) Elektroforesebeeld van syntheseproducten na PCR-amplificatie. (C) Bericht gecodeerd in 64 bytes opgesplitst in vier unieke reeksen van 104 basen (bovenaan). Inserties, deleties en substituties per locus van elk van de vier sequenties in de multiplexsyntheserun. In elke foutanalysegrafiek zijn de terminale 20 basen aan zowel de 3'- als de 5'-uiteinden afkomstig van de primers die worden gebruikt in PCR en zijn niet representatief voor de gesynthetiseerde fouten. Krediet:Wetenschappelijke vooruitgang , 10.1126/sciadv.abi6714
-
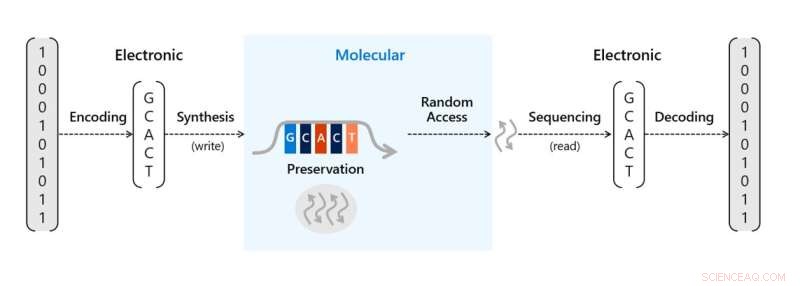
Opschalen van DNA-gegevensopslag met elektrodeputjes op nanoschaal. Klein schrijfmechanisme voor DNA-opslag op een chip. Krediet:Microsoft Research Blog, Wetenschapsvooruitgang , 10.1126/sciadv.abi6714
Vooruitzichten:synthetiseren van korte oligonucleotiden op de elektrode-array voor gegevensopslag
Met behulp van de opstelling, Nguyen et al. demonstreerde ook ruimtelijk gecontroleerde synthese van korte oligonucleotiden op de elektrode-array om de maximale lengte van DNA te bepalen die kon worden gevormd. De wetenschappers creëerden een enkele DNA-sequentie met 180 nucleotiden en PCR-geamplificeerde producten van verschillende lengte uit de volledige lengte van de oligonucleotiden. Naarmate het amplicon langer werd, leken de verwachte PCR-producten zwakker en minder goed gedefinieerd, terwijl kortere amplicons sterkere en beter gedefinieerde banden vertoonden die wijzen op hogere synthesefouten. Op basis van de resultaten selecteerden de onderzoekers een sequentielengte van 100 basen voor gemakkelijke zuivering om een praktische demonstratie van DNA-gegevensopslag te bieden zonder verdere optimalisatie. Op deze manier effende de proof-of-concept-methode die in dit werk door Bichlien H. Nguyen en collega's wordt gedemonstreerd, de weg voorwaarts om grootschalige en unieke DNA-sequenties parallel te genereren voor gegevensopslag. Het werk overtrof eerdere rapporten over dichte synthetische DNA-sequenties om een eerste experimentele indicatie te bieden om de schrijfbandbreedte te bereiken die nodig is voor gegevensopslag op nanoschaal. De wetenschappers verwachten onmiddellijke toepassingen van de apparaten in de informatietechnologie en voorzien hun praktische toepassingen in materiaalwetenschap, synthetische biologie en grootschalige moleculaire biologie-assays. + Verder verkennen
Enzymatische DNA-synthese ziet het licht
© 2021 Science X Network
 Natuurlijke antibiofilm en antimicrobiële peptiden van micro-organismen
Natuurlijke antibiofilm en antimicrobiële peptiden van micro-organismen Vacaturedynamiek op met CO bedekte Pt(111)-elektroden
Vacaturedynamiek op met CO bedekte Pt(111)-elektroden Gassen scheiden met flexibele moleculaire zeven
Gassen scheiden met flexibele moleculaire zeven Water efficiënter omzetten in waterstof
Water efficiënter omzetten in waterstof Instaan voor Vonnegut:ingenieurs bestuderen polymeer dat bevriest bij kamertemperatuur
Instaan voor Vonnegut:ingenieurs bestuderen polymeer dat bevriest bij kamertemperatuur
 Voedselkeuzes van consumenten kunnen de uitstoot van broeikasgassen helpen verminderen en bijdragen aan klimaatverandering
Voedselkeuzes van consumenten kunnen de uitstoot van broeikasgassen helpen verminderen en bijdragen aan klimaatverandering Studie brengt stroomgebieden tussen stad en platteland in kaart en wijst op manieren om de beleids- en planningscoördinatie voor de landbouw te optimaliseren
Studie brengt stroomgebieden tussen stad en platteland in kaart en wijst op manieren om de beleids- en planningscoördinatie voor de landbouw te optimaliseren We beginnen het mysterie te ontrafelen van hoe bliksem en onweer werken
We beginnen het mysterie te ontrafelen van hoe bliksem en onweer werken Interne waakhond zegt dat EPA het opruimen van giftige sites verkeerd beheert
Interne waakhond zegt dat EPA het opruimen van giftige sites verkeerd beheert Benzinestations laten veel meer giftige dampen ontsnappen dan eerder werd gedacht
Benzinestations laten veel meer giftige dampen ontsnappen dan eerder werd gedacht
Hoofdlijnen
- Florida probeert het tij te keren bij de invasie van leguaan
- Volledige structuur van mitochondriaal respiratoir supercomplex gedecodeerd
- Hoe Agarose Gel te interpreteren
- Video:Op weg naar nul honger wereldwijd
- Plantenveredeling:onzichtbare chromosomen gebruiken om pakketjes positieve eigenschappen door te geven
- Angst voor haaien beïnvloedt zeewiergroei op Fijische koraalriffen
- Computerprogramma detecteert verschillen tussen menselijke cellen
- Wat is een homoloog allel?
- Ideeën voor Cookie Science Fair Projects
- Energie-efficiënte grafeenmembranen kunnen leiden tot verhoogde aardgasproductie, minder CO2-vervuiling

- Nano-antioxidanten bewijzen hun potentieel
- Op grafeen gebaseerde schijven zorgen voor veilige opslag

- Composietmateriaal slim maken met edelmetaal

- Weefselgravende nanodrills richten net genoeg schade aan
 Welke soorten objecten worden aangetrokken door magneten?
Welke soorten objecten worden aangetrokken door magneten?  Vergulde nano-bits vinden, kankercellen vernietigen
Vergulde nano-bits vinden, kankercellen vernietigen Als het zee-ijs verdwijnt, er ontstaat een groener en bruiner noordpoolgebied
Als het zee-ijs verdwijnt, er ontstaat een groener en bruiner noordpoolgebied Vitale economische gegevens zijn waarschijnlijk verloren gegaan tijdens de sluiting - dit is waarom het belangrijk is voor alle Amerikanen
Vitale economische gegevens zijn waarschijnlijk verloren gegaan tijdens de sluiting - dit is waarom het belangrijk is voor alle Amerikanen Wetenschappers ontwikkelen elektrochemisch platform voor celvrije synthetische biologie
Wetenschappers ontwikkelen elektrochemisch platform voor celvrije synthetische biologie De kleinste regenbogen van de natuur, gemaakt door pauwspinnen, kan nieuwe optische technologie inspireren
De kleinste regenbogen van de natuur, gemaakt door pauwspinnen, kan nieuwe optische technologie inspireren Centimeters converteren naar Cubic Feet
Centimeters converteren naar Cubic Feet Kunstmatige fotosynthese krijgt grote boost van nieuwe katalysator
Kunstmatige fotosynthese krijgt grote boost van nieuwe katalysator
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

