
Wetenschap
Snelle atoomkrachtmicroscopie neemt intrinsiek ongeordende eiwitten aan
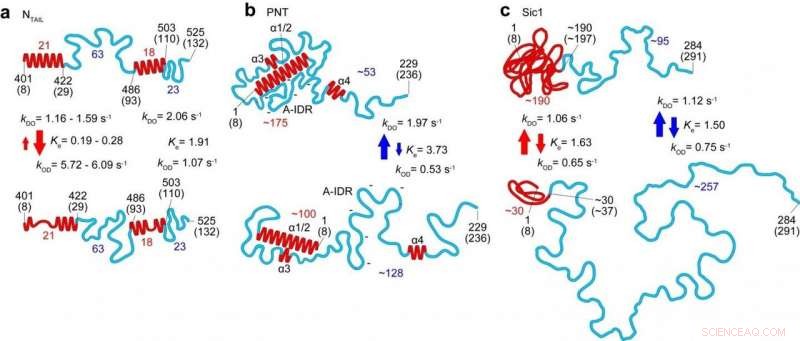
Structurele en dynamische kenmerken van drie IDP's (N STAART , PNT en Sic1) onthuld door HS-AFM-beeldvorming. De boven- en onderpanelen komen overeen met de meer geordende en minder geordende toestanden, respectievelijk. De cijfers in rood en blauw vertegenwoordigen het aantal aminozuren in de respectievelijke gevouwen regio's en volledig ongeordende regio's, respectievelijk. De rode en blauwe pijlen geven de kinetische aard aan van veranderingen in hoogte van gevouwen gebieden en end-to-end afstand van volledig ongeordende gebieden, respectievelijk. Krediet:Kanazawa University
Ons begrip van biologische eiwitten correleert niet altijd met hoe vaak of belangrijk ze zijn. De helft van alle eiwitten, moleculen die een integrale rol spelen in celprocessen, zijn intrinsiek ongeordend, wat betekent dat veel van de standaardtechnieken voor het onderzoeken van biomoleculen er niet op werken. Nu hebben onderzoekers van de Kanazawa University in Japan aangetoond dat hun in eigen land ontwikkelde high-speed atomic force microscopie-technologie niet alleen informatie kan verschaffen over de structuren van deze eiwitten, maar ook over hun dynamiek.
Begrijpen hoe een eiwit in elkaar zit, geeft waardevolle aanwijzingen voor zijn functies. De ontwikkeling van eiwitkristallografie in de jaren dertig en vijftig bracht voor het eerst verschillende eiwitstructuren in beeld, maar het werd geleidelijk duidelijk dat een groot deel van de eiwitten geen enkele vaste structuur heeft, waardoor ze onhandelbaar zijn voor röntgenkristallografie. Omdat ze te dun zijn voor elektronenmicroscopie, de enige haalbare alternatieven voor veel van deze intrinsiek ongeordende eiwitten (IDP's) zijn kernspinresonantiebeeldvorming en röntgenverstrooiing onder kleine hoeken. Gegevens die met deze technieken zijn verzameld, worden gemiddeld over ensembles en geven dus geen duidelijke indicatie van individuele eiwitconformaties of hoe vaak ze voorkomen. Atoomkrachtmicroscopie daarentegen is in staat tot biologische beeldvorming op nanoschaal met hoge snelheid, dus het kan zowel dynamiek als eiwitstructuren vastleggen.
In dit laatste werk onderzoekers van Kanazawa University naast medewerkers in Japan, Frankrijk en Italië pasten de techniek toe bij de studie van verschillende ontheemden en identificeerden parameters die de vorm bepalen, grootte en ketenlengte van eiwitgebieden, evenals een machtswet die de eiwitgrootte relateert aan de eiwitlengte, en een kwantitatieve beschrijving van het effect van het mica-oppervlak op eiwitafmetingen. De dynamiek van de eiwitconformaties die zijn vastgelegd dankzij de hogesnelheidscapaciteiten van de techniek, onthulde bolletjes die verschijnen en verdwijnen, en transformaties tussen volledig ongestructureerde en los gevouwen conformaties in segmenten tot 160 aminozuren lang.
Studies van met name het nucleoproteïne van het mazelenvirus hielpen niet alleen de vorm en afmetingen te identificeren, maar ook kenmerken van de orde-stoornis-overgangen in het gebied dat verantwoordelijk is voor moleculaire herkenning, waarmee virussen gastheerfactoren kunnen identificeren zodat ze zich kunnen voortplanten. Ze zouden ook grotere schaalstructuren van het fosfoproteïne van het virus kunnen bepalen die niet toegankelijk zijn voor nucleaire magnetische resonantie (die alleen een indicatie kan geven van afstanden tussen aminozuren die van elkaar gescheiden zijn door minder dan 2 nm). De onderzoekers suggereren dat de vorming van bepaalde compacte vormen die worden waargenomen, de weerstand tegen proteolyse kan verklaren - eiwitafbraak.
In hun verslag van het werk, de onderzoekers benadrukken dat, naast een krachtig hulpmiddel op zich, "Wanneer alle moleculaire kenmerken die door HS-AFM worden onthuld, worden gecombineerd met de gevouwen lokale structuur die wordt gegeven door NMR, de gecombineerde informatie maakt een kwantitatieve afbakening van de structurele en dynamische karakters van ontheemden mogelijk, op een meer realistische manier vergeleken met de afzonderlijk afgebeelde foto's, zoals aangetoond voor PNT [fosfoproteïne van het mazelenvirus]."
Atomaire krachtmicroscopie met hoge snelheid
Atoomkrachtmicroscopie werd in de jaren tachtig ontwikkeld en bracht de resolutie op atomaire schaal die werd bereikt door scanning tunneling microscopie (die in 1986 de Nobelprijs voor natuurkunde won) naar niet-geleidende monsters. Het werkt met behulp van een kleine cantilever met een punt op nanoschaal aan het einde, die ofwel het oppervlak voelt als een naald voor vinylplaten of erop tikt. Whether by adjusting the tip height or the resonant frequency of the tapping, the interactions between tip and surface provide a signal that can be used to generate an image.
While AFM images brought huge benefits to biological research, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
Intrinsically disordered proteins
The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.
 Hoe maak je een 3D-model van een koolstof-atoom
Hoe maak je een 3D-model van een koolstof-atoom  Vooruit of achteruit? Nieuwe routes voor protonen in water of methanol
Vooruit of achteruit? Nieuwe routes voor protonen in water of methanol Het ion vormen dat het universum heeft gemaakt
Het ion vormen dat het universum heeft gemaakt Silica-paradox:wetenschappers ontdekken schijnbaar onmogelijk materiaal
Silica-paradox:wetenschappers ontdekken schijnbaar onmogelijk materiaal Onderzoekers fabriceren transparant keramiek van hoge kwaliteit
Onderzoekers fabriceren transparant keramiek van hoge kwaliteit
Hoofdlijnen
- Wat zijn de niveaus van organisatie in de biologie?
- Wat is het Genotype voor de Roan-kleur?
- Wat een grote handtekening kan zeggen over je persoonlijkheid
- Epitheliale cellen: definitie, functie, typen en voorbeelden
- Neanderthalers waren eigenlijk prehistorische Picasso's
- Wat zijn de meest voorkomende oorzaken van uitsterven?
- Snelle papegaaien gefokt op roofdiervrije eilanden die met uitsterven worden bedreigd
- Gewassen evolueerden 10 millennia eerder dan gedacht
- Marmoset-baby's krijgen een boost van attente vaders
- Corrosie van oliepijpleidingen tegengaan door een schurk op nanoschaal te identificeren
- Immuuncellen weren koolstof-nano-indringers niet altijd af

- Simulatie toont aan dat het mogelijk is om H2O@C60 te verplaatsen met behulp van elektrische lading
- Natuurkundigen binden platen van één atoom met dezelfde kracht die gekko's gebruiken om muren te beklimmen
- Onderzoekers ontwikkelen veelzijdige optomechanische sensoren voor atoomkrachtmicroscopie
 Een tombe met uitzicht:egyptoloog herschept ervaring na de dood in VR
Een tombe met uitzicht:egyptoloog herschept ervaring na de dood in VR Decennialang dubieuze investeringen blootleggen
Decennialang dubieuze investeringen blootleggen Wetenschap zet nog een stap in het wissen van pijnlijke herinneringen
Wetenschap zet nog een stap in het wissen van pijnlijke herinneringen  NASA's Zon-waarnemende IRIS-missie
NASA's Zon-waarnemende IRIS-missie Studie toont geografische verschuiving in sociale mobiliteit in de VS
Studie toont geografische verschuiving in sociale mobiliteit in de VS Senator Russell ziet een UFO
Senator Russell ziet een UFO  Online intimidatie een probleem voor veel Amerikaanse tieners:enquête
Online intimidatie een probleem voor veel Amerikaanse tieners:enquête Mede-oprichter van Apple protesteert tegen Facebook door account af te sluiten
Mede-oprichter van Apple protesteert tegen Facebook door account af te sluiten
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

