
Wetenschap
Nieuwe beeldvormingsmethode onthult details over DNA op nanoschaal
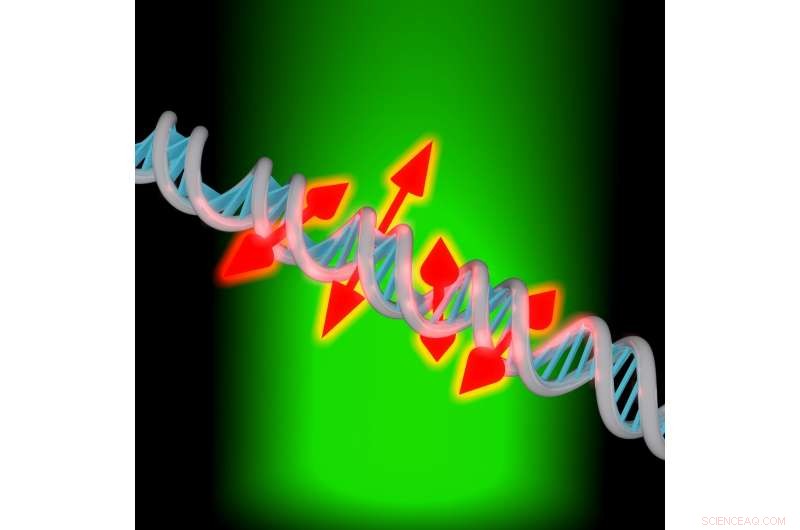
Met een nieuwe beeldvormingstechniek kunnen onderzoekers zowel de positie als de oriëntatie van afzonderlijke fluorescerende moleculen die aan DNA zijn bevestigd, in beeld brengen. Krediet:Maurice Y. Lee, Stanford universiteit
Onderzoekers hebben een nieuwe verbeterde DNA-beeldvormingstechniek ontwikkeld die de structuur van individuele DNA-strengen op nanoschaal kan onderzoeken. Omdat DNA aan de basis ligt van veel ziekteprocessen, de techniek kan wetenschappers helpen om belangrijke inzichten te krijgen in wat er mis gaat wanneer DNA beschadigd raakt of wanneer andere cellulaire processen de genexpressie beïnvloeden.
De nieuwe beeldvormingsmethode bouwt voort op een techniek die microscopie met één molecuul wordt genoemd door informatie toe te voegen over de oriëntatie en beweging van fluorescerende kleurstoffen die aan de DNA-streng zijn bevestigd.
W.E. Moerner, Stanford universiteit, VS, is de grondlegger van single-molecule spectroscopie, een doorbraakmethode uit 1989 waarmee wetenschappers voor het eerst afzonderlijke moleculen konden visualiseren met optische microscopie. Van de 2014 Nobelprijswinnaars voor optische microscopie voorbij de diffractielimiet (Moerner, Hel &Betzig), Moerner en Betzig gebruikten afzonderlijke moleculen om een dichte reeks moleculen op verschillende tijdstippen in beeld te brengen.
In het tijdschrift The Optical Society voor high impact research, optiek , het onderzoeksteam onder leiding van Moerner beschrijft hun nieuwe techniek en demonstreert deze door superresolutiebeelden en oriëntatiemetingen te verkrijgen voor duizenden enkele fluorescerende kleurstofmoleculen die aan DNA-strengen zijn bevestigd.
"Je kunt deze nieuwe metingen zien als kleine tweekoppige pijlen die de oriëntatie aangeven van de moleculen die langs de DNA-streng zijn bevestigd, " zei Moerner. "Deze oriëntatie-informatie rapporteert over de lokale structuur van de DNA-basen omdat ze het molecuul beperken. Als we deze oriëntatie-informatie niet hadden, zou de afbeelding slechts een plek zijn."
Meer informatie op nanoschaal toevoegen
Een DNA-streng is een zeer lange, maar smal touw, slechts enkele nanometers groot. Single-molecuul microscopie, samen met fluorescerende kleurstoffen die hechten aan DNA, kan worden gebruikt om deze kleine string beter te visualiseren. Tot nu, het was moeilijk te begrijpen hoe die kleurstoffen waren georiënteerd en onmogelijk om te weten of de fluorescerende kleurstof op een stijve of enigszins losse manier aan het DNA was gehecht.
Adam S. Backer, eerste auteur van het artikel, een vrij eenvoudige manier ontwikkeld om oriëntatie en rotatiedynamica te verkrijgen van duizenden afzonderlijke moleculen parallel. "Onze nieuwe beeldvormingstechniek onderzoekt hoe elk individueel kleurstofmolecuul dat het DNA labelt, is uitgelijnd ten opzichte van de veel grotere structuur van DNA, "zei Backer. "We meten ook hoe wankel elk van deze moleculen is, die ons kan vertellen of dit molecuul vastzit in een bepaalde uitlijning of dat het ronddraait in de loop van onze meetreeks."
De nieuwe techniek biedt meer gedetailleerde informatie dan de huidige zogenaamde "ensemble"-methoden, die de oriëntaties voor een groep moleculen gemiddeld, en het is veel sneller dan confocale microscopietechnieken, die één molecuul tegelijk analyseren. De nieuwe methode kan zelfs worden gebruikt voor moleculen die relatief zwak zijn.
Omdat de techniek informatie op nanoschaal geeft over het DNA zelf, het kan nuttig zijn voor het volgen van veranderingen in de DNA-conformatie of schade aan een bepaald gebied van het DNA, die zou verschijnen als veranderingen in de oriëntatie van kleurstofmoleculen. Het kan ook worden gebruikt om interacties tussen DNA en eiwitten te volgen, die veel cellulaire processen aansturen.
30, 000 oriëntaties van één molecuul
De onderzoekers testten de verbeterde DNA-beeldvormingstechniek door deze te gebruiken om een intercalerende kleurstof te analyseren; een soort fluorescerende kleurstof die in de gebieden tussen DNA-basen glijdt. In een typisch beeldvormend experiment, ze verwerven tot 300, 000 locaties met één molecuul en 30, 000 oriëntatiemetingen van één molecuul in iets meer dan 13 minuten. De analyse toonde aan dat de individuele kleurstofmoleculen loodrecht op de as van de DNA-streng waren georiënteerd en dat terwijl de moleculen de neiging hadden om in deze loodrechte richting te oriënteren, ze bewogen ook rond binnen een beperkte kegel.
De onderzoekers voerden vervolgens een vergelijkbare analyse uit met een ander type fluorescerende kleurstof die uit twee delen bestaat:een deel dat aan de zijkant van het DNA hecht en een fluorescerend deel dat is verbonden via een slappe ketting. De verbeterde DNA-beeldvormingstechniek detecteerde deze floppiness, waaruit blijkt dat de methode nuttig kan zijn om wetenschappers te helpen begrijpen, molecuul per molecuul, of verschillende labels zich mobiel of vast aan DNA hechten.
In de krant, de onderzoekers toonden een ruimtelijke resolutie van ongeveer 25 nanometer en oriëntatiemetingen van één molecuul met een nauwkeurigheid van ongeveer 5 graden. Ze maten ook de rotatiedynamiek, of slapheid, van enkelvoudige moleculen met een nauwkeurigheid van ongeveer 20 graden.
Hoe het werkt
Om informatie over de oriëntatie van één molecuul te verkrijgen, the researchers used a well-studied technique that adds an optical element called an electro-optic modulator to the single-molecule microscope. For each camera frame, this device changed the polarization of the laser light used to illuminate all the fluorescent dyes.
Since fluorescent dye molecules with orientations most closely aligned with the laser light's polarization will appear brightest, measuring the brightness of each molecule in each camera frame allowed the researchers to quantify orientation and rotational dynamics on a molecule-by-molecule basis. Molecules that switched between bright and dark in sequential frames were rigidly constrained at a particular orientation while those that appeared bright for sequential frames were not rigidly holding their orientation.
"If someone has a single-molecule microscope, they can perform our technique pretty easily by adding the electro-optic modulator, " said Backer. "We've used fairly standard tools in a slightly different way and analyzed the data in a new way to gain additional biological and physical insight."
Hoofdlijnen
- Een model van een menselijke cel bouwen
- Chromosomale afwijkingen: wat is het?, Typen en oorzaken
- Welke drie dingen helpen bloed door aderen te duwen?
- Wetenschappers identificeren verborgen genetische variatie die de evolutie helpt stimuleren
- Welke soort weefsel geeft de meeste tijd in interfase?
- Wat zijn de voordelen van prokaryoten?
- Meer bewijs dat Neanderthalers niet dom waren:ze maakten hun eigen touwtje
- Een fokprogramma voor natuurbehoud opzetten om de laatste saola te redden
- Huidige vee-injecties verhogen het risico op letsel, onderzoek vindt
- Wetenschappers ontdekken twee soorten bubbels met weinig elektronen in supervloeibaar helium
- De techniek van micronaalden kan snellere diagnoses van ernstige ziekten betekenen

- Onderzoekers creëren voorspelbare patronen van onvoorspelbare koolstofnanobuisjes

- Nieuwe grafeenoxide-biosensoren kunnen het onderzoek naar hiv- en kankermedicijnen versnellen

- Nieuwe methode gebruikt lichte en gouden nanodeeltjes voor zeer gerichte, niet-invasieve medicijnafgifte
 Werkloosheid dwingt meer mannen om door vrouwen gedomineerde banen op zich te nemen
Werkloosheid dwingt meer mannen om door vrouwen gedomineerde banen op zich te nemen Onderzoek suggereert dat het vergroten van de gemeenschapsband tussen politie en jonge zwarte mannen gewelddadige ontmoetingen kan verminderen
Onderzoek suggereert dat het vergroten van de gemeenschapsband tussen politie en jonge zwarte mannen gewelddadige ontmoetingen kan verminderen Nieuw ontdekt fossiel onthult dat oude pinguïns enorm waren
Nieuw ontdekt fossiel onthult dat oude pinguïns enorm waren  Tientallen doden bij uitbarsting vulkaan Nieuw-Zeeland
Tientallen doden bij uitbarsting vulkaan Nieuw-Zeeland Shhh! Hoe March Madness te streamen als de baas er niet is
Shhh! Hoe March Madness te streamen als de baas er niet is Biosensor-chip maakt zeer gevoelige eiwitanalyse mogelijk voor ziektediagnose
Biosensor-chip maakt zeer gevoelige eiwitanalyse mogelijk voor ziektediagnose Onderzoek toont effecten van Chinese echtscheidingswet op het welzijn van vrouwen aan
Onderzoek toont effecten van Chinese echtscheidingswet op het welzijn van vrouwen aan Rooknevel omhult Sydney terwijl de dreiging van bosbranden toeneemt
Rooknevel omhult Sydney terwijl de dreiging van bosbranden toeneemt
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com


