
Wetenschap
Beelden als eerste in hun soort kunnen helpen bij het gebruik van DNA om apparaten op nanoschaal te bouwen
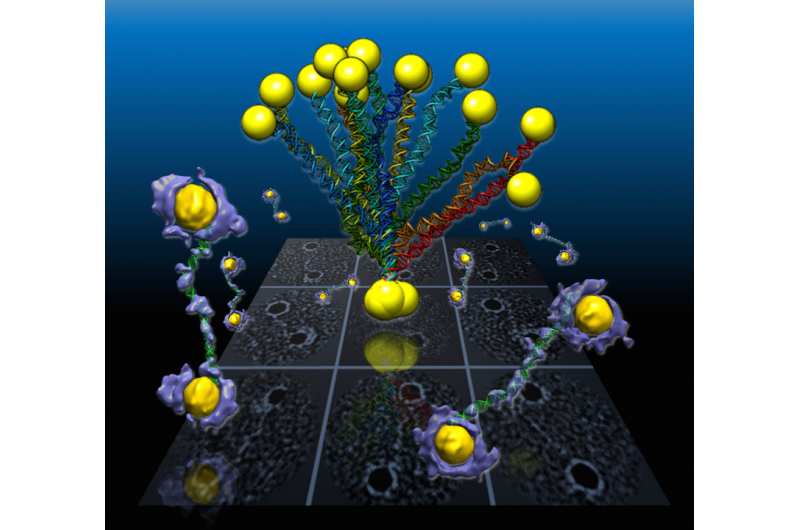
In een door Berkeley Lab geleid onderzoek flexibele dubbele helix DNA-segmenten verbonden met gouden nanodeeltjes worden onthuld uit de 3D-dichtheidskaarten (paars en geel) die zijn gereconstrueerd uit individuele monsters met behulp van een door Berkeley Lab ontwikkelde techniek genaamd individuele deeltjeselektronentomografie of IPET. Projecties van de structuren worden weergegeven in het achtergrondraster. Krediet:Berkeley Lab
Een internationaal team dat werkt bij het Lawrence Berkeley National Laboratory (Berkeley Lab) van het Department of Energy heeft de eerste 3D-beelden met hoge resolutie vastgelegd van individuele DNA-segmenten met dubbele helix die aan beide uiteinden zijn bevestigd aan gouden nanodeeltjes. De afbeeldingen geven details over de flexibele structuur van de DNA-segmenten, die verschijnen als springtouwen op nanoschaal.
Deze unieke beeldvorming, ontwikkeld door Berkeley Lab-wetenschappers, zou kunnen helpen bij het gebruik van DNA-segmenten als bouwstenen voor moleculaire apparaten die functioneren als medicijnafgiftesystemen op nanoschaal, markers voor biologisch onderzoek, en componenten voor computergeheugen en elektronische apparaten. Het zou ook kunnen leiden tot beelden van belangrijke ziekterelevante eiwitten die ongrijpbaar zijn gebleken voor andere beeldvormingstechnieken, en van het assemblageproces dat DNA vormt uit afzonderlijke, individuele strengen.
De vormen van de opgerolde DNA-strengen, die waren ingeklemd tussen polygoonvormige gouden nanodeeltjes, werden in 3D gereconstrueerd met behulp van een geavanceerde elektronenmicroscooptechniek in combinatie met een eiwitkleuringsproces en geavanceerde software die structurele details opleverde op een schaal van ongeveer 2 nanometer, of twee miljardsten van een meter.
"We hadden geen idee hoe het dubbelstrengs DNA eruit zou zien tussen de nanogouddeeltjes, " zei Bende "Gary" Ren, een Berkeley Lab-wetenschapper die het onderzoek leidde. "Dit is de eerste keer dat een individueel dubbelstrengs DNA-segment direct in 3D wordt gevisualiseerd, " zei hij. De resultaten werden gepubliceerd in de editie van 30 maart van Natuurcommunicatie .

Gang Ren (staand) en Lei Zhang namen deel aan een onderzoek bij Berkeley Lab's Molecular Foundry, dat 3D-reproducties produceerde van individuele monsters van DNA-segmenten met dubbele helix die aan gouden nanodeeltjes waren bevestigd. Krediet:Roy Kaltschmidt/Berkeley Lab
De door dit team ontwikkelde methode, zogenaamde individuele-deeltjeselektronentomografie (IPET), had eerder de 3D-structuur vastgelegd van een enkel eiwit dat een sleutelrol speelt in het cholesterolmetabolisme van de mens. Door 2D-beelden van hetzelfde object vanuit verschillende hoeken te pakken, de techniek stelt onderzoekers in staat om een 3D-beeld van dat object samen te stellen. Het team heeft de techniek ook gebruikt om de fluctuatie van een ander bekend flexibel eiwit te ontdekken, humaan immunoglobuline 1, die een rol speelt in ons immuunsysteem.
Voor deze laatste studie van DNA-nanostructuren, Ren gebruikte een elektronenstraal-studietechniek genaamd cryo-elektronenmicroscopie (cryo-EM) om bevroren DNA-nanogold-monsters te onderzoeken, en gebruikte IPET om 3D-beelden te reconstrueren van monsters die waren gekleurd met zouten van zware metalen. Het team gebruikte ook moleculaire simulatietools om de natuurlijke vormvariaties te testen, genaamd "conformaties, " in de monsters, en vergeleek deze gesimuleerde vormen met waarnemingen.
Ren legde uit dat de van nature flexibele dynamiek van monsters, als een man die met zijn armen zwaait, kan niet volledig worden gedetailleerd door een methode die een gemiddelde van veel waarnemingen gebruikt.
Een populaire manier om de structurele details op nanoschaal van delicate biologische monsters te bekijken, is ze in kristallen te vormen en ze te zappen met röntgenstralen, hoewel dit hun natuurlijke vorm niet behoudt en de DNA-nanogold-monsters in deze studie ongelooflijk uitdagend zijn om te kristalliseren. Andere veelgebruikte onderzoekstechnieken kunnen een verzameling van duizenden bijna identieke objecten vereisen, bekeken met een elektronenmicroscoop, een enkele samenstellen, gemiddelde 3D-structuur. Maar dit 3D-beeld geeft mogelijk niet voldoende de natuurlijke vormschommelingen van een bepaald object weer.
De monsters in het laatste experiment werden gevormd uit individuele polygoon gouden nanostructuren, met een diameter van ongeveer 5 nanometer, verbonden met enkele DNA-segmentstrengen met 84 basenparen. Basenparen zijn chemische basisbouwstenen die het DNA zijn structuur geven. Elk afzonderlijk DNA-segment en gouden nanodeeltje zijn van nature aan elkaar geritst met een partner om het dubbelstrengs DNA-segment te vormen met aan beide uiteinden een gouddeeltje.
De monsters werden snel ingevroren om hun structuur te behouden voor studie met cryo-EM-beeldvorming, en de afstand tussen de twee gouddeeltjes in individuele monsters varieerde van 20-30 nanometer op basis van verschillende vormen die in de DNA-segmenten werden waargenomen. Onderzoekers gebruikten voor dit onderzoek een cryo-elektronenmicroscoop van Berkeley Lab's Molecular Foundry.
Ze verzamelden een reeks gekantelde afbeeldingen van de gekleurde objecten, en reconstrueerde 14 kaarten met elektronendichtheid die de structuur van individuele monsters gedetailleerd beschreven met behulp van de IPET-techniek. Ze verzamelden een dozijn conformaties voor de monsters en ontdekten dat de variaties in de DNA-vorm consistent waren met die gemeten in de snel ingevroren cryo-EM-monsters. De vormen waren ook consistent met monsters die werden bestudeerd met behulp van andere op elektronen gebaseerde beeldvormings- en röntgenverstrooiingsmethoden, en met computersimulaties.
Terwijl de 3D-reconstructies de basisstructuur op nanoschaal van de monsters laten zien, Ren zei dat de volgende stap zal zijn om te werken aan het verbeteren van de resolutie tot op sub-nanometerschaal.
"Zelfs in deze huidige staat beginnen we 3D-structuren te zien met een resolutie van 1 tot 2 nanometer, " zei hij. "Door betere instrumentatie en verbeterde computationele algoritmen, het zou veelbelovend zijn om de resolutie naar het visualiseren van een enkele DNA-helix in een individueel eiwit te duwen."
De techniek, hij zei, heeft al belangstelling gewekt bij enkele vooraanstaande farmaceutische bedrijven en onderzoekers op het gebied van nanotechnologie, en zijn wetenschappelijke team heeft al tientallen gerelateerde onderzoeksprojecten in de pijplijn.
In toekomstige studies, onderzoekers zouden kunnen proberen de beeldresolutie te verbeteren voor complexe structuren die meer DNA-segmenten bevatten als een soort "DNA-origami, " zei Ren. Onderzoekers hopen moleculaire apparaten op nanoschaal te bouwen en beter te karakteriseren met behulp van DNA-segmenten die kunnen, bijvoorbeeld, medicijnen opslaan en afleveren aan gerichte gebieden in het lichaam.
"DNA is gemakkelijk te programmeren, synthetiseren en repliceren, dus het kan worden gebruikt als een speciaal materiaal om snel zelf te assembleren tot nanostructuren en om de werking van apparaten op moleculaire schaal te begeleiden, " zei hij. "Onze huidige studie is slechts een proof of concept voor het in beeld brengen van de structuren van dit soort moleculaire apparaten."
 Ultrasone trillingen dwingen een polymeer om een halfgeleider te zijn
Ultrasone trillingen dwingen een polymeer om een halfgeleider te zijn Hoe te bepalen of een atoom polair of niet-polair is?
Hoe te bepalen of een atoom polair of niet-polair is?  Darwin in een reageerbuis:Trio wint Nobelprijs voor het benutten van evolutie
Darwin in een reageerbuis:Trio wint Nobelprijs voor het benutten van evolutie Groovy foto-elektroden:hoe een gestructureerd oppervlak de prestaties drastisch kan verbeteren
Groovy foto-elektroden:hoe een gestructureerd oppervlak de prestaties drastisch kan verbeteren Poreuze zouten voor brandstofcellen
Poreuze zouten voor brandstofcellen
Hoofdlijnen
- Hoe glycolyse aan kinderen te leren
- DNA is digitaal geworden - wat kan er mis gaan?
- Gist kan worden ontwikkeld om eiwitgeneesmiddelen te maken
- Gemuteerd kikkergen stoot roofdieren af
- Zit moraliteit in de hersenen?
- Kan slapen me gelukkig maken?
- Nieuwe studie geeft aan hoe bevruchting veranderingen teweegbrengt in duizenden eiwitten in kikkereieren
- Een niet-verslavende opioïde pijnstiller zonder bijwerkingen
- Informatie over bloedvaten
- Ingenieurs ontwikkelen eerste methode voor aansturing nanomotoren

- Ultradun, volledig anorganische moleculaire nanodraden succesvol samengesteld
- Twee-fotonmicroscopie:nieuw onderzoek kan de kosten van krachtige microscooptechniek drastisch verlagen
- Geen goede trillingen meer nodig voor luidsprekers, omdat onderzoek grafeen aanmoedigt om te praten

- Technische onderzoekers vereenvoudigen het proces om de kleinste draden ter wereld te maken

 Waren op een splitsing in de weg:kiezen we wijken om te wonen, werken en spelen?
Waren op een splitsing in de weg:kiezen we wijken om te wonen, werken en spelen? Zijn de religieuze overtuigingen van wijlen Stephen Hawkings typerend voor Britse wetenschappers?
Zijn de religieuze overtuigingen van wijlen Stephen Hawkings typerend voor Britse wetenschappers? Studie onthult nieuwe supersymmetrie-anomalieën in superconforme kwantumveldentheorieën
Studie onthult nieuwe supersymmetrie-anomalieën in superconforme kwantumveldentheorieën Kunnen we 100 procent van onze energie uit hernieuwbare bronnen halen?
Kunnen we 100 procent van onze energie uit hernieuwbare bronnen halen? Biotische factoren in een overstroomd ecosysteem
Biotische factoren in een overstroomd ecosysteem  Autovrij Parijs? Het was al een droom in 1790
Autovrij Parijs? Het was al een droom in 1790 Noord-Amerika zorgt voor een wereldwijde explosie van olie- en gaspijpleidingen
Noord-Amerika zorgt voor een wereldwijde explosie van olie- en gaspijpleidingen Een nieuwe benadering van zeer efficiënte, luchtstabiele perovskiet zonnecellen
Een nieuwe benadering van zeer efficiënte, luchtstabiele perovskiet zonnecellen
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | German | Dutch | Danish | Swedish | Norway |

-
Wetenschap © https://nl.scienceaq.com

