
Wetenschap
Verbeterde statistische methoden voor high-throughput omics data-analyse
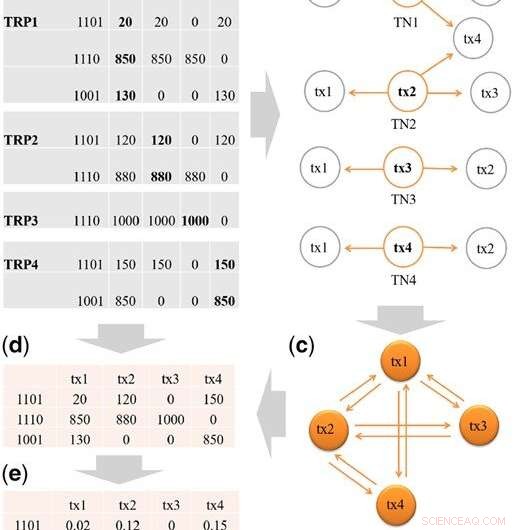
Stappen om de startontwerpmatrix X te construeren. (a) TRP's van tx1, tx2, tx3 en tx4, en de samenvatting van binaire bezettingspatronen uit de TRP's. Transcript tx5 passeert de filtering niet (H = 2,5%) en wordt uit TRP1 gefilterd. In elk binair patroon, cijfer 1 betekent dat er reads afkomstig zijn van een eqclass, en 0 anders. Bijvoorbeeld, er zijn drie eqclasses in TRP1:eqclass1, eqclass2 en eqclass3. Voor eq1 is het binaire patroon 1101, wat betekent drie transcripties, d.w.z. tx1, tx2 en tx4 hebben reads van eq1. (b) Transcript buren (TN's) voor tx1 tot tx4. (c) Illustratie van de constructie van transcriptiecluster (TC) van de TN's. We verzamelen eerst de TN's van tx1, tx2, tx3 en tx4, en voeg vervolgens de verbindingen tussen transcripties toe aan de TC. Bijvoorbeeld, van TN1, we voegen de verbinding van tx1-tx2 toe, tx1-tx3 en tx1-tx4. Uiteindelijk, een TC zou alle verbindingen bevatten tussen transcripties die exons delen. (d) De unieke reeks binaire patronen wordt bewaard, dus er blijven drie unieke patronen over:1101, 1001, 1110. Vervolgens vullen we de gelezen tellingen van elke bron TRP in. Bijvoorbeeld, voor patroon 1101, in TRP1 is de leestelling 20 voor tx1, in TRP2 is de leestelling 120 voor tx2 en in TRP4 is de leestelling 150 voor tx4. (e) De totale aflezingen van elk transcript in (d) zijn gestandaardiseerd om op te tellen tot 1 om de startontwerpmatrix X te creëren. Credit:DOI:10.1093/bioinformatics/btz640
High-throughput omics-technologie heeft een revolutie teweeggebracht in biologisch en biomedisch onderzoek en er zijn grote hoeveelheden omics-gegevens geproduceerd. Voor deze, Er zijn computerhulpmiddelen ontwikkeld om de omics-gegevens te beheren en analyseren en er zijn grote uitdagingen bij het verwerken en interpreteren van de omics-gegevens op de beste manier. Wenjiang Deng heeft gewerkt aan de ontwikkeling van nieuwe statistische methoden en algoritmen voor de analyse van omics-gegevens, met behulp van zowel gesimuleerde als echte kankergegevens om de methoden te testen.
Kun je een aantal resultaten in je scriptie beschrijven?
Ja, in mijn eerste studie, we identificeren verschillende genen die geassocieerd zijn met de overleving van neuroblastoompatiënten met een hoog risico, zegt Wenjiang Deng, doctoraat student aan de afdeling medische epidemiologie en biostatistiek, CBG. Neuroblastoom is de meest voorkomende en dodelijkste kanker bij jonge kinderen onder de vijf jaar. Wij zijn van mening dat onze bevindingen significant bewijs zullen leveren voor de behandeling en het beheer van patiënten. Onze resultaten kunnen ook zinvol zijn om de fysiologische mechanismen van de ziekte te begrijpen.
Hoe komt het dat je ervoor hebt gekozen om dit specifieke gebied te bestuderen?
We leven in het tijdperk van "big data, " en de high-throughput sequencing-gegevens zijn de overheersende "big data" in de biowetenschappen. Toen ik voor het eerst het concept van omics-gegevens hoorde, Ik stond versteld van het enorme volume en het grote potentieel in medisch onderzoek. Tegenwoordig is het vrij eenvoudig om sequentiegegevens te produceren, maar we hebben nog steeds efficiënte en nauwkeurige instrumenten nodig om ze te analyseren, dus besloot ik de ontwikkeling van algoritmen te bestuderen tijdens mijn tijd als Ph.D. student.
Wat ga je hierna doen?
Na mijn verdediging, Ik blijf nog een tijdje in het CBG om mijn manuscripten af te ronden. Ik ga dan naar Shenzhen, China, en gaan werken in een biotechnologiebedrijf dat nieuwe methoden wil ontwikkelen voor vroege diagnose van kanker. Ik hoop dat ons werk daar zal bijdragen aan de algehele gezondheid van de mens.
 De structuur van RNA ontdekken
De structuur van RNA ontdekken Colloïdale geleigenschappen onder de microscoop
Colloïdale geleigenschappen onder de microscoop Magnetische behandeling kan helpen bij het verwijderen van de smaak van wijnen
Magnetische behandeling kan helpen bij het verwijderen van de smaak van wijnen Nieuw algoritme helpt vergeten figuren onder Da Vinci-schilderij te ontdekken
Nieuw algoritme helpt vergeten figuren onder Da Vinci-schilderij te ontdekken Moleculair doelwit UNC45A is essentieel voor kanker, maar niet voor normale celproliferatie
Moleculair doelwit UNC45A is essentieel voor kanker, maar niet voor normale celproliferatie
 Cycloonfeiten voor kinderen
Cycloonfeiten voor kinderen Leider van de Marshalleilanden roept op om hulp bij klimaatverandering
Leider van de Marshalleilanden roept op om hulp bij klimaatverandering Nieuw rapport over klimaatverandering in de Sierra Nevada toont noodzaak voor menselijke aanpassing
Nieuw rapport over klimaatverandering in de Sierra Nevada toont noodzaak voor menselijke aanpassing Studie onthult toename van fotosynthese in het droge seizoen in het Amazone-regenwoud
Studie onthult toename van fotosynthese in het droge seizoen in het Amazone-regenwoud Onomkeerbare opwarming van de oceaan bedreigt de Filchner-Ronne Ice Shelf
Onomkeerbare opwarming van de oceaan bedreigt de Filchner-Ronne Ice Shelf
Hoofdlijnen
- Hagedis, schildpad tussen meer dan 100 nieuwe soorten gevonden in Mekong-regio
- Wat zijn de functies van mRNA & tRNA?
- Welke organen maken het vaatstelsel op?
- Hebben vogelgezang en menselijke spraak biologische wortels?
- Moeder-kind linkerzijde face-to-face voorkeur bleek zich uit te strekken tot walrussen en Indiase vliegende vossen
- Antibioticaresistentie:onderzoekers slagen erin resistentiegenen te blokkeren
- Openbare bronnen stimuleren de ontdekking van medicijnen en bieden inzicht in de eiwitfunctie
- Hoe water door planten beweegt
- Research Paper Onderwerpen in Biochemistry
- Aan banken gelieerde fondsen dragen bij aan de financiering van hun moederbank in tijden van crisis

- Mensachtige loopmechanica evolueerde vóór het geslacht Homo
- 530 miljoen jaar oud fossiel ziet eruit als oudste oog ter wereld studie suggereert:

- Dumpen vliegtuigen routinematig hun brandstof voordat ze landen?

- Echte patronen onderscheiden van eenvoudige menselijke misvattingen

 Wat doet ethanol in een DNA-extractie?
Wat doet ethanol in een DNA-extractie?  Uitbarsting van Okmok-vulkaan in Alaska gekoppeld aan periode van extreme kou in het oude Rome
Uitbarsting van Okmok-vulkaan in Alaska gekoppeld aan periode van extreme kou in het oude Rome Flash droogte verergert in 14 zuidelijke staten van de VS
Flash droogte verergert in 14 zuidelijke staten van de VS Zuivering van afvalwater kan leiden tot disbalans tussen stikstof en fosfor
Zuivering van afvalwater kan leiden tot disbalans tussen stikstof en fosfor Calcium:goed voor botten, goed voor cultureel behoud
Calcium:goed voor botten, goed voor cultureel behoud Statistieken onthullen nieuwe, nauwkeuriger inzicht in opwaartse mobiliteit tussen generaties
Statistieken onthullen nieuwe, nauwkeuriger inzicht in opwaartse mobiliteit tussen generaties NASA ziet Paul een overblijfsel van een lagedrukgebied worden
NASA ziet Paul een overblijfsel van een lagedrukgebied worden Wetenschapsprojecten om een model van een aardbevingsbestendig huis te maken
Wetenschapsprojecten om een model van een aardbevingsbestendig huis te maken
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

