
Wetenschap
Een webapplicatie om belangrijke informatie uit tijdschriftartikelen te halen

Een screenshot van de DIVE-website. Krediet:Gupta et al.
Academische papers bevatten vaak verslagen van nieuwe doorbraken en interessante theorieën met betrekking tot een verscheidenheid aan vakgebieden. Echter, de meeste van deze artikelen zijn geschreven in jargon en technische taal die alleen begrepen kan worden door lezers die bekend zijn met dat specifieke studiegebied.
Niet-deskundige lezers zijn dus doorgaans niet in staat om wetenschappelijke artikelen te begrijpen, tenzij ze zijn samengesteld en toegankelijker zijn gemaakt door derden die de concepten en ideeën die erin staan begrijpen. Met dit in gedachten, een team van onderzoekers van het Texas Advanced Computing Center aan de Universiteit van Texas in Austin (TACC), Oregon State University (OSU) en de American Society of Plant Biologists (ASPB) hebben een tool ontwikkeld die automatisch belangrijke zinnen en terminologie kan extraheren uit onderzoekspapers om bruikbare definities te geven en hun leesbaarheid te vergroten.
"Ons project is gemotiveerd door de noodzaak om de leesbaarheid van tijdschriftartikelen te verbeteren, "Weijia Xu, die het team leiden bij TACC, vertelde TechXplore. "Het is een gezamenlijke inspanning van biologische curatoren, tijdschriftuitgevers en computerwetenschappers gericht op het ontwikkelen van een webservice die auteurcuratie van belangrijke terminologie die in tijdschriftpublicaties wordt gebruikt, kan herkennen en mogelijk maken. De terminologie en woorden worden vervolgens aan het einde van het tijdschriftartikel toegevoegd om de toegankelijkheid voor lezers te vergroten."
Xu en zijn collega's ontwikkelden een uitbreidbaar raamwerk dat kan worden gebruikt om informatie uit documenten te extraheren. Vervolgens implementeerden ze dit raamwerk binnen een webservice genaamd DIVE (Domain Information Vocabulary Extraction), integratie met de publicatiepijplijn van tijdschriften van de ASPB. In tegenstelling tot bestaande tools voor het extraheren van domeininformatie, hun raamwerk combineert verschillende benaderingen, inclusief ontologie-geleide extractie, op regels gebaseerde extractie, natuurlijke taalverwerking (NLP) en deep learning-technieken.

Het door de onderzoekers voorgestelde architectuuroverzicht van het systeem. Krediet:Gupta et al.
"De resultaten die door verschillende modellen worden bereikt, worden vervolgens opgeslagen in een gecentraliseerde database, " legde Xu uit. "We hebben ook een webservice ontworpen waarmee gebruikers extractieresultaten kunnen beheren. De webservice is geïntegreerd met de productiepublicatiepijplijn bij ASPB."
Zodra de preview-versie van een tijdschriftartikel is ingediend en in de pijplijn van de ASPB komt, het manuscript wordt automatisch naar DIVE gestuurd, die het verwerkt en een URL produceert waarmee de auteur toegang heeft tot de verwerkingsresultaten van DIVE. De auteur van de paper wordt gevraagd om de verstrekte link te bezoeken en de geëxtraheerde informatie te bekijken voordat hij/zij de paper officieel kan indienen.
"De auteur moet de DIVE-site bezoeken om de extractieresultaten te bekijken en de definitieve goedkeuring te geven van de lijst met informatie die aan het einde van hun artikel moet worden opgenomen, Xu zei. "DIVE houdt ook auteurscorrecties bij om toekomstige extractietaken te verbeteren. Momenteel, geen enkele andere tijdschriftuitgever heeft een vergelijkbare aanpak gevolgd en geïntegreerd in hun publicatiepijplijn."
Tijdens zijn analyses en bij het extraheren van belangrijke gegevens uit documenten, het door de onderzoekers ontwikkelde raamwerk maakt gebruik van verschillende technieken. Hierdoor kan het meer informatie vastleggen dan andere methoden, zoals ABNER (A Biomedical Named Entity Recognizer), dat is een open source softwaretool voor moleculaire biologie text mining die alleen algemene termen (bijv. genen en eiwitten) kan extraheren. In tegenstelling tot DIVE, ABNER is alleen gebaseerd op voorwaardelijke willekeurige velden (CRF's), een statistische modelleringsmethode die veel wordt gebruikt in toepassingen voor patroonherkenning en machine learning.
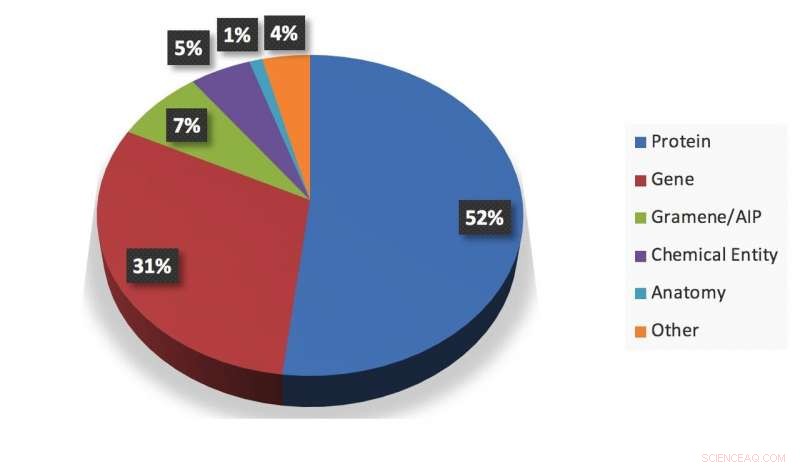
Een visuele samenvatting van een momentopname van informatie die door het systeem is geëxtraheerd. Krediet:Gupta et al.
"Een belangrijke bijdrage van ons project is dat het helpt bij het bouwen van datasets en modellen die auteurs onderzoeksinteresses kunnen afleiden uit hun publicaties, " zei Xu. "Ons project kan bredere gemeenschappen van biologische onderzoekers ten goede komen. Voor auteurs, de extracties en opname van de belangrijkste informatie kunnen de toegankelijkheid van hun artikelen vergroten."
Xu en zijn collega Amit Gupta evalueerden hun raamwerk en vergeleken de prestaties ervan met die van andere tools voor het extraheren van informatie, inclusief ABN. Uit hun bevindingen bleek dat het gebruik van meerdere benaderingen, inclusief diep leren, DIVE behaalt hogere precisiescores dan andere vooraf getrainde modellen die uitsluitend op CRF's zijn gebaseerd. interessant, het DIVE-framework kan ook continu worden bijgewerkt, omdat er op elk moment extra extractiemodellen aan kunnen worden toegevoegd.
Met de DIVE-webtoepassing kunnen niet-deskundige lezers niet alleen academische papers beter begrijpen, het kan hen ook helpen om papieren te identificeren die zijn afgestemd op hun interesses. onderzoekers, anderzijds, kan DIVE gebruiken om op de hoogte te blijven van bepaalde onderzoeksgebieden, evenals om te leren over nieuwe terminologie en trends met betrekking tot hun interessegebied. Eindelijk, de informatie die door de applicatie wordt gegenereerd, kan ook biologiecuratoren begeleiden bij hun beslissingen en gegevensverzamelingsprocessen.
"We zetten ons project voort door twee richtingen te verkennen, " zei Xu. "Aan de ene kant, we onderzoeken nieuwe methoden om op te nemen in onze informatie-extractiemodellen om de prestaties te verbeteren. Anderzijds, we proberen onze service ook uit te breiden door deze aan te bieden aan extra gebruikersgemeenschappen en tijdschriftuitgevers."
© 2019 Wetenschap X Netwerk
 Onderzoekers komen dichter bij auto's op waterstof
Onderzoekers komen dichter bij auto's op waterstof Collageen-nanofibrillen in weefsels van zoogdieren worden sterker bij inspanning
Collageen-nanofibrillen in weefsels van zoogdieren worden sterker bij inspanning Upcycling van sponsachtig plastic schuim van schoenen, matrassen en isolatie
Upcycling van sponsachtig plastic schuim van schoenen, matrassen en isolatie Schuifkracht:hoe goede materialen beter worden gemaakt?
Schuifkracht:hoe goede materialen beter worden gemaakt? Nieuwe beeldvormingstechniek onthult hoe mechanische schade begint op moleculaire schaal
Nieuwe beeldvormingstechniek onthult hoe mechanische schade begint op moleculaire schaal
Hoofdlijnen
- Een biologische oplossing voor het afvangen en recyclen van koolstof?
- Onderzoekers detecteren signalen van parasieten in mest van amfibieën
- Cephalization of Earthworms
- Waar bevinden lipiden zich in het lichaam?
- Zijn getrouwde mensen gelukkiger dan alleenstaanden?
- Graafwespen en hun chemie
- Geheugenverlies en hoofdtrauma
- Hoe geel en blauw groen maken bij papegaaien
- Wat zijn de stadia van de celcyclus?
 3D-bewegingsvolgsysteem kan visie voor autonome technologie stroomlijnen
3D-bewegingsvolgsysteem kan visie voor autonome technologie stroomlijnen Met 2D-materialen kunnen elektrische voertuigen 500 mijl afleggen op één lading
Met 2D-materialen kunnen elektrische voertuigen 500 mijl afleggen op één lading Natuurkundige onderzoekt de mogelijkheid van overblijfselen van een universum voorafgaand aan de oerknal
Natuurkundige onderzoekt de mogelijkheid van overblijfselen van een universum voorafgaand aan de oerknal Voorloper van hernieuwbare plastic zou de cellulose-biobrandstofindustrie kunnen laten groeien
Voorloper van hernieuwbare plastic zou de cellulose-biobrandstofindustrie kunnen laten groeien Orde en structuur vinden in de atomaire chaos waar materialen elkaar ontmoeten
Orde en structuur vinden in de atomaire chaos waar materialen elkaar ontmoeten Keplers vergeten ideeën over symmetrie helpen spiraalstelsels te verklaren zonder dat donkere materie nodig is
Keplers vergeten ideeën over symmetrie helpen spiraalstelsels te verklaren zonder dat donkere materie nodig is Gebrek aan garanties voor ziekteverlof brengt de gezondheid en economische veiligheid van landen in gevaar, studie vondsten
Gebrek aan garanties voor ziekteverlof brengt de gezondheid en economische veiligheid van landen in gevaar, studie vondsten WeWork klaagt Japans SoftBank aan voor terugtrekken uit deal
WeWork klaagt Japans SoftBank aan voor terugtrekken uit deal
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Dutch | Danish | Norway | Swedish | German |

-
Wetenschap © https://nl.scienceaq.com






