
Wetenschap
Nieuwe toolkit maakt simulaties van moleculaire dynamica toegankelijker
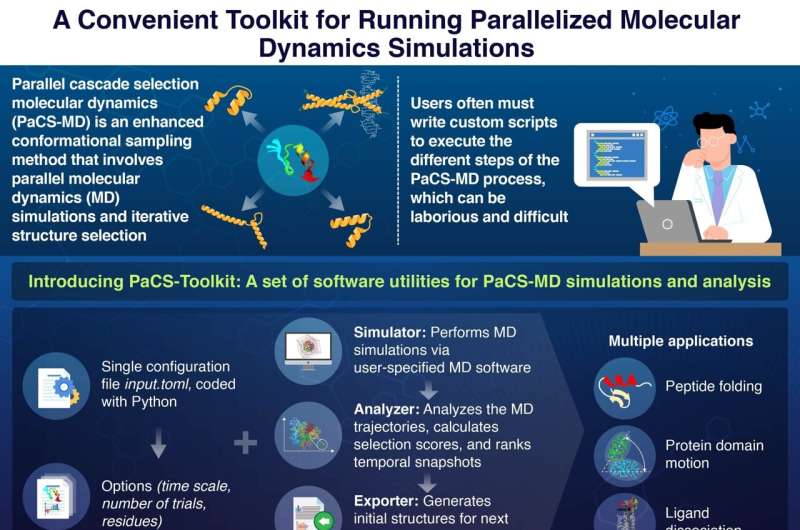
Moleculaire dynamica (MD)-simulaties zijn een krachtig hulpmiddel geworden op de steeds groeiende gebieden van de moleculaire biologie en de ontwikkeling van geneesmiddelen. Hoewel er veel MD-simulatietechnieken bestaan, is parallelle cascadeselectie MD (PaCS-MD) bijzonder nuttig bij het bestuderen van de vouwing (of "conformatie") van eiwitten of de interacties tussen eiwitten en liganden.
De kern van deze methode bestaat uit het parallel uitvoeren van meerdere MD-simulaties, waardoor tegelijkertijd verschillende mogelijke conformaties worden onderzocht. Met behulp van zorgvuldig opgestelde selectiecriteria kunnen veelbelovende conformaties automatisch worden gedetecteerd in ‘temporele snapshots’ en verder worden onderzocht. Deze strategie versnelt enorm de ontdekking van belangrijke moleculaire interacties en dynamische processen die wetenschappers helpen de functionele bewegingen van eiwitten te begrijpen.
Een van de hindernissen van PaCS-MD is echter dat gebruikers aangepaste scripts moeten schrijven om de gewenste MD-simulaties uit te voeren. In deze scripts moeten ze de initiële voorwaarden en doelfuncties specificeren, de te gebruiken MD-software selecteren, een snapshot-rangschikkingsprocedure implementeren en de initiële structuren voorbereiden voor de volgende simulatiecyclus. Dit proces, dat behoorlijk complex en foutgevoelig kan zijn, creëert een aanzienlijke toegangsbarrière voor wetenschappers die geïnteresseerd zijn in het gebruik van PaCS-MD.
Gelukkig is een team van onderzoekers van de School of Life Science and Technology van het Tokyo Institute of Technology, Japan, onlangs begonnen dit probleem aan te pakken. In hun nieuwste onderzoek, gepubliceerd in The Journal of Physical Chemistry B en onder leiding van professor Akio Kitao ontwikkelde het team een softwarepakket genaamd PaCS-Toolkit om PaCS-MD toegankelijker en gebruiksvriendelijker te maken.
Een opvallend voordeel van PaCS-Toolkit is dat het gehele simulatieproces via één configuratiebestand wordt opgezet. In dit bestand specificeren de gebruikers belangrijke parameters voor de simulaties, waaronder het type PaCS-MD, het aantal MD-simulaties dat parallel moet worden uitgevoerd en de eiwitresiduen of atomen die moeten worden bijgehouden als selectiecriteria voor parallelle vertakkingen.
P>PaCS-Toolkit neemt dit configuratiebestand, naast standaard MD-invoerbestanden, en voert PaCS-MD-simulaties uit volgens de gespecificeerde MD-software. Vermeldenswaard is dat, aangezien het pakket open software is, geschreven in Python, een populaire programmeertaal, gebruikers kunnen helpen PaCS-Toolkit te verbeteren en de functionaliteiten ervan uit te breiden.
"Onze toolkit behoudt de flexibiliteit zodat nieuwe functies, bibliotheken en MD-software kunnen worden toegevoegd door verantwoorde klassen in Python te introduceren. Gebruikers die in deze taal kunnen programmeren, moeten de code van PaCS-Toolkit kunnen aanpassen en indien nodig nieuwe methoden kunnen implementeren, " zegt prof. Kitao.
Een ander cruciaal voordeel van PaCS-Toolkit ligt in de optimalisatie en compatibiliteit met verschillende computeromgevingen, of het nu gaat om een reeks supercomputers met een message-passing interface (MPI), servers uitgerust met meerdere grafische kaarten (GPU's) of personal computers zoals laptops.
"PaCS-Toolkit maakt in grote mate gebruik van parallellisatie met behulp van MPI, GPU en het multiprocessingpakket van Python, waardoor optimalisatie van de rekentijd mogelijk is, afhankelijk van de beschikbare rekenbronnen", legt prof. Kitao uit.
Om het potentieel van hun toolkit te demonstreren, voerden de onderzoekers PaCS-MD-simulaties uit die waren afgestemd op drie verschillende toepassingen. Deze omvatten de vouwing van het mini-eiwit chignoline, de beweging van eiwitdomeinen in een SARS-CoV-2-enzym en de dissociatie van liganden van een belangrijke adenosinereceptor.
"Alles bij elkaar geven onze resultaten aan dat PaCS-Toolkit eenvoudig kan worden gebruikt om een grote verscheidenheid aan dynamieken voor verschillende soorten moleculaire systemen te simuleren", besluit prof. Kitao.
Deze toolkit zou kunnen helpen het ware potentieel van PaCS-MD-simulaties op verschillende gebieden te ontsluiten, waardoor geïnteresseerde onderzoekers licht kunnen werpen op complexe moleculaire processen en de ontdekking van geneesmiddelen kunnen versnellen.