Wetenschap
Computermodel voor het ontwerpen van eiwitsequenties die zijn geoptimaliseerd om te binden aan medicijndoelen
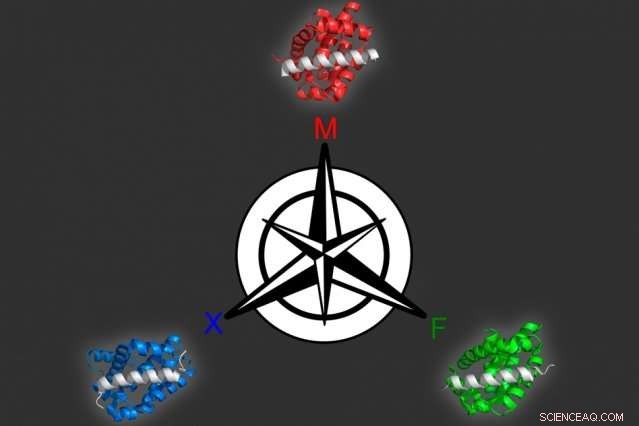
Met behulp van een computermodelleringsaanpak die ze ontwikkelden, MIT-biologen hebben drie verschillende eiwitten geïdentificeerd die selectief kunnen binden aan elk van drie vergelijkbare doelen, alle leden van de Bcl-2-familie van eiwitten. Krediet:Vincent Xue
Het ontwerpen van synthetische eiwitten die kunnen dienen als medicijnen tegen kanker of andere ziekten kan een moeizaam proces zijn:het gaat over het algemeen om het creëren van een bibliotheek van miljoenen eiwitten, vervolgens de bibliotheek screenen om eiwitten te vinden die het juiste doelwit binden.
MIT-biologen hebben nu een meer verfijnde aanpak bedacht waarbij ze computermodellering gebruiken om te voorspellen hoe verschillende eiwitsequenties zullen interageren met het doelwit. Deze strategie genereert een groter aantal kandidaten en biedt ook meer controle over een verscheidenheid aan eiwitkenmerken, zegt Amy Keating, een professor in biologie en biologische engineering en de leider van het onderzoeksteam.
"Onze methode geeft je een veel groter speelveld waar je oplossingen kunt selecteren die erg van elkaar verschillen en die verschillende sterke punten en verplichtingen zullen hebben, " zegt ze. "Onze hoop is dat we een breder scala aan mogelijke oplossingen kunnen bieden om de doorvoer van die eerste hits te vergroten tot bruikbare, functionele moleculen."
In een krant die verschijnt in de Proceedings van de National Academy of Sciences de week van 15 oktober, Keating en haar collega's gebruikten deze aanpak om verschillende peptiden te genereren die zich kunnen richten op verschillende leden van een eiwitfamilie genaamd Bcl-2, die de groei van kanker helpen stimuleren.
Recente promovendi Justin Jenson en Vincent Xue zijn de hoofdauteurs van het artikel. Andere auteurs zijn postdoc Tirtha Mandal, voormalig laboratoriumtechnicus Lindsey Stretz, en voormalig postdoc Lothar Reich.
Interacties modelleren
Eiwitgeneesmiddelen, ook wel biofarmaceutica genoemd, zijn een snelgroeiende klasse van geneesmiddelen die veelbelovend zijn voor de behandeling van een breed scala aan ziekten. De gebruikelijke methode voor het identificeren van dergelijke medicijnen is het screenen van miljoenen eiwitten, ofwel willekeurig gekozen of geselecteerd door het creëren van varianten van eiwitsequenties waarvan al is aangetoond dat ze veelbelovende kandidaten zijn. Dit omvat het manipuleren van virussen of gist om elk van de eiwitten te produceren, ze vervolgens bloot te stellen aan het doelwit om te zien welke het beste binden.
"Dat is de standaardbenadering:ofwel volledig willekeurig, of met enige voorkennis, ontwerp een bibliotheek van eiwitten, en ga dan vissen in de bibliotheek om de meest veelbelovende leden eruit te halen, ' zegt Keating.
Hoewel die methode goed werkt, het produceert meestal eiwitten die zijn geoptimaliseerd voor slechts één eigenschap:hoe goed het bindt aan het doelwit. Het biedt geen controle over andere functies die nuttig kunnen zijn, zoals eigenschappen die bijdragen aan het vermogen van een eiwit om in cellen te komen of de neiging om een immuunrespons uit te lokken.
"Er is geen voor de hand liggende manier om dat soort dingen te doen - specificeer een positief geladen peptide, bijvoorbeeld - met behulp van de brute force-bibliotheekscreening, ' zegt Keating.
Een ander wenselijk kenmerk is het vermogen om eiwitten te identificeren die stevig aan hun doelwit binden, maar niet aan vergelijkbare doelwitten, die ervoor zorgt dat medicijnen geen onbedoelde bijwerkingen hebben. De standaardaanpak stelt onderzoekers wel in staat om dit te doen, maar de experimenten worden omslachtiger, zegt Keating.
De nieuwe strategie omvat eerst het creëren van een computermodel dat peptidesequenties kan relateren aan hun bindingsaffiniteit voor het doeleiwit. Om dit model te maken, de onderzoekers kozen er eerst ongeveer 10, 000 peptiden, elk 23 aminozuren lang en spiraalvormig van structuur, en testten hun binding aan drie verschillende leden van de Bcl-2-familie. Ze kozen opzettelijk enkele sequenties waarvan ze al wisten dat ze goed zouden binden, plus anderen waarvan ze wisten dat ze dat niet zouden doen, dus het model kan gegevens bevatten over een reeks bindende vaardigheden.
Uit deze set gegevens het model kan een "landschap" produceren van hoe elke peptidesequentie interageert met elk doelwit. De onderzoekers kunnen het model vervolgens gebruiken om te voorspellen hoe andere sequenties zullen interageren met de doelen, en genereer peptiden die aan de gewenste criteria voldoen.
Met behulp van dit model, de onderzoekers produceerden 36 peptiden waarvan werd voorspeld dat ze aan één familielid stevig zouden binden, maar niet aan de andere twee. Alle kandidaten presteerden buitengewoon goed toen de onderzoekers ze experimenteel testten, dus probeerden ze een moeilijker probleem:het identificeren van eiwitten die aan twee van de leden binden, maar niet aan de derde. Veel van deze eiwitten waren ook succesvol.
"Deze benadering vertegenwoordigt een verschuiving van het stellen van een heel specifiek probleem en het ontwerpen van een experiment om het op te lossen, om wat werk vooraf te investeren om dit landschap te genereren van hoe sequentie gerelateerd is aan functie, het landschap vastleggen in een maquette, en dan in staat zijn om het naar believen te verkennen voor meerdere eigendommen, ' zegt Keating.
Sagar Khare, een universitair hoofddocent scheikunde en chemische biologie aan de Rutgers University, zegt dat de nieuwe aanpak indrukwekkend is in zijn vermogen om onderscheid te maken tussen nauw verwante eiwitdoelen.
"Selectiviteit van medicijnen is van cruciaal belang voor het minimaliseren van off-target effecten, en selectiviteit is vaak erg moeilijk te coderen omdat er zoveel op elkaar lijkende moleculaire concurrenten zijn die het medicijn ook los van het beoogde doelwit zullen binden. Dit werk laat zien hoe deze selectiviteit in het ontwerp zelf kan worden gecodeerd, " zegt Char, die niet bij het onderzoek betrokken was. "Toepassingen in de ontwikkeling van therapeutische peptiden zullen vrijwel zeker volgen."
selectieve medicijnen
Leden van de Bcl-2-eiwitfamilie spelen een belangrijke rol bij het reguleren van geprogrammeerde celdood. Ontregeling van deze eiwitten kan celdood remmen, tumoren helpen ongecontroleerd te groeien, zoveel farmaceutische bedrijven hebben gewerkt aan het ontwikkelen van medicijnen die gericht zijn op deze eiwitfamilie. Om dergelijke medicijnen effectief te laten zijn, het kan voor hen belangrijk zijn om zich op slechts één van de eiwitten te richten, omdat het verstoren van al deze schadelijke bijwerkingen in gezonde cellen kan veroorzaken.
"Vaak, kankercellen lijken slechts één of twee leden van de familie te gebruiken om celoverleving te bevorderen, " zegt Keating. "Over het algemeen, het wordt erkend dat het hebben van een panel van selectieve agenten veel beter zou zijn dan een ruw instrument dat ze allemaal uitschakelde."
De onderzoekers hebben patenten aangevraagd op de peptiden die ze in deze studie hebben geïdentificeerd, en ze hopen dat ze verder zullen worden getest als mogelijke medicijnen. Het lab van Keating werkt nu aan het toepassen van deze nieuwe modelleringsaanpak op andere eiwitdoelen. Dit soort modellering kan niet alleen nuttig zijn voor het ontwikkelen van potentiële geneesmiddelen, maar ook het genereren van eiwitten voor gebruik in landbouw- of energietoepassingen, ze zegt.
Dit verhaal is opnieuw gepubliceerd met dank aan MIT News (web.mit.edu/newsoffice/), een populaire site met nieuws over MIT-onderzoek, innovatie en onderwijs.
Hoofdlijnen
- Honden en wolven delen parasieten
- Hoe genenbanken werken
- Een natuurlijke schimmelstam kan olielekkages opruimen en de oliezanden van Albertas weer tot leven brengen
- Poema's zijn socialer dan eerder werd gedacht
- Prokaryotische versus eukaryotische cellen: overeenkomsten en verschillen
- E. Coli kweken in een petrischaal
- Onderzoekers kijken naar de fruitvlieg om het menselijk brein te begrijpen
- Transformatie, transductie en vervoeging: gentransfer in Prokaryotes
- Stille code van nucleotiden, geen aminozuren, bepaalt functies van vitale eiwitten
 Dagelijks gebruik van Helium Gas
Dagelijks gebruik van Helium Gas Tegenpolen trekken elektrokatalysator aan en inspireren
Tegenpolen trekken elektrokatalysator aan en inspireren Snelle inschakeling van een door astronomen waargenomen nucleaire transiënt
Snelle inschakeling van een door astronomen waargenomen nucleaire transiënt De ineenstorting van de Atlantische circulatie zou de Britse akkerbouw kunnen verminderen
De ineenstorting van de Atlantische circulatie zou de Britse akkerbouw kunnen verminderen Polymer twin:nieuw implantaat imiteert botstructuur
Polymer twin:nieuw implantaat imiteert botstructuur Legalisatie van marihuana – een zeldzaam probleem waarbij vrouwen conservatiever zijn dan mannen
Legalisatie van marihuana – een zeldzaam probleem waarbij vrouwen conservatiever zijn dan mannen Deponeren van ijzersoorten in ZSM-5 om cyclohexaan te oxideren tot cyclohexanon
Deponeren van ijzersoorten in ZSM-5 om cyclohexaan te oxideren tot cyclohexanon Nieuwe polymeerfilm haalt energie uit waterdamp, kan nano-elektronische apparaten van stroom voorzien (met video)
Nieuwe polymeerfilm haalt energie uit waterdamp, kan nano-elektronische apparaten van stroom voorzien (met video)
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com