Wetenschap
Nauwkeuriger lezen van RNA-modificaties in een zakformaatapparaat
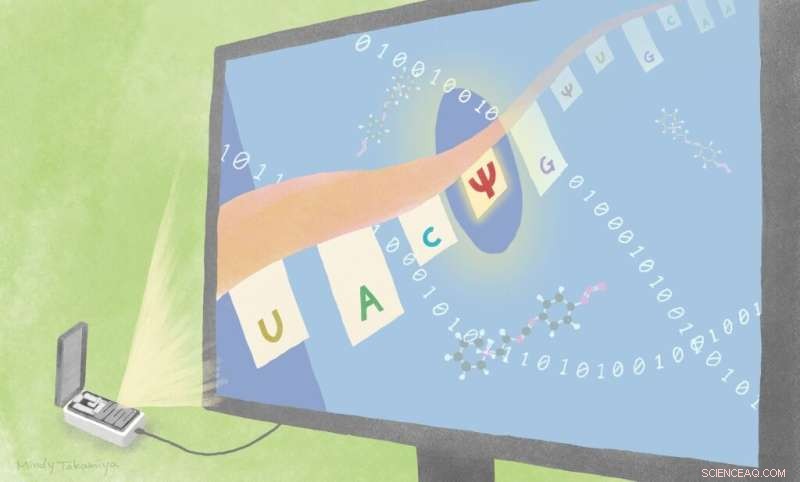
Wetenschappers hebben twee nieuwe manieren gevonden om substituties van uracil naar pseudouridine te identificeren, door bestaande sequencing-technologie te combineren met verschillende benaderingen. Een daarvan is met op algoritmen gebaseerde berekening en een is met een chemische sonde CMC. Krediet:Mindy Takamiya/Kyoto University iCeMS
Twee nieuwe benaderingen kunnen wetenschappers helpen om bestaande sequencing-technologie te gebruiken om RNA-veranderingen die van invloed zijn op hoe genetische code wordt gelezen, beter te onderscheiden.
Wetenschappers van de Universiteit van Kyoto komen steeds dichter bij het vinden van manieren om veranderingen in RNA-sequenties te identificeren die de eiwitvorming beïnvloeden en ziekten kunnen veroorzaken. Hun aanpak, gepubliceerd in het tijdschrift Genomics , maakt gebruik van waarschijnlijkheidsalgoritmen samen met een reeds beschikbaar, zakformaat sequencing-apparaat.
"Modificaties die in alle soorten biologisch RNA worden aangetroffen, beïnvloeden de genregulatie, die uiteindelijk bepaalt hoe verschillende cellen in ons lichaam functioneren", legt Ganesh Pandian Namasivayam van het Institute for Integrated Cell-Material Science (WPI-iCeMS) van de Universiteit van Kyoto uit. "Afwijkingen in deze modificaties kunnen leiden tot ernstige ziekten, zoals diabetes, neurologische ontwikkelingsstoornissen en kanker. Weten hoe en waar deze RNA-modificaties zijn, is vanuit klinisch oogpunt van het grootste belang", voegt Soundhar Ramaswamy, de eerste auteur van de studie, toe.
Er zijn al manieren om RNA-modificaties te identificeren, maar deze zijn onvoldoende. Biofysische benaderingen zoals chromatografie en massaspectrometrie kunnen slechts kleine hoeveelheden RNA tegelijk verwerken. Sequentiemethoden met hoge doorvoer, die grote hoeveelheden RNA kunnen verwerken, vereisen een moeizame monstervoorbereiding, kunnen niet tegelijkertijd meerdere modificaties in kaart brengen en zijn foutgevoelig.
Namasivayam, Hiroshi Sugiyama en collega's van de Universiteit van Kyoto hebben twee benaderingen getest en gevonden die relatief succesvol onderscheid kunnen maken tussen een bekende en overvloedige RNA-modificatie waarbij de nucleotidebase uracil wordt vervangen door een andere genaamd pseudouridine.
Net als DNA wordt RNA gevormd uit een streng van verschillende combinaties van vier verschillende nucleotidebasen:uracil, cytosine, adenine en guanine. Hoe deze basen zijn gerangschikt, bepaalt de code die aangeeft welk eiwit moet worden gemaakt. Wanneer pseudouridine uracil in de RNA-ruggengraat vervangt, kan dit leiden tot een verhoogde eiwitproductie of tot het veranderen van de code van een code die de onderbreking van de informatievertaling aangeeft naar een code die aminozuurvorming aangeeft.
De aanpak van het team omvat het gebruik van een reeds beschikbaar platform voor directe RNA-sequencing, ontwikkeld door Oxford Nanopore Technologies. In dit platform gaan RNA-strengen door kleine poriën in een membraan. Verstoringen worden veroorzaakt in de stroom die door het membraan beweegt, afhankelijk van de volgorde van de verschillende RNA-basen. Hierdoor kunnen wetenschappers de reeks "lezen". Maar wetenschappers die deze benadering gebruiken, vinden het vaak moeilijk om verschillende soorten modificaties van elkaar te onderscheiden.
Shubham Mishra, een gezamenlijke eerste auteur van deze studie, ontwikkelde algoritmen om een hoge waarschijnlijkheid van het bestaan van een pseudouridine-substitutie te identificeren in vergelijking met de mogelijkheid dat het een ander soort baseverandering was.
Een van hun strategieën vergelijkt korte RNA-runs van vijf nucleotidebasen waarin uracil, pseudouridine of cytosine aan weerszijden zijn omgeven door dezelfde basen. De metingen gaan vervolgens door algoritmen die de waarschijnlijkheid berekenen dat de middelste basis een van de drie is. Ze gebruikten hun strategie, genaamd Indo-Compare (Indo-C), op gemanipuleerde RNA-sequenties en vervolgens op gist en menselijk RNA en ontdekten dat het goed was in het onderscheiden van de pseudouridine-substituties van de andere.
Ze waren ook in staat om pseudouridine-substituties te identificeren door een chemische sonde te mengen met RNA-monsters, die zich er vervolgens selectief aan hechten. Dit veranderde de sequentielezingen op een manier die de wijziging identificeert.
"We geloven dat ons werk de op nanopore-sequencing gebaseerde methoden minder bewerkelijk zal maken voor het detecteren van RNA-modificaties en beter in staat zal zijn om de effecten van deze modificaties op ontwikkeling en ziekte te karakteriseren", zegt Namasivayam.
Het team wil vervolgens het gebruik van beide benaderingen samen optimaliseren om RNA- en DNA-modificaties nauwkeuriger te identificeren. Dit houdt in dat er nieuwe chemische sondes worden gefabriceerd die overeenkomen met specifieke veranderingen. Ze zijn ook van plan om geavanceerde algoritmen voor machinaal leren verder te ontwikkelen die een aanvulling vormen op op chemische sondes gebaseerde directe RNA-sequencing-benaderingen. + Verder verkennen
Meerdere RNA-basemodificaties tegelijk sequencen:een nieuw tijdperk van RNA-biologie
 Naarmate het klimaat verandert, planten zuigen mogelijk niet snel genoeg koolstof uit de lucht
Naarmate het klimaat verandert, planten zuigen mogelijk niet snel genoeg koolstof uit de lucht Nat en wild:er is veel water in 's werelds meest explosieve vulkaan
Nat en wild:er is veel water in 's werelds meest explosieve vulkaan Mexico Stad, uiterst kwetsbaar voor aardbevingsdreigingen
Mexico Stad, uiterst kwetsbaar voor aardbevingsdreigingen Dierlijke aanpassingen rond vulkanen
Dierlijke aanpassingen rond vulkanen  Lichtvervuiling:de donkere kant van het aanhouden van de lichten
Lichtvervuiling:de donkere kant van het aanhouden van de lichten
Hoofdlijnen
- Wat is een eencellige eukaryoot?
- Wat is Embryo Cloning?
- Herstel van verlaten landbouwgrond in het Murray-Darling Basin
- Een afweermechanisme om darmwormen te doden
- Nieuwe studie geeft aan hoe bevruchting veranderingen teweegbrengt in duizenden eiwitten in kikkereieren
- Voorbeelden van een recessief allel
- Meerdere stressoren kunnen interactief leiden tot verslechtering van aquatische ecosystemen
- Droogte geen dingo's achter het uitsterven van tijgers op het vasteland van Australië:studie
- Duizenden burgerwetenschappers helpen onderzoekers kelpbossen in kaart te brengen
 Wat is een bereidingsapotheek?
Wat is een bereidingsapotheek?  Een model om de impact van DDoS-aanvallen te bepalen met behulp van Twitter-gegevens
Een model om de impact van DDoS-aanvallen te bepalen met behulp van Twitter-gegevens Hoe het periodiek systeem voor kinderen
Hoe het periodiek systeem voor kinderen Biologische landbouw kan helpen de wereld te voeden, maar alleen als we minder vlees eten en stoppen met het verspillen van voedsel
Biologische landbouw kan helpen de wereld te voeden, maar alleen als we minder vlees eten en stoppen met het verspillen van voedsel De jacht op de ruimte begint terwijl de Binar-1-missie van West-Australië de volgende grote sprong maakt
De jacht op de ruimte begint terwijl de Binar-1-missie van West-Australië de volgende grote sprong maakt Vier lijken en tien ton afval verzameld op Everest
Vier lijken en tien ton afval verzameld op Everest Airbus houdt zich aan leveringsdoelen ondanks motorvertragingen
Airbus houdt zich aan leveringsdoelen ondanks motorvertragingen Studie:Met Twitter, ras van de boodschapper is belangrijk
Studie:Met Twitter, ras van de boodschapper is belangrijk
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com