
Wetenschap
Op AI gebaseerde screeningmethode zou de snelheid van nieuwe geneesmiddelenontdekking kunnen verhogen
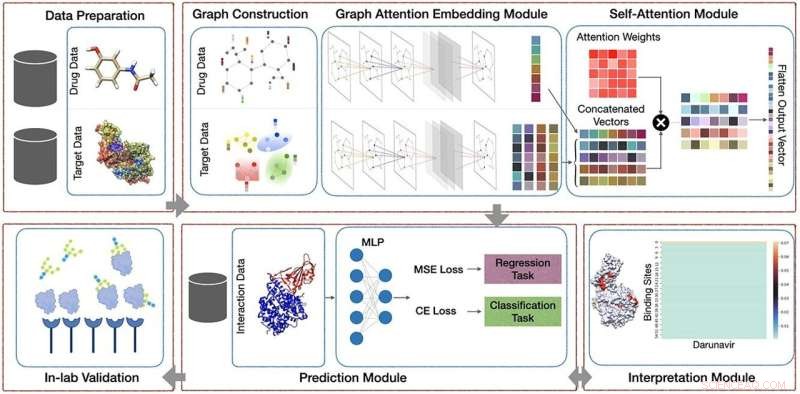
Ons voorgestelde raamwerk omvat vijf hoofdmodules:(1) Preprocessing-module die bestaat uit het vinden van de bindingsplaatsen van eiwitten; (2) AttentionSiteDTI deep learning-module, waar we grafiekrepresentaties construeren van de SMILE- en eiwitbindingsplaatsen van liganden, en we creëren een convolutioneel neuraal netwerk voor grafieken, gewapend met een aandachtspoolmechanisme om leerbare inbeddingen uit grafieken te extraheren, evenals een zelf- aandachtsmechanisme om de relatie tussen liganden en bindingsplaatsen van eiwitten te leren; (3) Voorspellingsmodule om onbekende interactie in een geneesmiddel-doelwitpaar te voorspellen, die zowel classificatie- als regressietaken kan aanpakken; (4) Interpretatiemodule om een dieper begrip te verschaffen van welke bindingsplaatsen van een doeleiwit waarschijnlijker zijn om te binden met een bepaald ligand. (5) In-lab validaties, waarbij we onze computationeel voorspelde resultaten vergelijken met experimenteel waargenomen (gemeten) interacties tussen geneesmiddelen in het laboratorium om het praktische potentieel van ons voorgestelde model te testen en te valideren. Krediet:Briefings in bio-informatica (2022). DOI:10.1093/bib/bbac272
Het ontwikkelen van levensreddende medicijnen kan miljarden dollars en tientallen jaren kosten, maar onderzoekers van de University of Central Florida willen dit proces versnellen met een nieuw op kunstmatige intelligentie gebaseerd screeningproces voor geneesmiddelen dat ze hebben ontwikkeld.
Met behulp van een methode die interacties tussen geneesmiddelen en doeleiwitten modelleert met behulp van natuurlijke taalverwerkingstechnieken, bereikten de onderzoekers een nauwkeurigheid tot 97% bij het identificeren van veelbelovende kandidaat-geneesmiddelen. De resultaten zijn onlangs gepubliceerd in het tijdschrift Briefings in Bioinformatics .
De techniek vertegenwoordigt interacties tussen geneesmiddelen en eiwitten door middel van woorden voor elke eiwitbindingsplaats en maakt gebruik van deep learning om de kenmerken te extraheren die de complexe interacties tussen de twee bepalen.
"Nu AI steeds meer beschikbaar komt, is dit iets geworden dat AI kan aanpakken", zegt co-auteur Ozlem Garibay, een assistent-professor aan de afdeling Industrial Engineering and Management Systems van UCF. "Je kunt zoveel variaties van eiwitten en geneesmiddelinteracties uitproberen en ontdekken welke meer kans hebben om te binden of niet."
Het model dat ze hebben ontwikkeld, bekend als AttentionSiteDTI, is het eerste dat interpreteerbaar is met behulp van de taal van eiwitbindingsplaatsen.
Het werk is belangrijk omdat het medicijnontwerpers zal helpen bij het identificeren van kritieke eiwitbindingsplaatsen samen met hun functionele eigenschappen, wat essentieel is om te bepalen of een medicijn effectief zal zijn.
De onderzoekers hebben dit bereikt door een zelfaandachtsmechanisme te bedenken waardoor het model leert welke delen van het eiwit interageren met de medicijnverbindingen, terwijl het state-of-the-art voorspellingsprestaties bereikt.
Het zelfaandachtsvermogen van het mechanisme werkt door selectief te focussen op de meest relevante delen van het eiwit.
De onderzoekers valideerden hun model met behulp van in-lab-experimenten die bindingsinteracties tussen verbindingen en eiwitten maten en vergeleken de resultaten met de resultaten die hun model rekenkundig voorspeld had. Omdat medicijnen voor de behandeling van COVID nog steeds interessant zijn, omvatten de experimenten ook het testen en valideren van medicijnverbindingen die zouden binden aan een spike-eiwit van het SARS-CoV2-virus.
Garibay zegt dat de grote overeenkomst tussen de laboratoriumresultaten en de computationele voorspellingen het potentieel illustreert van AttentionSiteDTI om potentieel effectieve medicijnverbindingen vooraf te screenen en de verkenning van nieuwe medicijnen en de herbestemming van bestaande medicijnen te versnellen.
"Dit high-impact onderzoek was alleen mogelijk dankzij interdisciplinaire samenwerking tussen materiaaltechnologie en AI/ML en computerwetenschappers om COVID-gerelateerde ontdekkingen aan te pakken", zegt Sudipta Seal, co-auteur van de studie en voorzitter van UCF's Department of Materials Science and Engineering.
Mehdi Yazdani-Jahromi, een doctoraalstudent aan het UCF's College of Engineering and Computer Science en de hoofdauteur van de studie, zegt dat het werk een nieuwe richting introduceert in de pre-screening van geneesmiddelen.
"Hierdoor kunnen onderzoekers AI gebruiken om medicijnen nauwkeuriger te identificeren om snel te reageren op nieuwe ziekten", zegt Yazdani-Jahromi. "Deze methode stelt de onderzoekers ook in staat om de beste bindingsplaats van het eiwit van een virus te identificeren om zich op te concentreren bij het ontwerpen van geneesmiddelen."
"De volgende stap van ons onderzoek wordt het ontwerpen van nieuwe medicijnen met behulp van de kracht van AI", zegt hij. "Dit kan natuurlijk de volgende stap zijn om voorbereid te zijn op een pandemie." + Verder verkennen
Onderzoekers identificeren nieuwe medicijnen met behulp van interpreteerbare deep learning-voorspellingen
 Een nieuwe procedure voor het verkrijgen van een goedkope, ultrahard materiaal dat bestand is tegen radioactiviteit
Een nieuwe procedure voor het verkrijgen van een goedkope, ultrahard materiaal dat bestand is tegen radioactiviteit Chemici creëren nieuwe route naar PHA's:natuurlijk afbreekbare bioplastics
Chemici creëren nieuwe route naar PHA's:natuurlijk afbreekbare bioplastics Evaporatie-experiment voor kinderen bekijken
Evaporatie-experiment voor kinderen bekijken Chocolade, thee of koffie met zink kan oxidatieve stress verminderen
Chocolade, thee of koffie met zink kan oxidatieve stress verminderen Kunnen huizen van de toekomst gemaakt worden door bacteriën?
Kunnen huizen van de toekomst gemaakt worden door bacteriën?
 Amerikaanse houtindustrie verlamd door dubbele klap van handelsoorlog en COVID-19
Amerikaanse houtindustrie verlamd door dubbele klap van handelsoorlog en COVID-19 Wat de aardbevingswetenschap ons kan vertellen over de kernproef van Noord-Korea?
Wat de aardbevingswetenschap ons kan vertellen over de kernproef van Noord-Korea? Elk gebouw laten meetellen bij het behalen van de emissiedoelstellingen van Australië
Elk gebouw laten meetellen bij het behalen van de emissiedoelstellingen van Australië Heeft Salt Affect Photosynthesis?
Heeft Salt Affect Photosynthesis?  Wat is rivierafvoer?
Wat is rivierafvoer?
Hoofdlijnen
- Is het tijd dat ons begrip van evolutie evolueert?
- Warme wateren in het noordwesten trekken paaiende vissen naar het noorden
- Wat gebeurt er wanneer glucose een cel binnengaat?
- Wat zijn de biomoleculen van ribosomen?
- Wat gebruiken chloroplasten om glucose te maken?
- De evolutie van slijm:hoe zijn we aan al dit slijm gekomen?
- Hoe Spaced Repetition examen tijd een fluitje van een cent kan maken
- Wat breekt een dubbele helix van DNA af?
- De effecten van temperatuur op enzymactiviteit en biologie
 Met behulp van twee CRISPR-enzymen, een COVID-diagnose in slechts 20 minuten
Met behulp van twee CRISPR-enzymen, een COVID-diagnose in slechts 20 minuten Er bestaan zelfs genderverschillen tussen de loonverwachtingen van universiteitsstudenten
Er bestaan zelfs genderverschillen tussen de loonverwachtingen van universiteitsstudenten Hoe beïnvloedt chloor de ozonlaag?
Hoe beïnvloedt chloor de ozonlaag?  Röntgenwaarnemingen onthullen nieuwe details over de zonne-type ster HD 209458
Röntgenwaarnemingen onthullen nieuwe details over de zonne-type ster HD 209458 Wat veroorzaakt een spijker in roest?
Wat veroorzaakt een spijker in roest?  Welk klimaat is ingesloten en krijgt weinig neerslag?
Welk klimaat is ingesloten en krijgt weinig neerslag?  Kale superaarden bieden aanwijzingen voor de evolutie van hete atmosferen
Kale superaarden bieden aanwijzingen voor de evolutie van hete atmosferen Expertise in wetenschappen en de beslissing van wat publiceerbaar is
Expertise in wetenschappen en de beslissing van wat publiceerbaar is
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com






