
Wetenschap
Een nieuwe bioimaging-methode voor het versnellen en vereenvoudigen van de identificatie van chemicaliën in weefsels
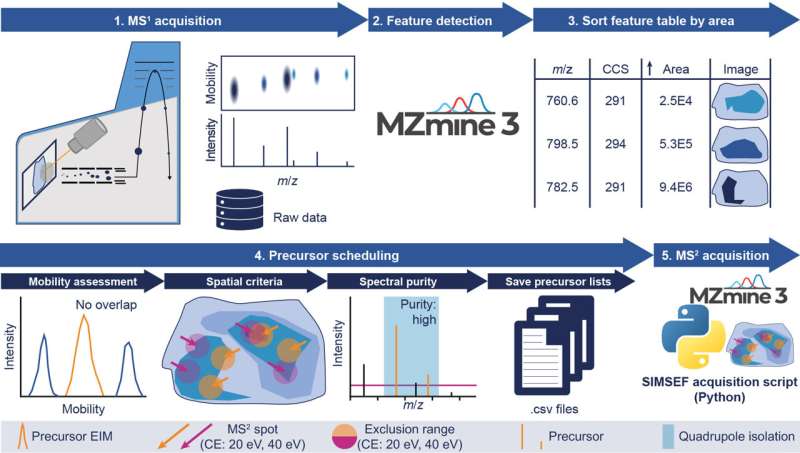
Wetenschappers verbonden aan het internationale softwareproject MZmine, geleid door Dr. Robin Schmid en Dr. Tomáš Pluskal van het Instituut voor Organische Chemie en Biochemie van de Tsjechische Academie van Wetenschappen, hebben een nieuw stuk software bedacht dat het proces aanzienlijk versnelt en vereenvoudigt. de identificatie van chemicaliën in weefsels.
Het stelt onderzoekers in staat om met vertrouwen chemicaliën te herkennen en hun verspreiding in organen te visualiseren. Vergeleken met bestaande workflows vereist de nieuwe pijplijn minder werk in het laboratorium om vergelijkbare inzichten te krijgen in de chemie die bijvoorbeeld plaatsvindt in tumoren en ontstekingslaesies. Het artikel waarin deze software aan de mondiale wetenschappelijke gemeenschap wordt gepresenteerd, is zojuist gepubliceerd in het tijdschrift Nature Communications .
Bij IOCB Praag is Dr. Robin Schmid vooral betrokken bij de ontwikkeling van de software SIMSEF (spatial ion Mobility-scheduled exhaustive fragmentation), samen met zijn collega's uit de groep van Prof. Dr. Karst aan de Universiteit van Münster, Duitsland, waar hij eerder werkte. gebaseerd.
Het werk wordt gedaan in samenwerking met de ontwikkelaars van de ultramoderne massaspectrometer timsTOF fleX van Bruker Daltonics, die de samenstelling van moleculen op hoog niveau kan ophelderen door de mobiliteit van ionen te meten.
“Tot nu toe konden wetenschappers met een massaspectrometer zien dat dit de formule was van het molecuul waar ze naar keken, maar toen ze vervolgens naar de database keken en deze stof probeerden te identificeren, was het erg moeilijk. De reden is dat biologische monsters kunnen talloze verschillende lipiden en combinaties van moleculen bevatten die vaak biologisch heel verschillend zijn."
“Dankzij het nieuwe algoritme is het nu mogelijk om in het molecuul te kijken, te leren waaruit het bestaat en zelfs afbeeldingen met elkaar te vergelijken”, legt Robin Schmid uit. Dit kan belangrijke informatie opleveren, bijvoorbeeld dat een specifiek deel van de hersenen een ander type lipide bevat dan een ander deel van dit essentiële orgaan en dat dit specifieke lipide in ander weefsel afwezig is.
Het SIMSEF-algoritme is ontwikkeld in samenwerking met medische experts van Duitse en Zwitserse universiteiten. Van cruciaal belang voor artsen is dat de detectie van klinische biomarkers voor diagnostische doeleinden ook aanzienlijk wordt versneld. Heel snel leren of ze bijvoorbeeld te maken hebben met een lipide of metaboliet die alleen in kankerweefsel voorkomt, speelt een cruciale rol bij de beslissing over verdere behandeling. Onnodig te zeggen dat dit grote gevolgen heeft voor de uitkomst.
Het nieuwe algoritme maakt deel uit van het open-source softwarepakket MZmine, dat experts wereldwijd sinds 2005 helpt bij het analyseren van massaspectrometriegegevens. Steffen Heuckeroth van het MZmine-team en het Instituut voor Anorganische en Analytische Chemie van de Universiteit van Münster was de hoofdontwikkelaar van het SIMSEF-algoritme en is de eerste auteur van het artikel dat nu is gepubliceerd in Nature Communications .
Hij voegt eraan toe:"Het artikel in kwestie bevat geen specifieke ontdekking op het gebied van de biologie. Dankzij onze nieuwe methode kunnen veel wetenschappers hun werk echter verbeteren en versnellen, wat ruimte opent voor nieuwe ontdekkingen."
"Ik vind het altijd geweldig als we andere mensen in staat stellen de werkomstandigheden voor hun onderzoek te verbeteren en dit ook aan andere wetenschappers te communiceren. Dankzij de feedback van de wetenschappelijke gemeenschap die MZmine gebruikt, helpt het programma zelf hun onderzoek direct vooruit", voegt hij eraan toe. Robin Schmid van IOCB Praag.
De directeur van IOCB Praag, prof. Jan Konvalinka, wijst erop:"Iedereen heeft het tegenwoordig over machinaal leren en kunstmatige intelligentie. Onze collega's van het team van Dr. Pluskal gebruiken het om zeer specifieke problemen in onderzoek en in de klinische praktijk op te lossen."
De derde generatie MZmine, gerapporteerd in het voorjaar van 2023 in het tijdschrift Nature Biotechnology , kan duizenden monsters per uur verwerken. De nieuw door dit internationale team ontwikkelde algoritmen versterken dus de bestaande mogelijkheden om verschillende soorten gegevens te koppelen, inclusief het combineren van gegevens uit analytische en beeldvormende methoden, wat tot nu toe niet mogelijk was met enige academische of commerciële software.
Meer informatie: Steffen Heuckeroth et al, On-tissue dataset-afhankelijke MALDI-TIMS-MS2 bioimaging, Nature Communications (2023). DOI:10.1038/s41467-023-43298-9
Journaalinformatie: Natuurcommunicatie , Natuurbiotechnologie
Geleverd door het Instituut voor Organische Chemie en Biochemie van de Tsjechische Academie van Wetenschappen (IOCB Praag)
 Chemische reacties op bimetaaloppervlakken visualiseren
Chemische reacties op bimetaaloppervlakken visualiseren Onderzoekers ontwikkelen 's werelds eerste aerogels gemaakt van schrootbanden
Onderzoekers ontwikkelen 's werelds eerste aerogels gemaakt van schrootbanden Nieuwe klasse stoffen voor redoxreacties
Nieuwe klasse stoffen voor redoxreacties Mariene bacterie werpt licht op beheersing van giftige metalen
Mariene bacterie werpt licht op beheersing van giftige metalen waren heet, vochtige zomers de sleutel tot de oorsprong van het leven?
waren heet, vochtige zomers de sleutel tot de oorsprong van het leven?
 Indonesiërs verstikt door bosbrandnevel bidden om regen
Indonesiërs verstikt door bosbrandnevel bidden om regen Overstromingen in China om de Amerikaanse economie te treffen - klimaateffecten via handelsketens
Overstromingen in China om de Amerikaanse economie te treffen - klimaateffecten via handelsketens In de bossen van Siberië, klimaatverandering wakkert zombiebranden aan
In de bossen van Siberië, klimaatverandering wakkert zombiebranden aan Tropische storm Barijat lijkt ongeorganiseerd voor NASA-NOAA-satelliet
Tropische storm Barijat lijkt ongeorganiseerd voor NASA-NOAA-satelliet Waar kunnen overstroomde velden helpen om het grondwater aan te vullen?
Waar kunnen overstroomde velden helpen om het grondwater aan te vullen?
Hoofdlijnen
- Weten eiwitten het nog?
- Wetenschappers beperken, bestuderen Chinook in het herstelde leefgebied van de Snoqualmie-rivier
- Abiogenese: definitie, theorie, bewijs & voorbeelden
- Dierenartsen werken samen met vissers om de gezondheid van per ongeluk gevangen zeeschildpadden te evalueren
- Ontdekking legt het immuunsysteem bloot
- Kinesins negeren zwakke krachten omdat ze zware lasten dragen
- Schimmelmicroben als biomeststoffen in landbouw en tuinieren:is de beloning groter dan het risico?
- Onderzoek wijst op jaarlijkse migratie van roodkeelduikers in het oosten van de VS
- Onderzoek onthult veranderingen in de koolstof- en nutriëntenvoorraden in de bodem in landbouwgrond in woestijnoases
- Voor CRISPR, het tweaken van DNA-fragmenten voordat ze worden ingevoegd, levert de hoogste efficiëntie tot nu toe op

- Een doorbraak in onderzoek naar chirale polymere dunne films zou een nieuwe generatie apparaten mogelijk kunnen maken
- Techniek maakt afdrukbare en herschrijfbare kleurenafbeeldingen mogelijk
- Onderzoekers onthullen hoe de MRSA-bacterie omgaat met stress

- Onderzoekers vinden veelbelovende kandidaten voor nanodeeltjes voor het afvangen en omzetten van koolstofdioxide

 Hoe biomethaan kan helpen gas om te zetten in een hernieuwbare energiebron
Hoe biomethaan kan helpen gas om te zetten in een hernieuwbare energiebron Hier is waarom uw telefoon stopt met werken in de kou
Hier is waarom uw telefoon stopt met werken in de kou  Koeienmest en micro-organismen gebruiken om luiers en maandverband te composteren
Koeienmest en micro-organismen gebruiken om luiers en maandverband te composteren  Wetenschappers ontdekken driedimensionale structuur in kleinere waterdruppels
Wetenschappers ontdekken driedimensionale structuur in kleinere waterdruppels NASA smeekt toeschouwers om astronautenlancering:blijf alsjeblieft thuis!
NASA smeekt toeschouwers om astronautenlancering:blijf alsjeblieft thuis! Een bedrukbare inkt die zowel geleidend als transparant is, blokkeert ook radiogolven
Een bedrukbare inkt die zowel geleidend als transparant is, blokkeert ook radiogolven Machine learning-software voorspelt gedrag van bacteriën
Machine learning-software voorspelt gedrag van bacteriën Onderzoek onderzoekt factoren van veerkrachtige stadsontwikkeling
Onderzoek onderzoekt factoren van veerkrachtige stadsontwikkeling
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

