
Wetenschap
Nieuwe benadering van metabolomics-onderzoek kan game changer zijn
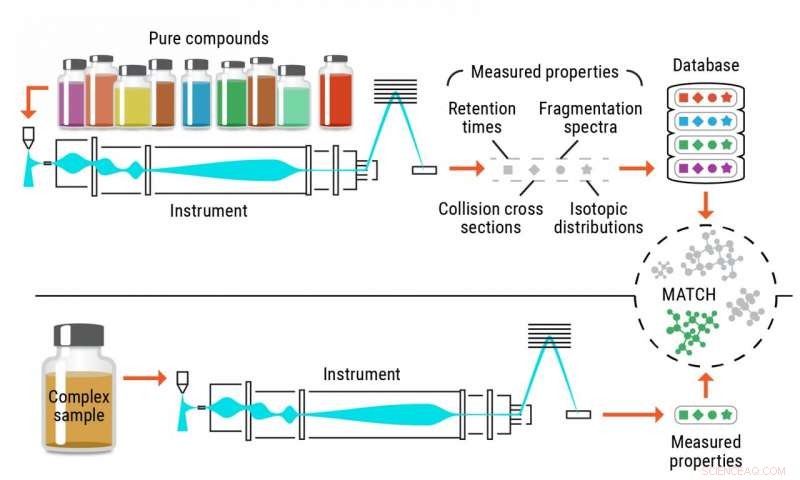
Illustratie van het conventionele identificatieproces van metabolieten. Krediet:Pacific Northwest National Laboratory
Nauwkeurige identificatie van metabolieten, en andere kleine chemicaliën, in biologische en milieumonsters is historisch gezien tekortgeschoten bij het gebruik van traditionele methoden. Conventionele tactieken zijn gebaseerd op pure referentieverbindingen, normen genoemd, dezelfde moleculen herkennen in complexe monsters. Deze benaderingen worden beperkt door de beschikbaarheid van de zuivere chemicaliën die als normen worden gebruikt.
"We wilden echt voorbijgaan aan het huidige paradigma van hoe een metabolomics-experiment wordt uitgevoerd en hoe moleculen met vertrouwen worden geïdentificeerd, " zei Tom Metz, biomedisch wetenschapper bij Pacific Northwest National Laboratory (PNNL) en directeur van de Pacific Northwest Advanced Compound Identification Core.
Een probleem met de huidige methode is dat er maar zoveel zuivere verbindingen zijn die onderzoekers van leveranciers kunnen kopen; de meeste leveranciers hebben toegang tot ongeveer 3, 000-4, 000 verbindingen.
"Als je bedenkt wat er in de natuur naar verwachting zal gebeuren, je kijkt naar> 1030 verbindingen of meer die mogelijk zouden kunnen zijn, "zei Metz. "Dus, als je de paar duizend standaardchemicaliën waar je toegang toe hebt vergelijkt met het enorme aantal potentiële verbindingen, je bent niet eens in de buurt."
Identificatiebenadering zonder normen
Om dit probleem op te lossen, hebben Metz en zijn team bij PNNL een benadering geconceptualiseerd - standaardvrije metabolomics - waarmee ze informatie over meerdere eigenschappen voor moleculen van belang berekenen of voorspellen om uitgebreide referentiebibliotheken te genereren en vervolgens experimentele gegevens met dezelfde eigenschappen te matchen met deze bibliotheken, identificatie van verbindingen mogelijk maken.
Door gebruik te maken van deze nieuwe aanpak, onderzoekers sturen chemische structuren via machine learning of kwantumchemieprogramma's om de experimentele eigenschappen van de metabolieten nauwkeurig te voorspellen.
"Als we nauwkeurig genoeg zijn met deze voorspellingen, zouden we theoretisch nooit meer een zuivere verbinding hoeven te analyseren, " zei Metz. "Deze verzameling tools zal het huidige paradigma in metabolomics veranderen, en in de nabije toekomst zullen er een aantal echt goede toepassingen zijn om de onderzoeksgemeenschap de voordelen van deze nieuwe aanpak te laten zien."
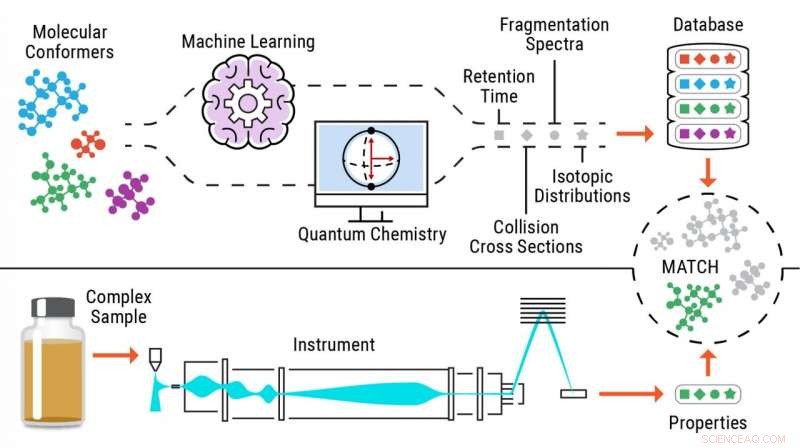
Illustratie van normvrij identificatieproces van metabolieten. Krediet:Pacific Northwest National Laboratory
Door niet te hoeven vertrouwen op gegevens uit analyses van pure standaarden om kleine moleculen te identificeren, de standaardvrije benadering maakt de identificatie van tot 90 procent meer chemicaliën in monsters mogelijk en maakt deze rekenhulpmiddelen zeer nuttig in verschillende toepassingsgebieden, waaronder de ontdekking van nieuwe medicijnen, chemisch forensisch onderzoek, en milieu- en biomedisch onderzoek.
"Bijvoorbeeld, in een nieuw medicijnontwerp zou een gebruiker kunnen zeggen:'Ik heb een bepaald aantal eigenschappen met deze bepaalde medicijnen, maar ze zijn toevallig giftig. Kunnen we een verbinding voorspellen die vergelijkbare eigenschappen heeft, maar niet giftig is?'" zei Metz. "Als de juiste trainingsgegevens aan het DarkChem-programma kunnen worden gegeven, DarkChem zou dan die voorspelling kunnen doen."
Aanpasbare reeks programma's
De nieuwe benadering voor de identificatie van metabolomics zonder normen maakt gebruik van vier belangrijke instrumenten om uitgebreide, in van silicium afgeleide metabolietreferentiebibliotheken, en om experimentele gegevens te extraheren en te matchen om verbindingsidentificaties op te leveren:
- In Silico Chemical Library Engine (ISiCLE), een high-performance-computing-vriendelijke, kwantumchemische benadering voor het genereren van voorspelde chemische eigenschappen.
- DarkChem, een variabele autoencoder die een continue numerieke of latente weergave van de moleculaire structuur leert, die referentiebibliotheken kunnen karakteriseren en uitbreiden.
- Gegevensextractie voor geïntegreerde multidimensionale spectrometrie (DEIMoS), een modulaire softwaretool die functies kan extraheren uit gegevens die zijn verzameld op multidimensionale analytische platforms.
- Multi Attribuut Matching Engine (MAME), die experimentele gegevens koppelt aan referentiebibliotheken op basis van verschillende chemische kenmerken.
De tools zijn ontworpen om samen te werken, maar ze kunnen ook afzonderlijk worden gebruikt. Onderzoekers kunnen de verschillende toepassingen aanpassen op basis van de behoeften of onderzoeksgebieden van een klant, het creëren van een volledig modulaire aanpak.
Een onderzoeksveld vooruit helpen
Direct, in de metabolomics-gemeenschap, alle onderzoekers identificeren dezelfde set moleculen in elk monster. De reden daarvoor is dat ze allemaal dezelfde zuivere verbindingen hebben die ze hebben gekocht om hun referentiebibliotheken uit te bouwen.
"Onze visie is dat je door de standaardvrije benadering te gebruiken nooit wordt beperkt door de uitgestrektheid van kleine moleculen die in een monster kunnen worden geïdentificeerd, " zei Metz. "Dat is echt een gamechanger voor metabolomics. En het is heel spannend om te zien wat het komende jaar hiervoor in petto heeft."
 Semi-flexibele modelgebaseerde analyse van celadhesie aan hydrogels
Semi-flexibele modelgebaseerde analyse van celadhesie aan hydrogels Aluminiumoxidekristal getest als UV-stralingssensor
Aluminiumoxidekristal getest als UV-stralingssensor Micro-brouwen gaat meer micro
Micro-brouwen gaat meer micro Gecko-adhesietechnologie komt dichter bij industrieel gebruik
Gecko-adhesietechnologie komt dichter bij industrieel gebruik NIST beschrijft plannen voor het herzien van de wetenschappelijke grondslagen van forensische methoden
NIST beschrijft plannen voor het herzien van de wetenschappelijke grondslagen van forensische methoden
 Inheemse beschermde gebieden zijn de volgende generatie van natuurbehoud
Inheemse beschermde gebieden zijn de volgende generatie van natuurbehoud Soorten Desert Fungi
Soorten Desert Fungi 5 dingen die u moet weten over het nieuwe VN-rapport over klimaatverandering
5 dingen die u moet weten over het nieuwe VN-rapport over klimaatverandering Japan zet zich schrap voor meer regen na overstromingen aardverschuivingen
Japan zet zich schrap voor meer regen na overstromingen aardverschuivingen Kunnen paddenstoelen echt helpen de planeet te redden?
Kunnen paddenstoelen echt helpen de planeet te redden?
Hoofdlijnen
- Verklaring van celspecialisatie
- Hoge nachttemperaturen hebben een negatieve invloed op de productie van koolzaadplanten
- Wat is een gespecialiseerd gebied van het endoplasmatisch reticulum?
- Doel van Cell Lysis Solution
- Op pinguïn gemonteerde video legt gastronomische ontmoetingen van het gelatineuze soort vast
- Nepal op schema om doel van verdubbeling tijgerpopulatie tegen 2022 te halen
- Maak een lijst van de soorten informatie die gevonden kan worden door de sequentie van een DNA-molecuul te kennen Molecule
- Insecten plagen met bitterzoete smaak om gewassen te beschermen
- Toch niet zo verschillend:menselijke cellen, winterharde microben delen een gemeenschappelijke voorouder
- Realtime dampanalyse kan de training van explosievendetecterende honden verbeteren

- Een opschaalbare nanoporeuze membraancentrifuge voor ontzilting door omgekeerde osmose zonder vervuiling

- Een moleculaire routekaart naar het immuunsysteem van planten

- Wetenschappers kweken bacteriën die kleine energieke koolstofringetjes maken

- Verven en lakken op basis van aardappelzetmeel

 Onderzoekers stellen nieuwe krachtige dual-ion-batterijen voor met een 3D-poreuze structuur
Onderzoekers stellen nieuwe krachtige dual-ion-batterijen voor met een 3D-poreuze structuur Geneesmiddelenmaker Bristol-Myers Squibb koopt Celgene in een deal van $ 74 miljard
Geneesmiddelenmaker Bristol-Myers Squibb koopt Celgene in een deal van $ 74 miljard Door schadesporen in kaart te brengen, kunnen onderzoekers het water volgen in Photosystem II
Door schadesporen in kaart te brengen, kunnen onderzoekers het water volgen in Photosystem II Heeft een zwart gat dat een ster eet een neutrino gegenereerd? Onwaarschijnlijk, nieuwe studie toont
Heeft een zwart gat dat een ster eet een neutrino gegenereerd? Onwaarschijnlijk, nieuwe studie toont Geïntegreerde lab-on-a-chip gebruikt smartphone om snel meerdere ziekteverwekkers te detecteren
Geïntegreerde lab-on-a-chip gebruikt smartphone om snel meerdere ziekteverwekkers te detecteren In Israël, op zoek naar droogtes in het verleden en de toekomst
In Israël, op zoek naar droogtes in het verleden en de toekomst Hoe streaming media ons van gedachten kan doen veranderen over culturele verschillen
Hoe streaming media ons van gedachten kan doen veranderen over culturele verschillen WHO, Greta of perswaakhonden voor Nobelprijs voor de Vrede?
WHO, Greta of perswaakhonden voor Nobelprijs voor de Vrede?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

