
Wetenschap
Hoe markergenen in celclusters te vinden
- Invoer:eencellige RNA-seq-gegevens (telmatrix)
- Kwaliteitscontrole (QC):Verwijder cellen en genen van lage kwaliteit
- Gegevensnormalisatie:normaliseer de gegevens om technische vooroordelen te corrigeren
2. Clustering
- Voer clustering uit op de genormaliseerde gegevens om celclusters te identificeren
- Er kunnen verschillende clustermethoden worden gebruikt (bijv. k-means, hiërarchische clustering, Leuven)
3. Markergenidentificatie
- Voor elk cluster:
- Bereken de gemiddelde expressie van elk gen in de cellen in het cluster
- Vergelijk de gemiddelde expressie van genen in het cluster met die in andere clusters
- Identificeer genen die in het cluster sterk tot expressie komen in vergelijking met andere clusters
4. Validatie van markergenen
- Er kunnen aanvullende criteria worden toegepast om markergenen te selecteren:
- Vouwverandering:Denk aan genen met een hoge vouwverandering tussen het cluster en andere clusters
- Statistische significantie:gebruik statistische tests (bijv. t-test, Wilcoxon-test) om de significantie van expressieverschillen te beoordelen
- Specificiteit:Zorg ervoor dat markergenen selectief tot expressie worden gebracht in het betreffende cluster
5. Interpretatie en visualisatie
- Analyseer de functies en routes die verband houden met de geïdentificeerde markergenen
- Genereer heatmaps, vulkaanplots of andere visualisaties om de markergenen en hun expressiepatronen te presenteren
6. Validatie in onafhankelijke datasets (optioneel)
- Om het vertrouwen te vergroten, valideert u de geïdentificeerde markergenen in een onafhankelijke dataset, indien beschikbaar.
 Hoe concrete gebouwen worden gemaakt
Hoe concrete gebouwen worden gemaakt  Onderzoekers werpen licht op hoe fotopolymerisatie veel efficiënter kan worden gemaakt
Onderzoekers werpen licht op hoe fotopolymerisatie veel efficiënter kan worden gemaakt  Wat is het resultaat van het toevoegen van loodnitraat aan kaliumjodide?
Wat is het resultaat van het toevoegen van loodnitraat aan kaliumjodide?  Welk type chemische verwering is geassocieerd met de formatiestalagiteiten en stalagmieten?
Welk type chemische verwering is geassocieerd met de formatiestalagiteiten en stalagmieten?  Zijn ascorbinezuurtabletten vast bij kamertemperatuur?
Zijn ascorbinezuurtabletten vast bij kamertemperatuur?
 UNESCO dringt er bij Polen op aan te stoppen met het kappen van oerbos
UNESCO dringt er bij Polen op aan te stoppen met het kappen van oerbos Hoe gaan de straatverlichting 's nachts automatisch aan?
Hoe gaan de straatverlichting 's nachts automatisch aan?  Kan een nieuwe bosalliantie de natuurfinanciering ten goede veranderen?
Kan een nieuwe bosalliantie de natuurfinanciering ten goede veranderen?  De Amerikaanse 'allergie' voor de opwarming van de aarde:waarom?
De Amerikaanse 'allergie' voor de opwarming van de aarde:waarom?  Typen vervuiling gevonden in Brownfields
Typen vervuiling gevonden in Brownfields
Hoofdlijnen
- VN schotelt vijgcactus voor als antwoord op voedselzekerheid
- Onderzoeker koppelt zalmseks aan geologische verandering
- Relatie tussen DNA-basen Genen, eiwitten en eigenschappen
- Waarom de harde Sneeuwbalaarde onze vroegste meercellige voorouders op gang bracht
- Wat zijn etercellen?
- Hoe planten de genomische bagage van hun ouders achterlaten
- Hoe leren ziekteverwekkers ziekteverwekkers te zijn:partnerschappen tussen microben die leiden tot ziekte bij de mens
- Wat is een verschil tussen de twee soorten voortplanting?
- Hersenen hoe je werkt
- Sumatraanse tijgers op weg naar herstel in gevaar UNESCO-werelderfgoed

- Eiwit beëindigt opzettelijk de eigen synthese door de synthesemachinerie te destabiliseren - het ribosoom
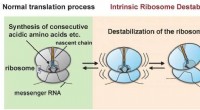
- Bijt hierop:onderzoeker ontdekt dat alligators haaien eten

- Onderzoekers rapporteren een fundamentele spiersnelheidslimiet

- Waarom zijn mensen altruïstisch?

 Een nieuwe synchrotrontechniek voor het bestuderen van diffusie in vaste stoffen
Een nieuwe synchrotrontechniek voor het bestuderen van diffusie in vaste stoffen Voedselverspilling:steden kunnen het verschil maken
Voedselverspilling:steden kunnen het verschil maken Kuststrook in Brazilië werpt nieuw licht op vroege landbouw
Kuststrook in Brazilië werpt nieuw licht op vroege landbouw Reflecterende daken kunnen oververhitting in steden verminderen en levens redden tijdens hittegolven
Reflecterende daken kunnen oververhitting in steden verminderen en levens redden tijdens hittegolven In tijden van oorlog heeft digitaal activisme macht. Hier leest u hoe u op verantwoorde wijze kunt deelnemen
In tijden van oorlog heeft digitaal activisme macht. Hier leest u hoe u op verantwoorde wijze kunt deelnemen  Consumenten kunnen onderscheid maken tussen bittere smaken in bier - verandert niets aan de smaak
Consumenten kunnen onderscheid maken tussen bittere smaken in bier - verandert niets aan de smaak Wanneer is een nova geen nova? Wanneer een witte dwerg en een bruine dwerg botsen
Wanneer is een nova geen nova? Wanneer een witte dwerg en een bruine dwerg botsen Achtervolgt de Bunyip echt de Australische wetlands?
Achtervolgt de Bunyip echt de Australische wetlands?
- Elektronica
- Biologie
- Zonsverduistering
- Wiskunde
- French | Italian | Spanish | Portuguese | Swedish | German | Dutch | Danish | Norway |

-
Wetenschap © https://nl.scienceaq.com

