
Wetenschap
Laboratorium- en natuurlijke stammen van darmbacteriën blijken vergelijkbare mutatieprofielen te hebben
Het begrijpen van mutatieprocessen in een cel biedt aanwijzingen voor de evolutie van een genoom. Het meest actief worden mutatieprocessen bestudeerd in menselijke kankercellen, terwijl andere genomen vaak worden verwaarloosd.
Een onderzoeksteam onderzocht en vergeleek het totale scala aan mutaties in laboratorium- en natuurlijke E. coli (darmbacteriën) en kwam tot de conclusie dat ze vrijwel niet van elkaar te onderscheiden zijn. De resultaten worden gepresenteerd in de Genome Biology and Evolution dagboek.
"Mutaties komen voor tijdens het replicatieproces – het kopiëren van DNA, of bij het 'repareren' van DNA als gevolg van strengbreuken of andere aandoeningen. Het reparatiesysteem kan ook fouten maken.
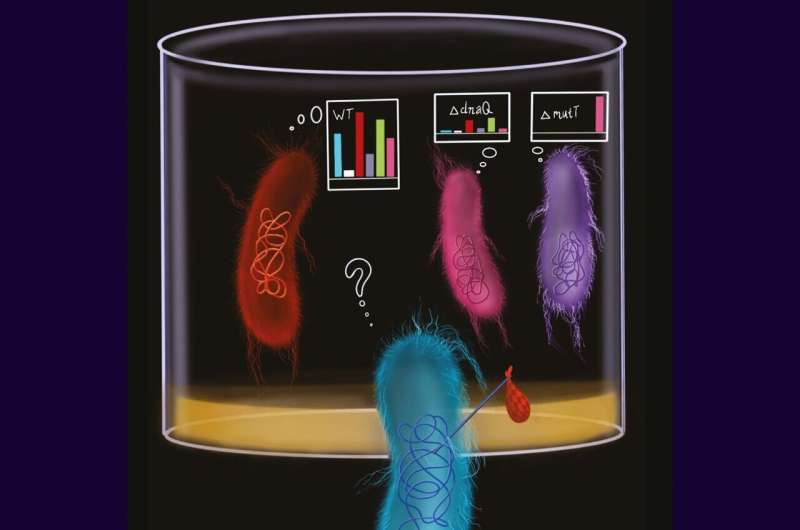
"Verschillende replicatie- en reparatiesystemen maken verschillende fouten, en deze zijn afhankelijk van de context. Er worden doorgaans drie nucleotiden onderzocht:degene waar de mutatie heeft plaatsgevonden, en twee aan de rechter- en linkerkant ervan.
"Een typische reeks mutaties, samen met de contexten die specifiek zijn voor een bepaald systeem, wordt een mutatiesignatuur genoemd. Mensen hebben bijvoorbeeld mutatiesignaturen van UV-blootstelling en roken die kenmerkend zijn voor bepaalde vormen van kanker", aldus het hoofd van de studie. Professor Mikhail Gelfand, directeur van het Biocentrum van Skoltech.
Het team voerde een allereerste onderzoek uit naar het mutatieprofiel van E. coli:het totaal aan mutatiesignaturen van replicatie- en reparatiesystemen. De eerste stap was het testen van de taak op middelbare scholieren tijdens de Summer School of Molecular and Theoretical Biology, waar onderzoekers ook in contact kwamen met Indiase collega's die experimenteel aan hetzelfde probleem werkten.
“Onze partners uit India leverden gegevens over E. coli met verstoorde replicatie- en reparatiesystemen. Van hen waren we in staat om pure mutatiesignaturen te extraheren die kenmerkend zijn voor verschillende systemen, en vervolgens de mutatieprofielen van E. coli op te splitsen in handtekeningen en te begrijpen welke systemen dragen meer bij aan mutaties”, vervolgde Gelfand.
De wetenschappers gebruikten ook experimentele gegevens over de langetermijnevolutie van E. coli. Dit laboratoriumonderzoek, dat al meer dan 30 jaar loopt, staat onder leiding van Richard Lenski. Het mutatieprofiel van laboratoriumstammen werd vergeleken met dat van natuurlijke stammen.
“We ontdekten dat deze profielen vergelijkbaar zijn. De laboratoriumstam leeft niet in omstandigheden die vergelijkbaar zijn met die in de natuur, dus dit resultaat is onverwacht. Bovendien pleiten de resultaten tegen het idee dat de evolutie van E. coli in de natuur omgeving omvat afwisselende episoden van normale langzame evolutie en kortere episoden van versnelde evolutie in een hypermuteerbare modus.
"Integendeel, tijdens de evolutie van natuurlijke stammen van E. coli is de bijdrage van atypische mutatieregimes aan de algehele accumulatie van mutaties onbeduidend", aldus Gelfand.